
Наука витает в облаках
09 июня 2017
Наука витает в облаках
- 910
- 1
- 2
коллаж автора статьи
-
Автор
-
Редакторы
Вокруг нас сотни и тысячи вычислительных устройств. Иная лампочка сейчас умнее марсохода. Но если вы возьметесь за разработку лекарства от... даже не рака, а гриппа, то одними умными лампочками, смартфонами и даже рабочими станциями уже не обойтись. Узнайте о том, какие задачи решают современные биоинформатики и в каких неожиданных местах они находят вычислительные ресурсы.

Написание и размещение этой статьи оплачено компанией HPC HUB — поставщиком сложных программно-аппаратных решений в сфере высокопроизводительных вычислений для решения ресурсоемких задач науки и бизнеса и предоставления их по модели вычислительных облаков.
HPC HUB никак не влиял на содержание этой статьи.
Современная наука немыслима без применения компьютеров для проектирования, получения или обработки результатов. В каждой области, от физики высоких энергий до систематики растений рода лютиковых, находится место, и вполне оправданное, вычислительным методам. Особое место, как очевидно уже из названия, они занимают в биоинформатике.
Океаны и моря данных
Часто биоинформатические задачи формулируются по принципу «загрузить все возможные объекты, а потом пару-тройку недель/месяцев ждать результат». Несмотря на кажущуюся абсурдность такого подхода, для него есть достаточно простая причина — взрывной рост количества данных, требующих анализа. Даже количество пространственных структур биомолекул, получаемых в ходе продолжительных и чрезвычайно трудозатратных экспериментов, растет практически экспоненциально (рис. 1 и 2).
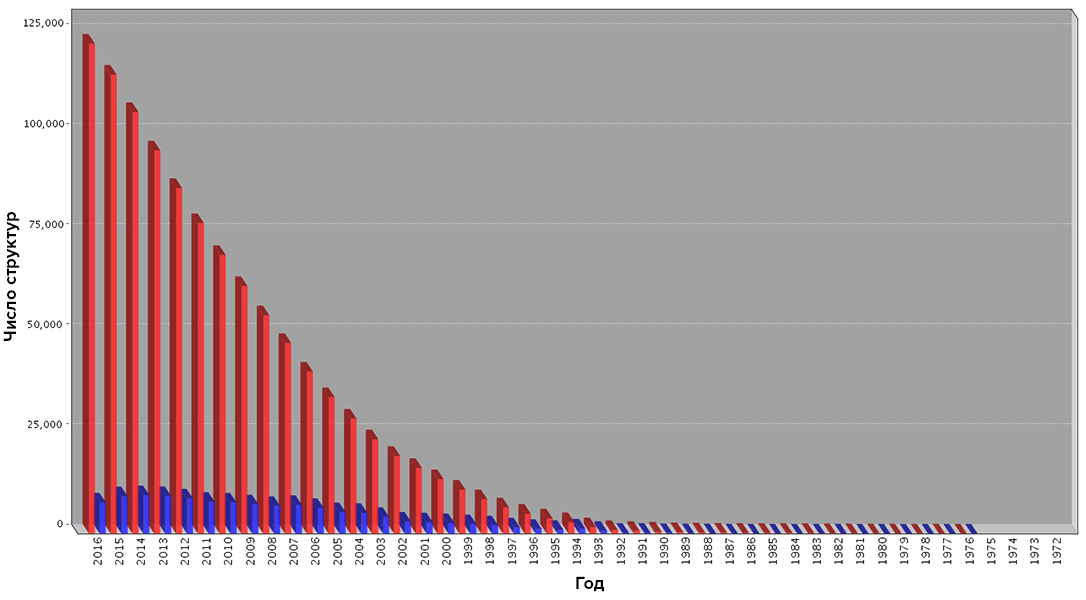
Рисунок 1. Рост числа пространственных структур биомолекул по данным банка PDB. Синим цветом показан ежегодный прирост, красным — общий за прошедшие годы. Чтобы увидеть рисунок в полном размере, нажмите на него.
сайт www.rcsb.org
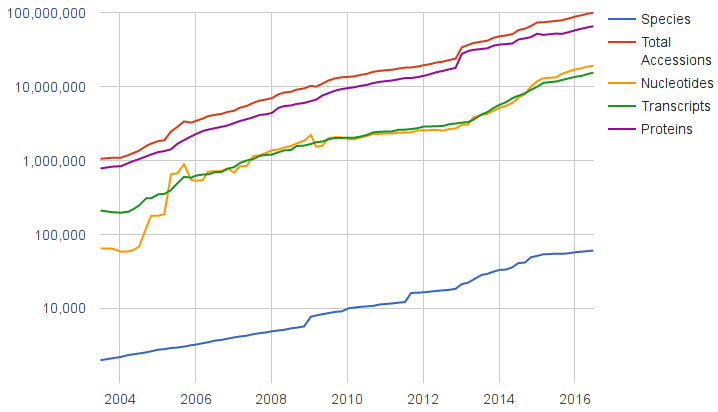
Рисунок 2. Рост числа записей базы данных RefSeq. Курируемая база RefSeq содержит неизбыточный набор геномных данных различных типов (последовательности геномов, транскриптомов, белков). Обратите внимание на логарифмическую шкалу!
сайт www.ncbi.nlm.nih.gov
Что уж говорить про геномные данные! Безумный рост их количества — следствие одного из самых интересных и значимых технологических прорывов последнего времени: высокопроизводительного секвенирования ДНК [1]. Прогресс в этой сфере настолько велик, что стоимость расшифровки одного генома снизилась с $2,7 млрд. за первый прочтенный геном в начале 2000-х годов [2] до $1000 в 2016 году [3] (рис. 3).

Рисунок 3. Снижение стоимости секвенирование генома человека по данным Национального института исследований человеческого генома (подразделение Национального института здравоохранения, США). Обратите внимание на драматическое отклонение от закона Мура!
Таким образом, из-за обилия данных у исследователя просто не остается других вариантов: он должен анализировать всю доступную информацию, иначе рецензенты могут упрекнуть его в том, что наблюдаемые явления — просто артефакт малой выборки данных, а вот если взять все... Традиционный подход к решению задач из разряда «пересчитать весь живой мир» — использование ресурсов суперкомпьютерных центров. Практически в каждом крупном научном учреждении есть свой собственный суперкомпьютер — монструозное сооружение заоблачной стоимости, для питания которого иногда требуется небольшая электростанция.
Самые мощные публичные суперкомпьютеры в России — «Ломоносов» и «Ломоносов-2» — расположены, как можно догадаться из названия, в МГУ им. М.В. Ломоносова. Полный список подобных установок можно посмотреть в ТОП 50 суперкомпьютеров СНГ. При этом в последней редакции международного рейтинга ТОП 500 оба суперкомпьютера находятся всего на 35 и 94 местах соответственно, более чем в 15 раз уступая по производительности китайскому Tianhe-2 (MilkyWay-2).
Несмотря на внушительные цифры в виде количества ядер (в одном только «Ломоносове-2» их 37 120) или пиковой производительности, оказывается, что для конечного пользователя — ученого — такие решения не всегда удобны. Типичные проблемы, с которыми вы столкнетесь, если захотите получить доступ к такому суперкомпьютеру:
- Отсутствие его в вашем учреждении.
- Большая очередь на доступ к ресурсам и их ограниченность. Типичное время ожидания в такой очереди — от нескольких часов до нескольких дней. Если же у вас очень большая задача (например, на несколько сотен узлов), то придется отдельно договариваться о приоритетном режиме выполнения. При этом администраторы вынуждены вручную приостанавливать выполнение задач других пользователей, чтобы высвободить ресурсы.
- Лимиты не только на время выполнения задач (обычно до нескольких десятков часов, реже сотен), но даже на количество одновременно запущенных задач (!).
- Не говоря уже о случае, когда вашу задачу физически нельзя запустить на доступном вам оборудовании, например, из-за недостатка дискового пространства или оперативной памяти. Например, такое часто происходит с генетическими данными — для сборки геномов или транскриптомов новых организмов требуются колоссальные объемы оперативной памяти (до 2–3 Тб). Узлы такой конфигурации практически не встречаются в суперкомпьютерах «общего назначения». Или ограниченные размеры дискового пространства: например, в моей лаборатории некоторые студенты (!) используют до 40 Тб дискового пространства на один проект, в то время как на упомянутом «Ломоносове-2» всего доступно ~400 Тб. При этом как-то повлиять на распределение ресурсов или их занятость, как правило, достаточно сложно, в силу масштабности и инертности таких центров.
- Cложность разворачивания необходимого ПО. Зачастую на таких суперкомпьютерах стоят «стабильные» версии программного обеспечения. При всех своих плюсах — отлаженности и стабильности работы, — к сожалению, нередко они не очень новые, если не сказать больше: устаревшие. В то время как разработчики, особенно аспиранты и постдоки, пишут свои программы с использованием самых свежих и функционально богатых версий библиотек. Что в свою очередь порождает существенные проблемы с переносом ПО с машины разработчика на кластер.
Подробнее — во врезке «Типичные биоинформатические задачи».
Как видите, неудобств хватает. В такой ситуации ученый, как человек, в первую очередь заинтересованный в получении результата, причем с наименьшими бюрократическими и техническими трудозатратами, постоянно ищет альтернативы.
Витаем в облаках
И тут на горизонте появляются облачные вычисления. Буквально за последние 10 лет произошел качественный скачок, и от обычного интернет-хостинга сайтов эта индустрия развилась до абсолютно гибких виртуальных окружений, предоставляющих удобный совместный доступ из любой точки мира к любым ресурсам: от видео с котятами до лабораторного журнала, который вы ведете вместе с коллегами.
Выглядит просто чудесно: перенося свои вычисления в облака (например, одно из самых известных — Amazon EC2), вы снимаете с себя или персонала вашего учреждения любые заботы, связанные с поддержкой работоспособности оборудования. Администрирование даже небольшого вычислительного кластера на сотню пользователей — занятие, сопряженное с достаточным количеством головной боли и бессонными ночами. Достаточно взглянуть на раздел «новости» суперкомпьютерного центра МГУ чтобы понять, как часто, в силу своей сложности, суперкомпьютеры выходят из строя, выводятся на профилактические работы или для выделенных расчетов. Облако же доступно везде и всегда (почти).
Вы получаете вычислительные ресурсы только тогда, когда они вам нужны. Не секрет, что в научной работе есть свои внутренние ритмы. Традиционно, вы несколько месяцев что-то упорно считаете, а потом до нескольких месяцев можете потратить на написание статей, отчетов, поездки по конференциями и учебным практикам, практически ничего не запуская. К тому же влияют и более глобальные ритмы — например, в силу общего расписания учебного процесса летом на суперкомпьютерах МГУ становится существенно просторнее, и время ожидания в очереди в некоторых случаях сокращается с дней до минут. Соответственно, в остальное время вам не приходится думать о том, как содержать этого монстра, завтракающего киловаттами энергии, а также штат высококвалифицированных системных администраторов и инженеров для поддержания его круглосуточной бесперебойной работы. А надо заметить, зарплатные ожидания таких специалистов отнюдь не самые скромные.
Вы легко можете подобрать только те ресурсы, которые вам нужны в настоящий момент. Например, задачи из области моделирования молекулярной динамики могут хорошо распараллеливаться и не требовать значительных объемов оперативной памяти, в то время как анализ геномных данных, как правило, наоборот, требует бóльшей оперативной памяти, но мало зависим от количества ядер. При этом, если по ходу решения научной проблемы требования к оборудованию изменяются, вы можете переконфигурировать ресурсы в течение минут.
Еще одна традиционная проблема — доступ пользователей на кластеры. Как правило, пользователь — владелец аккаунта — обязуется не передавать доступ третьим лицам. Что доставляет существенные неудобства, особенно при работе в международных группах. И потом приходится дополнительно регулировать доступ всех членов группы к набору данных и т. д. В случае же облачного «кластера» вы сами распоряжаетесь ресурсами и доступом — никакой внешней бюрократии.
Неудивительно, что такой набор достоинств заинтересовал не только обычных пользователей или коммерческие компании, но и ученых.
По дороге с облаками
Целый ряд лабораторий из самых разных областей биоинформатики попытались перенести свои рабочие процессы на виртуальные мощности облаков. Например, Институт геномной медицины Национального медицинского университета Сеула перенес в облако Amazon обработку данных RNA-seq, получаемых в ходе их исследований. Разработав FX — свой собственный инструмент для эффективного анализа RNA-seq [5], — они столкнулись с типичными проблемами поддержки собственной вычислительной инфраструктуры и перенесли платформу FX в облако.
Аналогично поступили исследователи из Broad Institute, решающие похожие задачи при изучении онкологических заболеваний (рис. 4). В частности, при помощи методов машинного обучения они пытаются выявить особенности профилей экспрессии генов, участвующих в развитии опухолей. По их собственным утверждениям, им удалось сформировать на платформе CycleCluster виртуальный кластер из ~50 000 ядер (а это, на секундочку, в 1,5 раза больше чем в «Ломоносове-2»!) и таким образом меньше, чем за $5000, и всего 6 часов реального времени провести анализ, который иначе потребовал бы 30 лет для расчета на одном процессоре или несколько миллионов долларов, чтобы построить аналогичный по производительности физический кластер.
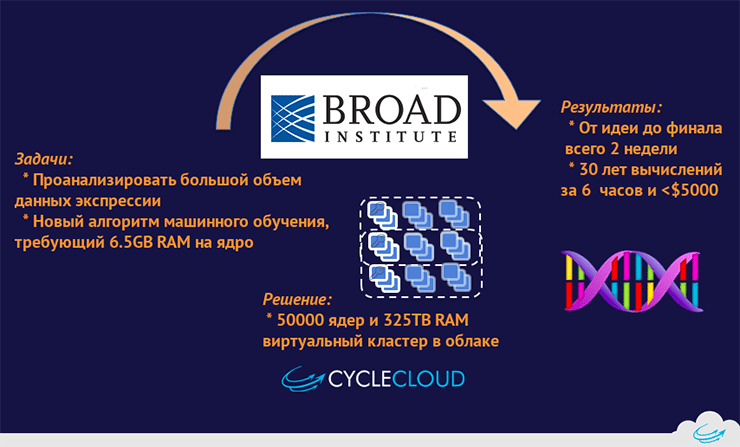
Рисунок 4. Схема анализа онкологических данных с применением облачных вычислений в Broad Institute. Объем использованных ресурсов сопоставим с суперкомпьютером «Ломоносов-2».
сайт cyclecomputing.com
Интересные примеры использования облаков есть и в моей любимой области — моделировании структур биомолекул. Оказалось, что традиционно хорошо масштабируемая область рационального дизайна лекарственных препаратов также успешно может быть перенесена в облачное окружение. Команда из компании InhibOx (теперь Oxford Drug Design), предоставляющая своим заказчикам библиотеки соединений, которые в будущем могут стать лекарствами, использовала облако Amazon для генерации 3D-структур своих соединений и расчета их физико-химических свойств.
А другая группа разработала инструмент, который позволяет одновременно использовать сразу несколько облаков (AWS и Microsoft Azure) [6]! Благодаря оптимизации программного обеспечения и распределению нагрузки между несколькими облаками им удалось достичь впечатляющей производительности в задаче моделирования сворачивания структуры белков [7]. Детальное понимание этого процесса позволяет выявить причины некоторых нейродегенеративных заболеваний, связанных с некорректным фолдингом белков, например, болезни Крейтцфельдта—Якоба. Есть и другие заболевания, к которым приводит неправильный фолдинг. Среди них известные (печально, особенно для российских пациентов) муковисцидоз и лизосомные болезни накопления.
Дедал или Икар?
К сожалению, реальность, как обычно, если не вдребезги разбила розовые очки, то, как минимум, заставила сменить оттенок линз.
Уже после первых опытов вскрылся целый ряд проблем.
Первая — обмен данными с облаком. Загрузить в облако несколько сотен гигабайт или даже терабайты «сырых» данных не так уж легко. Вы можете удивиться, но даже некоторые профильные (связанные с ИТ) научные институты до сих пор имеют внешние каналы пропускной способностью лишь 100 Мбит! Аналогичная проблема возникает и при скачивании результатов.
Вторая — специфика задач. Это уже более ожидаемо, но тем не менее оказалось, что лишь небольшой класс задач хорошо адаптируется и масштабируется на облачных ресурсах, которые, как правило, представляют из себя «фермы», состоящие из большого количества серверов, связанных не самыми быстрыми каналами передачи данных. Соответственно, на них приемлемо могут работать только массивно-параллельные задачи с малой степенью связности отдельных потоков (например, молекулярный докинг или виртуальный скрининг химических баз). Но согласно выводам нескольких работ, даже на таких задачах оптимальная производительность достигается только на самых мощных узлах, доступных у облачного провайдера [8], [9].
Третья — несмотря на кажущуюся простоту, для эффективного управления ресурсами облака необходимо соответствующее ПО. Чтобы учесть специфику конкретной задачи — потоки данных, периоды нагрузки на отдельных стадиях и так далее — необходимы достаточно квалифицированные специалисты, которые также знакомы с особенностями как предметной области задачи, так и облачными вычислениями. Из-за относительной молодости этой отрасли, их отнюдь не так много. Хотя подвижки есть, например Amazon собирает коллекцию биоинформатических инструментов, оптимизированных под их облако.
Но описанные проблемы, пожалуй, можно отнести к «детским» болезням, нежели к принципиальным и неразрешимым. Вопрос с широкополосным доступом научных учреждений вообще выглядит смешным и имеет отчетливую бюрократическую историю, нежели какую-то другую.
Решается также вопрос с облачной инфраструктурой, приспособленной для более широкого спектра задач. Например для молекулярной динамики, где узлы кластера должны постоянно общаться друг с другом. Реализовать такую инфраструктуру гораздо сложнее. Тем не менее, российский IT-стартап HPC Hub внедрил в свое облако высокопроизводительные решения, аналогичные тем, что используются во «взрослых» суперкомпьютерах: сеть обмена данными на основе технологии Infiniband и распределенную файловая система GFS2.
Также по мере увеличения популярности облачных расчетов появляются и более удобные инструменты с открытым исходным кодом для управления облачными ресурсами. Современные инструменты предоставляют практически прозрачный интерфейс для управления ресурсами сразу нескольких популярных публичных облаков, что существенно упрощает процесс создания виртуальных машин, распределение данных между ними и сбор результатов. Примеры — OpenStack, OpenNebula, Juju, Ansible.
Принципиальной, однако, остается проблема прогнозирования загрузки. В типичной исследовательской работе далеко не всегда можно разумно спрогнозировать необходимое количество ресурсов. Гипотезы могут не подтверждаться, применяемые уровни теории не показывать нужных результатов и так далее. Процесс исследовательского поиска может легко растягиваться на десятки тысяч процессоро-часов. И в этом случае локальный суперкомпьютер, который, как правило, доступен на достаточно пермиссивных и некоммерческих условиях, будет однозначно выигрывать у облака.
С другой стороны, если вы отчетливо представляете планируемое потребление ресурсов (например, у вас есть отработанный вычислительный конвейер), то облака могут стать исключительно удобным инструментом. А если можно разместить в облаке еще и весь инструментарий для анализа и выдачи готовых результатов, то эффективность облачного решения становится очевидной. Такие модели использования наверняка найдут (а на самом деле и уже находят) своих пользователей в среде биоинформатических стартапов (можно упомянуть «Кномикс» и «АйБином»; см. врезку), в учебных курсах и хакатонах.
Показательный пример — хакатон GeneHack-2, который проводился в облаке HPC Hub, и победителям которого вручили сертификаты на использование его ресурсов. Каждая команда участников получила абсолютно идентичные рабочие системы с одинаковым лимитом вычислительных ресурсов. В случае использования физических систем, развертывание и поддержка инфраструктуры для соревнования отняла бы у организаторов не минуты, а долгие мучительные часы, а то и дни.
И хочется акцентировать внимание на словосочетании «идентичные системы» из предыдущего абзаца. Использование «образов» систем — полнофункциональных окружений, которые могут быть в считанные секунды развернуты на любом вычислительном узле, — позволяет изящно обойти проблему с переносом ПО от разработчика на суперкомпьютер. Однажды сформированный образ с рабочей средой может быть легко растиражирован и свободно передан даже в другое облако.
Неожиданно оказалось, что такой способ организации рабочих сред помогает в достижении целей открытой и воспроизводимой науки, чтобы независимые исследователи проверяли валидность результатов в абсолютно идентичном окружении [15]. А это чрезвычайно важно, так как в современном геномном анализе, например, используются наборы из десятков инструментов, которые в сумме содержат сотни параметров, и чье поведение может меняться от версии к версии (и это не считая банальных ошибок в коде!). Отличным уже живым примером такого подхода является стартап InsideDNA, запустивший облачную платформу для воспроизводимого анализа геномных данных. Пример использования платформы показан в записи онлайн-трансляции воркшопа, проведенного при участии проекта FutureBiotech Live:
И чего, пожалуй, меньше всего ожидали исследователи — система образов рабочих сред может существенно сократить путь разработки уникальных алгоритмов или подобранных конвейеров программ от лабораторий до внедрения в стартапах или экспериментальных медицинских лабораториях. Например, высокопроизводительное облако HPC Hub планирует запустить своего рода «App Store», где исследователи и стартапы смогут на взаимовыгодных условиях обмениваться такими образами.
Заключение
Облака, будучи даже совсем молодой технологией, смогли привлечь значительное внимание. Перспективы их применения в наукоемких отраслях чрезвычайно заманчивы. Остается только пережить период «детских» болезней, принимая, по возможности, активное участие в оптимизации существующих и разработке новых инструментов для их использования.
Литература
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Геном человека: как это было и как это будет;
- Технология: $1000 за геном;
- Просто о корпоративном IaaS: что это, для кого, и как оплачивается. (2008). Сайт «Хабрахабр»;
- Hong D., Rhie A., Park S.S., Lee J., Ju Y.S., Kim S. et al. (2012). FX: an RNA-Seq analysis tool on the cloud. Bioinformatics. 28, 721–723;
- Abdul-Wahid B., Yu L., Rajan D., Feng H., Darve E., Thain D., Izaguirre J.A. (2012). Folding proteins at 500 ns/hour with Work Queue. Proc. IEEE Int. Conf. Escience. 2012, 1–8;
- Проблема фолдинга белка;
- Jackson K.R., Ramakrishnan L., Muriki K., Canon S., Cholia S., Shalf J. et al. (2010). Performance analysis of high performance computing applications on the Amazon Web Services Cloud. IEEE Second International Conference on Cloud Computing Technology and Science;
- Juve G., Deelman E., Vahi K., Mehta G., Berriman B., Berman B.P., Maechling P. (2009). Scientific workflow applications on Amazon EC2. 5th IEEE International Conference on E-Science Workshops;
- Виртуальные тропы реальных лекарств;
- I. V. Smirnov, A. V. Golovin, S. D. Chatziefthimiou, A. V. Stepanova, Y. Peng, et. al.. (2016). Robotic QM/MM-driven maturation of antibody combining sites. Science Advances. 2, e1501695-e1501695;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- Код жизни: прочесть не значит понять;
- За генный полиморфизм приходится платить;
- Bill Howe. (2012). Virtual Appliances, Cloud Computing, and Reproducible Research. Comput. Sci. Eng.. 14, 36-41.






