Мáркеры для клеток: как ученые «раскрашивают» клетки, чтобы отличить одну от другой
14 ноября 2017
Мáркеры для клеток: как ученые «раскрашивают» клетки, чтобы отличить одну от другой
- 2795
- 0
- 10
Выделить все важное и отличить от других — вот задача клеточных мáркеров.
-
Автор
-
Редакторы
Темы
Статья на конкурс «био/мол/текст»: В этой статье рассказывается о том, как ученые научились в широком смысле «раскрашивать» клетки — выделять из общей смеси только те, которые им интересны, а остальные отсеивать. Речь пойдет как о естественных поверхностных и внутриклеточных мáркерах и способах их выявления, так и о более изощренных генетических способах маркировать клетки. Читателя ждет открытие, что штрих-коды бывают не только в магазинах, но и в клетках, а вирус иммунодефицита человека превратился в послушный инструмент.
Конкурс «био/мол/текст»-2017
Эта работа опубликована в номинации «Свободная тема» конкурса «био/мол/текст»-2017.

Генеральный спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

Спонсором приза зрительских симпатий и партнером номинации «Биомедицина сегодня и завтра» выступила фирма «Инвитро».

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Что первое приходит на ум неискушенному в науке человеку при слове «мáркер»? Бьюсь об заклад, что добрая половина представляет себе желтый широкий фломастер для выделения текста. Другая половина, наверняка, представляет себе черный нестираемый маркер, которым одно время были исписаны все трубы, заборы и веранды. Начертания «Маша + Саша = Любовь» скрепляли чувства подростков навечно, а менее романтичные, но выразительные фразы надолго свидетельствовали о личной неприязни. Встречались даже стихи. Однако предлагаю уважаемым читателям узнать еще одно значение этого слова — очень научное и так нужное всей современной биологии и биомедицине. В научном понимании и конкретно в клеточной биологии, слово «маркер» в широком смысле означает что-то, что отличает одну живую клетку (или группу клеток) от других. Это может быть молекула на поверхности клеточной мембраны, или какой-нибудь внутриклеточный белок, или вещество, которое синтезируется/накапливается только в этом конкретном виде клеток, или генетическая особенность, присущая только данному типу клеток. Условно все маркеры можно разделить на естественные, которые существуют сами по себе, и искусственные — введенные намеренно. Давайте разберемся с ними по порядку.
Естественные клеточные маркеры и способы их рассмотреть
В многоклеточных организмах существует клеточная специализация. Несмотря на то, что клетки содержат одинаковую ДНК, они отличаются друг от друга: клетки мышц умеют сокращаться, нейроны — проводить нервные импульсы, и так далее. Это происходит из-за того, что в каждом виде клеток работает только определенный набор генов из всего их разнообразия, содержащегося в ДНК. Именно поэтому маркером клеток может служить любой ген, который экспрессируется избирательно — только в каком-нибудь конкретном виде клеток. Особенно удобно, когда такой отличительный ген кодирует белок, располагающийся на поверхности клетки.
В этом случае у ученых есть целый набор методов, как можно «увидеть» клетки, несущие такой маркер, отличить их от остальных клеток и даже отсортировать: в одну пробирку — клетки с поверхностным маркером (или с несколькими маркерами), в другую — остальные. Эти методы основаны на использовании антител.
Антитела — специальные белки, которые могут распознавать молекулы за счет пространственного взаимодействия. Именно антитела играют важную роль в нашем иммунитете — они связываются с белками или другими молекулами на поверхности бактерий и вирусов, что и запускает иммунную реакцию. Современная наука научилась делать антитела против практически любых белков — в том числе и против поверхностных клеточных маркеров. Чтобы использовать антитела для определения маркеров, ученые прибегают к еще одной хитрости. Они присоединяют к антителу еще один белок: или фермент, или флуоресцирующий (светящийся) белок. Тогда, если к исследуемым клеткам добавить антитела против поверхностного маркера, связанные с ферментом, потом отмыть лишние антитела и добавить субстрат для фермента, который при расщеплении приобретает какой-нибудь цвет, то клетки, несущие на своей поверхности нужный маркер, окажутся окрашенными, а остальные клетки — останутся бесцветными (рис. 1).
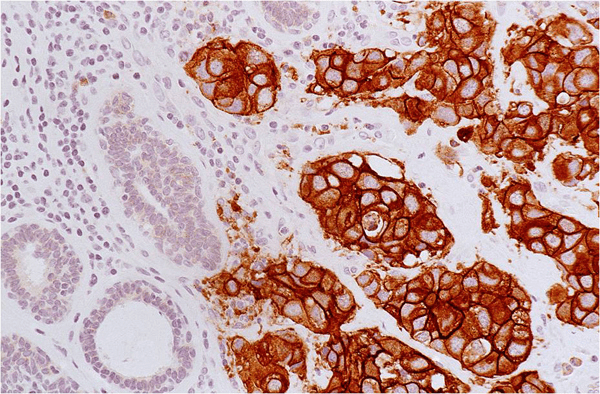
Рисунок 1. Пример выявления онкомаркера HER2 с помощью антител (иммуногистохимия). Коричневые клетки несут на своей поверхности HER2.
Такая модификация выявления маркера называется иммуногистохимией («иммуно» — из-за того, что используют иммунные антитела, «химия» — потому что в методе присутствует химическая реакция расщепления субстрата ферментом, «гисто» — потому что чаще всего такой метод применяют к гистологическим срезам — тонким слайсам исследуемой ткани, которые закреплены на стеклянной подложке).
Другой метод, позволяющий выявлять поверхностные маркеры, называется проточной цитофлуориметрией (fluorescence-activated cell sorting, FACS) [3]. Здесь исследуемые клетки (в виде одноклеточной взвеси) смешивают с антителами, к которым присоединены флуоресцирующие белки. Антитела связываются со своими маркерами-мишенями, a не связавшиеся антитела удаляют. Далее клетки загружают в прибор. Он создает тонкую струю из клеточной суспензии, проходящую через свет лазера буквально по одной клетке. Если на поверхности клетки есть искомые маркеры, то вместе с клеткой в луч лазера попадают и антитела, связавшиеся с маркерами. Происходит возбуждение флуорофора на антителе, и он испускает на своей длине волны свет, который и регистрируется детектором (рис. 2).
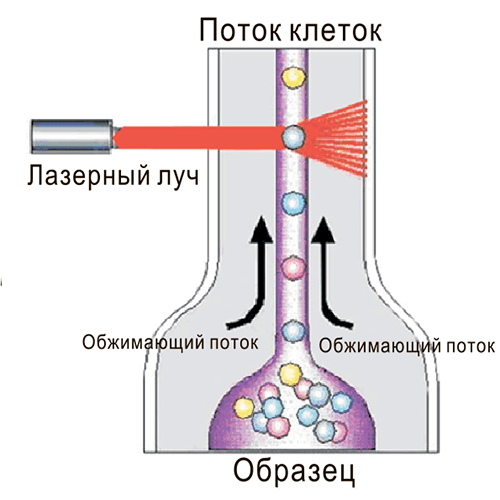
Рисунок 2. Общий принцип проточной цитофлуориметрии.
сайт istselenie-don.ru
После этого прибор может отклонить эту клетку из струи или в одну сторону, или в другую. Таким образом можно разделить клетки на две группы: с маркером и без. Получается, что прибор «знает», есть ли искомые маркеры на каждой из исследуемых клеток, сколько таких клеток, умеет сортировать нужные и ненужные клетки.
Описанные способы позволяют выявлять естественные маркеры — молекулы, которые и до начала исследования присутствовали на поверхности или внутри клеток. Эти методы сильно помогли ученым разобраться в том, чем опухолевые клетки отличаются от нормальных, стволовые клетки — от более зрелых и дифференцированных, и разные ткани — друг от друга.
Искусственные маркеры: внедрить и распознать
У описанных выше методов есть один существенный недостаток: они не позволяют установить родственные связи между клетками. Вот, предположим, у нас есть 100 клеток, и у 12 из них есть маркер А на поверхности. Все эти клетки А для нас будут на одно лицо, как близнецы (рис. 3).

Рисунок 3. Братья-близнецы из фильма «Приключения Электроника».
Мы не сможем сказать, произошли ли все эти 12 клеток от одной материнской и являются ли «близкими родственниками». Или, скажем, можно предположить, что они получились после деления нескольких клеток с исследуемым маркером и являются друг другу двоюродными, троюродными братьями и сестрами, а может быть и вовсе седьмой водой на киселе. Чтобы исследовать родственные связи в клеточной популяции (еще говорят — «исследовать клональный состав популяции» [4]) и следить за тем, как ведет себя потомство отдельных клеток, ученые научились вводить искусственные генетические маркеры в клетки. Делают это несколькими способами. Один из самых продвинутых способов на сегодняшний день — использование специальных вирусов.
Современная генная инженерия достигла таких невиданных высот, что специалисты научились конструировать вирусы с заранее известными, тщательно отобранными для целей исследователей свойствами. Например, можно создать вирус, который способен заражать клетку — проникать через ее оболочку в ядро, — встраиваться в ДНК зараженной клетки, и больше ничего не делать: не создавать новые вирусные частицы, не копировать себя в геноме зараженной клетки, не экспрессировать ни один из вирусных генов. Вместо этого ученые смогли заставить вирус, который встроился в ДНК клетки, экспрессировать нужные белки (например, на этом принципе основана генотерапия). В частности, можно сделать так, что с ДНК вируса экспрессируется флуоресцентный белок [5], и зараженная клетка начинает светиться во флуоресцентном микроскопе (рис. 4).
Вирусы, которые способны направленно доставлять гены в ДНК клетки, стали называть вирусными векторами.
Введение вектора в клетки для исследования их клонального состава принято называть маркированием клеток. Вы здесь совершенно справедливо спросите: «Позвольте, а где же здесь маркирование? Ну, ввели мы ДНК вируса в ДНК клетки, но дальше-то что?». А я Вам отвечу, что здесь-то и начинается самое интересное! Введенная ДНК действительно может выступать в роли маркера. Здесь есть два варианта: 1) маркером может служить не сама вирусная ДНК, а то место, куда она встроилась; 2) маркер может содержаться в самой вирусной ДНК.
Разберем эти возможности по порядку. Итак, наш вирус проник в клетку, сумел добраться до ядра, с боем прорвался через ядерную мембрану и встроился в ДНК клетки-хозяина. Но вот вопрос: в какое именно место он встроился? Вы когда-нибудь видели, как выглядит ДНК в клетке? Ну, вот примерно так, как на рисунке 5 .
Про то, каким удивительным образом в одном клеточном ядре величиной в несколько микрометров упаковываются молекулы ДНК, длина которых измеряется в метрах, читайте увлекательную статью на «Биомолекуле»: «Организовать геном: запутанная история гипотез и экспериментов» [6].
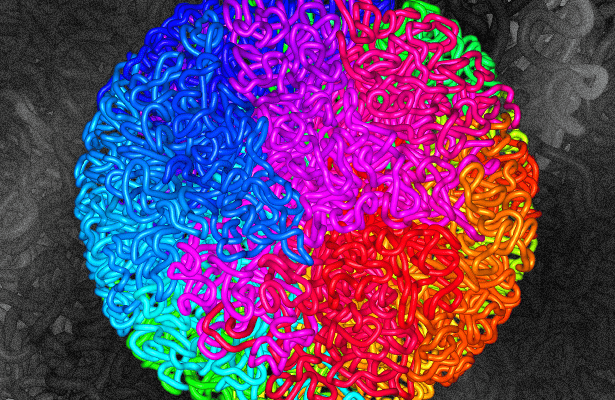
Рисунок 5. Модель фрактально-глобулярной структуры ДНК. Цветом здесь условно обозначены разные хромосомы.
Соответственно, у одного маленького вируса много возможностей, куда встроиться. В идеале — их бесконечно много, и поэтому найти две клетки, в которых бы вирусы встроились в абсолютно одинаковом месте, — довольно сложно. Еще один важный момент заключается в том, что если зараженная клетка решит поделиться, то обе ее дочерние клетки будут содержать провирус (так называется вирус, который встроился в геномную ДНК) в одном и том же месте генома! Другими словами, генетический маркер — наследуемый признак. Если научиться определять, в какое же именно место генома попал вирус в разных клетках, то можно было бы отличать потомство одной зараженной клетки от другой! И ученые умеют это! Существует несколько методов, позволяющих это сделать, главные из которых — метод саузерн-блоттинга и полимеразная цепная реакция, опосредованная лигированием [7], [8].
Про принцип гибридизации по Саузерну я отсылаю тебя, дорогой читатель, к другой замечательной статье, посвященной описанию важнейших методов молекулярной биологии: «Важнейшие методы молекулярной биологии и генной инженерии» [9]. А о полимеразной цепной реакции расскажет статья «12 методов в картинках: полимеразная цепная реакция» [10].
Оба метода основаны на том, что вследствие случайного характера интеграции вируса в геномную ДНК расстояние от провируса до ближайшего сайта рестрикции будет разным в двух независимо зараженных клетках. Соответственно, если из зараженных клеток выделить ДНК и разрезать ее одной из рестриктаз, то длина фрагмента, содержащего провирус, будет разной у потомства двух разных зараженных клеток, и будет одинаковой у всех клеток, которые произошли в результате деления одной зараженной клетки. Если после рестрикции разделить фрагменты гель-электрофорезом [11], перенести на нитроцеллюлозную мембрану и гибридизовать с короткой последовательностью дезоксирибонуклотидов, комплементарной провирусу (такие олигонуклеотиды принято называть зондами), то на фотобумаге проявится набор полос, который будет одинаковым для клеточного потомства одной клетки и разным — для клеточного потомства нескольких зараженных клеток (рис. 6).
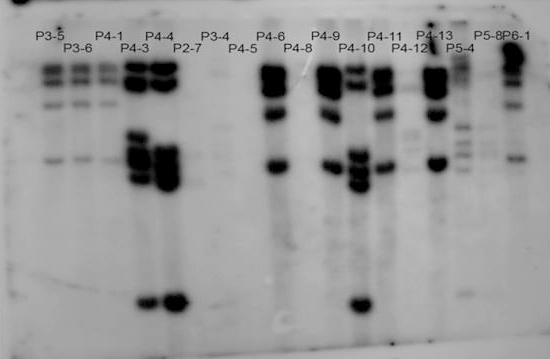
Рисунок 6. Саузерн-блот клеточных клонов, полученных из популяции, зараженной лентивирусным вектором. Каждая дорожка — результат саузерн-блоттинга ДНК из клеточного клона (клеточного потомства одной зараженной клетки). Набор полос в каждой дорожке соответствует продукту гибридизации зонда, комплементарного провирусу, с фрагментами расщепленной ДНК, содержащими провирус. Фрагменты разделены по длине с помощью гель-электрофореза.
собственные данные автора статьи, [4]
Из картинки видно, что на первых трех (и некоторых других дорожках — предлагаю читателю размяться и подсчитать их!) паттерн полос идентичен. Это говорит о том, что ДНК, проанализированная в этих образцах, получена из клеток, произошедших от одной исходно зараженной материнской клетки. Легко также заметить, что дорожка с таинственным названием Р5-4 содержит совершенно другой набор полос. Это означает, что ДНК, использованная здесь, принадлежала другой зараженной клетке. Недостаток такого метода в том, что он не позволяет в явном виде идентифицировать сайт интеграции провируса. Метод позволяет только сравнивать между собой клеточные клоны и судить об их родстве.
Другой метод, который позволяет анализировать сайты интеграции провирусов, — полимеразная цепная реакция, опосредованная лигированием [8]. Это достаточно сложный и многостадийный метод, который включает в себя рестрикцию ДНК, присоединение (лигирование) адаптеров, использование магнитных микрочастиц, ПЦР, электрофорез и секвенирование и заслуживает отдельной статьи. Прошу тебя, мой читатель, поверить мне на слово — этот метод позволяет с точностью до нуклеотида определить место в ДНК, в которое произошла интеграция провируса. С помощью этой методики можно не только различить потомство зараженных клеток между собой, но также судить о том, сколько вирусов проникло в ДНК зараженной клетки, в какие гены произошла интеграция, и не произошло ли изменение их экспрессии в результате внедрения вируса [12], [13].
Ранее я упомянул о том, что в качестве маркера, кроме места интеграции, можно использовать и саму ДНК провируса. Давайте рассмотрим подробнее, как это можно сделать. Здесь нам поможет химический синтез олигонуклеотидов! Дело в том, что в ДНК вируса можно вводить любую информацию: например, ген, кодирующий флуоресцирующий белок или фермент. Правда, тогда все вирусные частицы ничем не будут отличаться одна от другой и приобретут смысл маркеров только тогда, когда попадут в клетку. А почему бы не сделать сами вирусные частицы уникальными? Для этого ученые конструируют лентивирусные векторы, несущие так называемые генетические штрих-коды [14]. Суть заключается в том, что с помощью химического синтеза получают смесь олигонуклеотидов, где в некоторых позициях случайным образом содержится любой из четырех известных дезоксирибонуклеотидов. Дизайн штрих-кода может быть, например, таким:
ATCNNNGGANNNTATNNNCGANNNATTNNNGTGNNN
Здесь символы N обозначают любой из известных нуклеотидов (А, Т, C или G). Такой штрих-код ничего не кодирует, но он представляет собой смесь нуклеотидов с разными последовательностями. Приведенный штрих-код — на самом деле смесь из 418 (или приблизительно 1011 после несложных, но утомительных математических преобразований) различных олигонуклеотидов! Такая смесь используется для сборки вирусов. На выходе получаются уже не обезличенные вирусы, похожие друг на друга как две капли воды, но целая библиотека вирусов, где каждая вирусная частица несет какой-то из возможных олигонуклеотидов-штрих-кодов. Клетки заражают такой вирусной библиотекой, и каждая зараженная клетка получает в свою ДНК уникальный стабильный наследуемый маркер в виде генетического штрих-кода. Все дочерние клетки будут нести тот же штрих-код (или набор штрих-кодов), который приобрела материнская клетка при заражении. Клеточное потомство двух разных зараженных материнских клеток будет иметь различные штрих-коды. Подобно тому, как штрих-коды на товарах в обычном продуктовом магазине позволяют однозначно идентифицировать и отличить один товар от другого, так генетические штрих-коды позволяют отличить потомство одной зараженной клетки от другой. Если затем из зараженной популяции клеток выделить ДНК и определить все присутствующие в ней штрих-коды с помощью новых методов секвенирования, то можно однозначно установить клональный состав клеточной популяции, проследить за динамикой клонов и выявить те клоны, которые преобладают над другими. После заражения библиотекой лентивирусов со штрих-кодами клеточная популяция становится как будто раскрашенной маркерами с очень-очень-очень разнообразной палитрой [15], примерно вот так (рис. 7):
Рисунок 7. Как выглядела бы клеточная популяция, если бы каждый генетический штрих-код имел свой цвет.

Рисунок 8. Канцелярские принадлежности.
сайт kidar.ru
Предлагаю читателю найти 10 отличий с рисунком 8.
Такой подход уже успешно применяется в области изучения стволовых кроветворных клеток. С помощью него установлено, сколько стволовых клеток работают одновременно в организме, чтобы обеспечивать его потребности в клетках крови, как долго функционируют стволовые клетки, и как равномерно они распределяются по разным кроветворным территориям после трансплантации костного мозга [16], [17].
Заключение
Несомненно, охватить все виды клеточных маркеров и способы их определения невозможно в одной статье. Но все же я надеюсь, что мне удалось заинтересовать тебя, читатель, и хотя бы немного раскрыть смысл «маркеров» в биологии и рассказать о том, как ученые «раскрашивают» клетки и для чего они это делают. Современные исследования в области клеточной биологии, гистологии, онкологии немыслимы без определения естественных маркеров. Выявляют всё новые и новые их виды, улучшают методы их визуализации, уточняют профили экспрессии поверхностных антигенов, выявляют корреляции с различными заболеваниями, детально исследуют строение тканей и органов, находят мишени для лечения онкологических заболеваний. И все это благодаря клеточным естественным маркерам. Введение искусственных маркеров предлагает исследователям целый спектр новых методов, открывает новую главу в изучении клеточных популяций на уровне отдельных клеток. Можно быть уверенным, что в ближайшее время генетические маркеры для клеток откроют для человечества новые тайны работы стволовых клеток и функционирования различных органов и тканей [18].
Литература
- HER2 status. (2017). Breastcancer.org;
- Jennifer Laudadio, Denise I Quigley, Raymond Tubbs, Daynna J Wolff. (2007). HER2 testing: a review of detection methodologies and their clinical performance. Expert Review of Molecular Diagnostics. 7, 53-64;
- 12 методов в картинках: проточная цитофлуориметрия;
- Alexey E. Bigildeev, Oxana A. Zhironkina, Irina N. Shipounova, Natalia V. Sats, Svetlana Y. Kotyashova, Nina I. Drize. (2012). Clonal composition of human multipotent mesenchymal stromal cells. Experimental Hematology. 40, 847-856.e4;
- K Weber, U Mock, B Petrowitz, U Bartsch, B Fehse. (2010). Lentiviral gene ontology (LeGO) vectors equipped with novel drug-selectable fluorescent proteins: new building blocks for cell marking and multi-gene analysis. Gene Ther. 17, 511-520;
- Организовать геном: запутанная история гипотез и экспериментов;
- E.M. Southern. (1975). Detection of specific sequences among DNA fragments separated by gel electrophoresis. Journal of Molecular Biology. 98, 503-517;
- Manfred Schmidt, Gesa Hoffmann, Manuela Wissler, Nina Lemke, Arne Müßig, et. al.. (2001). Detection and Direct Genomic Sequencing of Multiple Rare Unknown Flanking DNA in Highly Complex Samples. Human Gene Therapy. 12, 743-749;
- Важнейшие методы молекулярной биологии и генной инженерии;
- 12 методов в картинках: полимеразная цепная реакция;
- 12 методов в картинках: очистка молекул и разделение смесей;
- Kustikova O.S., Baum C., Fehse B. (2008). Retroviral integration site analysis in hematopoietic stem cells. Methods Mol. Biol. 430, 255–267;
- Kustikova O.S., Modlich U., Fehse B. (2009). Retroviral insertion site analysis in dominant haematopoietic clones. Methods Mol. Biol. 506, 373–390;
- Bystrykh L.V., de Haan G., Verovskaya E. (2014). Barcoded vector libraries and retroviral or lentiviral barcoding of hematopoietic stem cells. Methods Mol. Biol. 1185, 345–360;
- Kristoffer Weber, Michael Thomaschewski, Daniel Benten, Boris Fehse. (2012). RGB marking with lentiviral vectors for multicolor clonal cell tracking. Nat Protoc. 5, 839-849;
- E. Verovskaya, M. J. C. Broekhuis, E. Zwart, M. Ritsema, R. van Os, et. al.. (2013). Heterogeneity of young and aged murine hematopoietic stem cells revealed by quantitative clonal analysis using cellular barcoding. Blood. 122, 523-532;
- Evgenia Verovskaya, Mathilde J.C. Broekhuis, Erik Zwart, Ellen Weersing, Martha Ritsema, et. al.. (2014). Asymmetry in skeletal distribution of mouse hematopoietic stem cell clones and their equilibration by mobilizing cytokines. J Exp Med. 211, 487-497;
- A. E. Bigildeev, K. Cornils, T. Aranyossy, N. V. Sats, N. A. Petinati, et. al.. (2016). Investigation of the mesenchymal stem cell compartment by means of a lentiviral barcode library. Biochemistry Moscow. 81, 373-381.