Организовать геном: запутанная история гипотез и экспериментов
09 октября 2017
Организовать геном: запутанная история гипотез и экспериментов
- 3948
- 0
- 12
Всем известно, что огромные геномы организмов остаются организованными. Но как это получается?
-
Автор
-
Редакторы
Молекула ДНК очень длинная, но в клетке находится в очень компактном, «сложенном» состоянии. Как геному удается оставаться организованным и какова его трехмерная структура? О том, как ученые распутывают этот Гордиев узел с помощью новой модели петлевой организации генома, помогает ли альпинизм науке и о самом большом вопросе в биологии генома сегодня — вы узнаете в этой статье. Причем из первых рук: комментарии предоставили Илья Флямер, исследователь из Эдинбургского университета, специализирующийся на пространственной организации генома, и Леонид Мирный, биофизик из Массачусетского технологического университета и один из авторов рассматриваемой модели loop extrusion.
Геном в клетке находится в сильно уплотненном состоянии — это, пожалуй, озвучивается во всех книгах по биологии. Но ведь для этого надо сложить в клеточное ядро 2 метра человеческой ДНК без потери биологической функции [1]! В этом задействованы специальные белки, играющие важную роль в структурировании интерфазных и митотических хромосом и поддерживающие конформацию хроматина на протяжении всего клеточного цикла. Как же тогда выглядит трехмерная структура хромосомы? Как нужные гены остаются активными при такой компактизации? Честно говоря, при попытке ответить на эти вопросы воображение рисует несколько миллионов километров запутанной колючей проволоки.
Однако варианты ответов появились сразу у двух исследовательских групп [2]. Одну из них представляет биофизик Леонид Мирный, заведующий лабораторией MIT [3], а другую — Эрез Либерман Айден (Erez Lieberman Aiden), генетик из Медицинского колледжа Бейлора в Хьюстоне, Техас [4] (рис. 1). Модель назвали loop extrusion — иногда на русский язык ее переводят как «модель растущей петли» или «модель выпетливания», но общепризнанного названия пока нет.

Рисунок 1. Great minds think alike — новые гипотезы петлеобразования хроматина представлены параллельно двумя группами: Леонида Мирного из MIT (слева) и Эреза Либермана Айдена из Медицинского колледжа Бейлора в Хьюстоне (справа).
Несмотря на это, остается ответить еще на многие вопросы, в том числе подтвердить или опровергнуть роль специфических белков, названных авторами моделей. Ким Насмит (Kim Nasmyth), генетик из Оксфордского университета, известный своими работами по разделению хромосом при делении клетки, и один из героев этой статьи, cчитает это «самым большим вопросом в биологии генома на сегодня». Но, если модели окажутся верными, то, по словам знаменитого британского генетика, «это абсолютная революция в энзимологии ДНК». Помимо практического применения, они могли бы предсказать многие процессы, которые пока не удалось увидеть в эксперименте.
Ниже мы погрузимся в историю изучения пространственной структуры ДНК, во все ее взлеты, падения и петли, и вернемся к главному героя рассказа: модели loop extrusion.
Трехмерная модель генома: от азов до последних публикаций
Компактизация ДНК необходима всем организмам для структурирования и передачи своего генома посредством клеточного деления [5]. Например, у эукариот хроматин образует однородные структуры — хромосомы (рис. 2). Их последующее разделение между двумя клетками должно быть очень выверенным, иначе геном дочерних клеток необратимо пострадает. (Повторить клеточный цикл и стадии митоза и мейоза помогут видео — далее рассказывается про структуру ДНК в таких стадиях клеточного цикла, как метафаза и интерфаза)
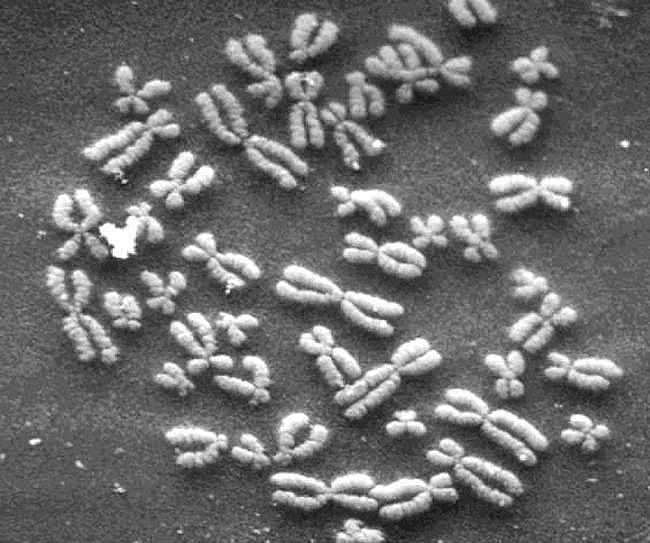
Рисунок 2. Электронная микрофотография четко различимых компактных метафазных хромосом, состоящих из двух хроматид (увеличение 2800×).
сайт home.cc.umanitoba.ca
У эукариот первый уровень уплотнения геномной ДНК — так называемые «бусины на нити», когда ДНК обертывается вокруг комплекса из восьми молекул гистонов с образованием нуклеосом — структурных единиц хроматина. Уже на этом уровне доступность ДНК для ядерных белков и вовлечение в клеточные процессы снижаются — появляется возможность регулировать то, какие гены будут «работать», а какие — «молчать». Этим руководят АТФ-зависимые ферменты, ремоделирующие хроматин [6]. Кроме того, такая минимальная компактизация защищает тонкую нить ДНК от повреждения.
Про роль гистонов и про то, что такое «шаперон гистонов», можно прочитать на «Биомолекуле»: «Катится, катится к ДНК гистон» [7] и «Это FACT» [6].
Менее ясно, как складки хроматина образуют структуры более высокого порядка, к примеру, хромосомы. Кроме того, хотя после обертывания вокруг гистонов нить ДНК становится примерно в пять раз короче, все же в таком виде длина «бусин на нити» — 40 см, и чтобы поместиться в ядро размером 10 микрон, ей надо сложиться еще в 40 000 раз. «Классическая версия укладки» описывала происходящее так: «бусины на нити» составляют фибриллы диаметром 10 нм, а те в свою очередь уложены в фибриллы диаметром 30 нм, причем нуклеосомы оказываются близко друг к другу (иллюстрацию к старой модели можно увидеть, например, в «Википедии»). Однако эта версия уже опровергнута [8]: как комментирует Илья Флямер, исследователь из Эдинбургского университета, специализирующийся на пространственной организации генома, фибриллу диаметром 30 нм видели исключительно in vitro, а в живой клетке ее, по крайней мере, в классическом виде, нет. Более того, в только что вышедшей в Science статье под авторством Клода О’Ши (Clodagh O’Shea) и коллег описано, что хроматин образует гибкие цепи диаметром от 5 до 24 нм без каких-либо фибрилл (рис. 3) [9].

Рисунок 3. Ультраструктура хроматина in situ, трехмерная упаковка и организация ДНК. Хроматин — это структурно неупорядоченная цепочка диаметром от 5 до 24 нм, плотность которой при свертывании варьирует.
В любом случае, в конденсации хроматина в метафазе перед делением (наглядно на видео 1), как считается, задействованы в том числе и два структурно связанных белковых комплекса — конденсин I и II. Совместные действия конденсинов и топоизомеразы II приводят к сборке отдельных митотических хромосом, которые состоят их двух соединенных сестринских хроматид, расходящихся в анафазе. Роль топоизомеразы II в конденсации митотической хромосомы — это удаление «сцепок» внутри любой хромосомы, между сестринскими хроматидами или любой другой парой запутавшихся хромосом.
Видео 1. Уплотнение хромосом и разделение сестринских хроматид в профазе (показан процесс выпетливания)
Очевидно, что для уплотнения митотической хромосомы важно и связывание конденсина с хроматином. Однако детали механизма петлеобразования до последнего времени не прояснялись и активно исследовались учеными [5], [10].
Вейся, веревочка...
То, что ДНК образует петли, известно еще с 1970–80-х годов [11]. Так, опыты по электронной микроскопии метафазных хромосом показали, что большие петли ДНК заякорены в некоторой белковой структуре. Более того, их прикрепление к ней носит регулярный характер [12]. Такие петли — «строительные блоки хромосомы» — можно найти в геноме во время интерфазы у различных видов, например, у человека или дрожжей [13]. При транскрипции в интерфазе подобная организация позволяет регуляторным элементам находиться в непосредственной близости от тех генов, которыми они управляют. Но долгое время оставалось тайной, как же эти петли образуются.
Различные группы ученых независимо друг от друга изучали этот вопрос и разрабатывали свои модели петлевой организации хроматина [2]. Термин DNA reeling — буквально «наматывание ДНК» — предложил американец Артур Риггс (Arthur Riggs) в статье 1990 года [14]. Однако обычно автором всего концепта считают Кима Насмита из Оксфордского университета (Великобритания), который упоминался выше. Его исследовательская группа выяснила, что когезин (комплекс белков, одна из функций которого — скрепление сестринских хроматид) имеет кольцеобразную форму. Насмит в свободное время занимался альпинизмом, и, по его словам, озарение пришло во время восхождения в итальянских Альпах. Глядя на свое снаряжение и то, как веревка была пронизана сквозь карабин, он подумал, что, возможно, то же самое происходит с хромосомой и когезином, не только «обнимающим» кольцом сестринские хроматиды, но и участвующим в формировании петель, которые позволили бы митотическим хромосомам конденсироваться и разделяться. Эта гипотеза был выдвинута в статье 2001 года [15], но ее никто не заметил.
Десять лет спустя появилась математическая модель конденсации хромосом в метафазе за счет loop extrusion («выпетливания»), дополнявшая предположение Насмита. Ее автор — Джон Марко (John Marko) из Северо-западного университета (штат Иллинойс, США) — тоже, по-видимому, не знал о работе британского ученого. И Марко, и Насмит интересовались вопросом плотной упаковки хромосом для клеточного деления.
Здесь стоит упомянуть еще одного исследователя — Джоба Деккера (Job Dekker) из Медицинской школы Массачусетского университета в Вустере. Его и коллег интересовали взаимосвязи активности генов и хромосомной организации — причем в масштабе всего генома. Для изучения этих вопросов ученые из лаборатории Деккера разработали метод Hi-C [16] (видео о том, как он работает, дано в разделе про методы изучения генома), который позволяет обнаружить то, как часто какие-либо участки генома оказываются рядом, даже если они расположены далеко друг от друга на одной хромосоме или даже на разных хромосомах.
«Это единственный подход, который позволяет анализировать пространственную организацию всего генома одновременно, — комментирует Илья Флямер, специалист из лаборатории Венди Бикмор (Wendy Bickmore) Эдинбургского университета. — С помощью этого метода в лаборатории Бинга Рена (Bing Ren) обнаружили, что геном поделен на участки длиной 0,2–1 млн пар нуклеотидов с повышенной частотой контактов внутри самих себя [17]. Их назвали TADs, topologically associating domains — „топологически ассоциированные домены“». Интересно, что открытие ТАДов тоже было совершено двумя группами параллельно — другой работой руководили Джоб Деккер и Эдит Херд (Edith Heard) [18].
Только что открытые домены заинтересовали одного из главных героев нашего рассказа — Леонида Мирного, биофизика из MIT. Мирный встретился на конференции с Марко, где тот рассказывал о своей модели образования петель для плотной упаковки ДНК в метафазе. В этот момент сотрудники лаборатории Мирного и Деккера пытались построить компьютерную модель, объясняющую Hi-C-данные для плотноупакованной метафазной хромосомы. «Тогда мы поняли, что механизм, предложенный Марко — активное формирование петель молекулярными моторами, — может позволить создать ряды петель ДНК, которые мы получали в своей модели, — делится Леонид Мирный. — Опубликованная в конце 2013 года статья показала, что метафазные хромосомы в клетке состоят из таких рядов плотно уложенных петель, которые видели еще в 80-е годы».
Мирный погрузился в изучения ТАДов — и далее наш рассказ смещается из метафазы хромосом в интерфазу, то есть из той фазы клеточного цикла, когда происходит деление клетки, в фазу роста клетки и синтеза белков. Как ученый рассказал «Биомолекуле», он с коллегами пытался объяснить образование ТАДов с помощью компьютерного моделирования: «В 2014 году сотрудники нашей лаборатории Максим Имакаев и Джеф Фюденберг занимались поиском модели формирования ТАДов. Они решили проверить, нет ли и в интерфазе молекулярных моторов, подобных тем, что упаковывают ДНК в плотные метафазные хромосомы. Эта гипотеза получила название model of loop extrusion. Как оказалось, такой механизм возникновения доменов — рост петель за счет активности молекулярных моторов и их остановка на границах доменов — идеально воспроизводила многие данные, полученные с помощью Hi-C. Физический же акт формирования петель хорошо организовывал локальные домены. Параллельно другой сотрудник моей лаборатории, Антон Голобородько в сотрудничестве с Марко показал, что в компьютерных моделях молекулярные моторы, создающие петли (видео 2), действительно могут формировать плотные и длинные метафазные хромосомы (видео 3), и при помощи топоизомеразы II разделять сестринские хромосомы (видео 4), как и предполагал Насмит».
Видео 2. Введение в модель loop extrusion
Видео 3. Формирование одиночной метафазной хромосомы
Видео 4. Сегрегация сестринских хроматид
Итак, в мае 2015 года Мирный рассказывает о своей работе на конференции в Институте Вейцмана (Weizmann Institute of Science) в Израиле, где встретились все ведущие ученые из этой области, включая Эреза Либермана Айдена, Эдит Херд и других героев этой истории. В августе того же года статья Мирного и коллег нашла своего читателя на сайте bioRxiv в виде препринта, а в 2016 году была опубликована в Cell Reports [3]. Главной загадкой было и остается то, что же это за моторы, которые растят петли, и как они это делают. Как предполагалось в тот момент, когезин играл свою роль в удержании сестринских хромосом, скорее, как карабин Насмита, чем как мотор.
Лаборатория Мирного же выдвинула гипотезу, что когезин играет главную роль в создании петель в интерфазе как мотор, способный наращивать петли, когда хромосомы «распущены». Конденсин же, как оговаривалось в совсем недавней статье Деккера и Мирного [10], выполнял ту же функцию во время деления клеток при плотной конденсации хромосом.
Новая модель loop extrusion объясняет физическое взаимодействие частей генома, наблюдаемое в экспериментах: и регуляцию экспрессии генов, и многие аспекты развития клеток и различных болезней. Это достигается за счет способности новой гипотезы показывать, как ДНК может образовывать петли с помощью специальных молекулярных машин. Такой метод компактизации генома не только придает форму и структуру хромосомам, но и сближает нужные участки ДНК друг с другом.
Одним из главных действующих лиц оказался белок CTCF. Было известно, что он взаимодействует с когезином в основании петель неконденсированных хромосом. Долгое время считалось, что петли ДНК образуются тогда, когда любые CTCF случайно сталкиваются друг с другом и соединяются вместе.
Как рассказывает Илья Флямер, в работе лаборатории Либермана Айдена 2014 года с использованием Hi-C метода были были картированы петли в очень высоком разрешении [19]. Установили, что в основании петель CTСF находится на сайтах строго определенной ориентации: «Сайт посадки CTCF асимметричен, и это определяет возможное направление петель, которые этот сайт может образовать. Такая преференция не может возникнуть при случайной диффузии и встрече белков — они будут встречаться случайными сторонами и формировать петли вне зависимости от направления их сайта посадки».
Как предполагается в модели Мирного и коллег, белки CTCF — это знаки «стоп» для когезина. Если когезин доходит до определенным образом ориентированного CTCF с каждой стороны растущей петли, то эти белки оказываются сведенными вместе, и когезин прекращает пропускать ДНК (рис. 4).
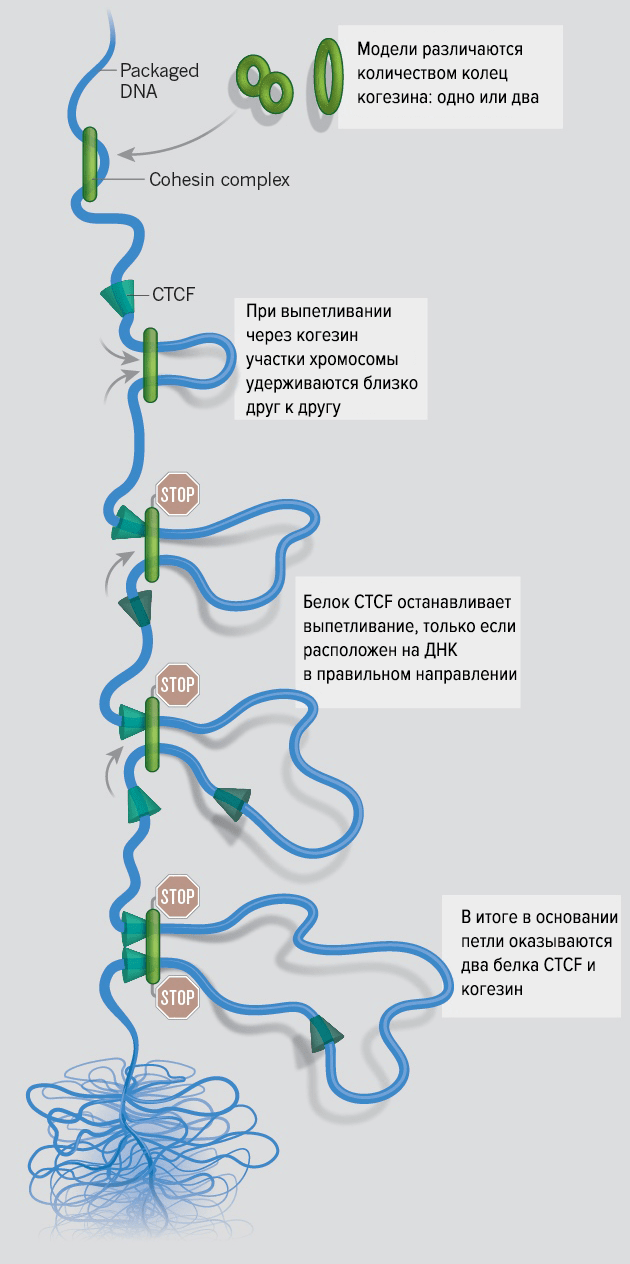
Рисунок 4. Наглядное изображение модели петлеобразования в геноме и роль CTCF и когезина. Модель геномной организации loop extrusion помогает объяснить, как определенные участки хромосом в интерфазе оказываются сближены и почему кольца протеинового комплекса когезина часто находятся в контакте с белками CTCF, которые связываются с определенными последовательностями ДНК.
Такое описание функции когезина помогает объяснить некоторые экспериментальные данные. Например, что когезин влияет на размер хроматиновых петель [20]. Повышенная его активность приводит к производству такого количества петель, что хромосомы становятся похожи на маленьких червячков (за это их даже окрестили «вермишельками» — vermicelli [21], видом пасты, которая с итальянского буквально переводится как «маленькие червячки»; увидеть их при окрашивании ДНК можно в только что вышедших препринтах [22], [23]).
Теперь остается ждать прямого подтверждения, что этот комплекс белков работает именно так. Ведь пока что утверждение о роли когезина как мотора, способного наращивать петли, приходится принять на веру, поскольку его поведение, описанное в модели, еще не подтверждили ни в живых клетках, ни даже in vitro [2]. Совсем недавняя работа, однако, показала, что близкий родственник когезина — конденсин, необходимый для упаковки хромосом и главный герой гипотез Насмита, Марко и Мирного про метафазные хромосомы, — ведет себя как мотор in vitro [24].
«Всю важность процесса петлеобразования ученые пока оценить не могут, — рассказывает „Биомолекуле“ Илья Флямер, — однако есть предположение, что это — ключ к разгадке механизма действия энхансеров. Также он может участвовать в поддержании незапутанного состояния хромосом и предотвращении двухцепочечных разрывов, особенно при делении. Более того, вероятно, что аналогичный процесс с конденсином вместо когезина обеспечивает конденсацию хромосом».
Версия Либермана Айдена: параллельная гипотеза
Как уже было сказано, параллельно гипотезу петлеобразования разрабатывал Эрез Либерман Айден из Хьюстонского Медицинского колледжа Бейлора (видео 6). По его словам, догадка о роли когезина и CTCF возникла у него во время телефонной конференции в марте 2015 года при обсуждении подробностей структуры генома, полученных с помощью Hi-C-метода им и его бывшим руководителем Эриком Ландером.
Видео 6. Модель петлеобразования в ДНК, предложенная группой Либермана Айдена
После многочисленных проверок гипотезы его группа подала статью на публикацию в июле 2015 года. В итоге работа была опубликована три месяца спустя (см. видео 6) [4]. В общем, обе модели — и Мирного, и Либермана Айдена — приходят примерно к одинаковым выводам.
Хотя экспериментальных данных в работе Мирного было меньше, его группа для подкрепления своей гипотезы включила в работу компьютерную модель. Некоторых это заставляет сомневаться в том, что работа Либермана Айдена (который, по словам Мирного, слышал его доклад в Институте Вейцманна и не сослался на статью из биоархива) была разработана полностью самостоятельно. Сам же ученый непреклонен. Он утверждает, что заявка на публикацию была подана до того, как он увидел труд Мирного и его группы.
Различия гипотез
Расхождения двух моделей незначительны [2]. Например, в гипотезе Мирного количество колец не уточняется. В версии Либермана Айдена же — это два соединенных кольца. Хотя различие выглядит довольно механическим, специалисты называют его крайне важным для определения роли этого комплекса.
Другое различие гипотез — по мнению Мирного, у когезина есть моторная функция, не имеющая отношения к кольцевой форме когезина-карабина. Либерман Айден, в целом, эту идею отвергает. Однако оба предположения возможны, и существуют данные, поддерживающие каждую из сторон. В частности, то, что когезин работает как мотор, подтверждает эксперимент под руководством Дэвида Руднера (David Rudner) — биолога, изучающего бактериальные клетки на медицинском факультете Гарварда (штат Массачусетс, США). Его группа сделала карту разных моментов складывания хромосомы бактерии Bacillus subtilis при использовании метода Hi-C. На ней исследователи увидели работу комплексов структурного поддержания хромосом (SMC) — бактериальных родственников когезина и конденсина — и то, что при этом создаются петли со скоростью более 50 000 пар нуклеотидов в минуту. По мнению ученых, этот темп примерно совпадает с тем, который должен бы быть в модели Мирного, примененной к клеткам человека. Пока что затрата АТФ в этом процессе не доказана, но Руднер считает, что он близок к этому. Более того, по его словам, он был бы шокирован, если бы когезин в клетках человека работал иначе.
Гипотезу Либермана Айдена поддерживают данные о малом потреблении энергии комплексом при захвате и высвобождении ДНК, что затрудняет его работу на тех скоростях, которые предполагаются в модели Мирного. Возможно, есть иные молекулы, которые помогают скольжению когезина вдоль генома на большие расстояния (например, РНК-полимеразы, которые транскрибируют гены при производстве РНК). Согласно другому мнению, моторные белки вообще не нужны для производства петель. Когезин может перемещаться по ДНК случайным образом до тех пор, пока не встретит CTCF и не сделает петлю.
Верить ли безоговорочно новой модели?
Среди исследователей сейчас идет академический спор: насколько признавать новую модель loop extrusion? Хотя множество факторов говорит в пользу того, что группы Мирного и Либермана Айдена правы, окончательно признавать когезин главным героем петлеобразования еще рано [13]. Многие исследователи призывают к здоровому скептицизму до окончательного подтверждения модели, как, например, Даг Кошланд (Doug Koshland) из Калифорнийского университета в Беркли. По словам ученого, его волнует преждевременное возведение гипотезы на пьедестал и то, что из-за своей простоты и элегантности она уже попадает в учебники [2].
«Я думаю, что эта модель прорывная! И, на мой взгляд, она, скорее всего, верна — слишком хорошо объясняет данные относительно когезина и CTCF», — делится с «Биомолекулой» Илья Флямер.
Однако для начала необходимо как минимум установить точные молекулярные механизмы, ведущие к «пропусканию» ДНК, и пути регуляции этого процесса. Кроме того, нужен и детальный анализ роли в формировании петель вспомогательных белков и CTCF и их взаимодействия с хроматином. По словам Мирного, на часть вопросов сможет ответить проверка функции когезина и CTCF в клетках людей, страдающими заболеваниями, где работа этих молекул нарушена, например, из-за мутации. С другой стороны, исследователь говорит, что не может объяснить образование петель никаким другим образом.
Литература
- Revealed: the human genome in 3D. (2009). BBC News;
- Dolgin E. (2017). DNA's secret weapon against knots and tangles. Nature News;
- Geoffrey Fudenberg, Maxim Imakaev, Carolyn Lu, Anton Goloborodko, Nezar Abdennur, Leonid A. Mirny. (2016). Formation of Chromosomal Domains by Loop Extrusion. Cell Reports. 15, 2038-2049;
- Adrian L. Sanborn, Suhas S. P. Rao, Su-Chen Huang, Neva C. Durand, Miriam H. Huntley, et. al.. (2015). Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proc Natl Acad Sci USA. 112, E6456-E6465;
- Xavier Robellet, Vincent Vanoosthuyse, Pascal Bernard. (2017). The loading of condensin in the context of chromatin. Curr Genet. 63, 577-589;
- Это FACT;
- Катится, катится к ДНК гистон;
- Sergey V Razin, Alexey A Gavrilov. (2014). Chromatin without the 30-nm fiber. Epigenetics. 9, 653-657;
- Horng D. Ou, Sébastien Phan, Thomas J. Deerinck, Andrea Thor, Mark H. Ellisman, Clodagh C. O’Shea. (2017). ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science. 357, eaag0025;
- Gibcus J.H., Samejima K., Goloborodko A., Samejima I., Naumova N., Kanemaki M. et al. (2017). Mitotic chromosomes fold by condensin-dependent helical winding of chromatin loop arrays. bioRxiv;
- Earnshaw W.C. and Laemmli U.K. (1983). Architecture of metaphase chromosomes and chromosome scaffolds. J. Cell Biol. 96, 84–93;
- O Iarovaia, R Hancock, M Lagarkova, R Miassod, S V Razin. (1996). Mapping of genomic DNA loop organization in a 500-kilobase region of the Drosophila X chromosome by the topoisomerase II-mediated DNA loop excision protocol.. Mol. Cell. Biol.. 16, 302-308;
- Christopher Barrington, Ronald Finn, Suzana Hadjur. (2017). Cohesin biology meets the loop extrusion model. Chromosome Res. 25, 51-60;
- A. D. Riggs. (1990). DNA Methylation and Late Replication Probably Aid Cell Memory, and Type 1 DNA Reeling Could Aid Chromosome Folding and Enhancer Function. Philosophical Transactions of the Royal Society B: Biological Sciences. 326, 285-297;
- Kim Nasmyth. (2001). Disseminating the Genome: Joining, Resolving, and Separating Sister Chromatids During Mitosis and Meiosis. Annu. Rev. Genet.. 35, 673-745;
- E. Lieberman-Aiden, N. L. van Berkum, L. Williams, M. Imakaev, T. Ragoczy, et. al.. (2009). Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome. Science. 326, 289-293;
- Jesse R. Dixon, Siddarth Selvaraj, Feng Yue, Audrey Kim, Yan Li, et. al.. (2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485, 376-380;
- Elphège P. Nora, Bryan R. Lajoie, Edda G. Schulz, Luca Giorgetti, Ikuhiro Okamoto, et. al.. (2012). Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature. 485, 381-385;
- Suhas S.P. Rao, Miriam H. Huntley, Neva C. Durand, Elena K. Stamenova, Ivan D. Bochkov, et. al.. (2014). A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping. Cell. 159, 1665-1680;
- Judith H.I. Haarhuis, Robin H. van der Weide, Vincent A. Blomen, J. Omar Yáñez-Cuna, Mario Amendola, et. al.. (2017). The Cohesin Release Factor WAPL Restricts Chromatin Loop Extension. Cell. 169, 693-707.e14;
- Antonio Tedeschi, Gordana Wutz, Sébastien Huet, Markus Jaritz, Annelie Wuensche, et. al.. (2013). Wapl is an essential regulator of chromatin structure and chromosome segregation. Nature. 501, 564-568;
- Gassler J., Brandao H.B., Imakaev M., Flyamer I.M., Ladstatter S., Bickmore W.A. et al. (2017). A mechanism of cohesin-dependent loop extrusion organizes zygotic genome architecture. bioRxiv;
- Wutz G., Varnai C., Nagasaka K., Cisneros D.A., Stocsits R., Tang W. et al. (2017). CTCF, WAPL and PDS5 proteins control the formation of TADs and loops by cohesin. bioRxiv;
- Terekawa T., Bisht S., Eeftens J., Dekker C., Haering C., Greene E. (2017). The condensin complex is a mechanochemical motor that translocates along DNA. bioRxiv;
- Баттулин Н.Р., Фишман В.С., Орлов Ю.Л., Мензоров А.Г., Афонников Д.А., Серов О.Л. (2012). 3С-методы в исследованиях пространственной организации генома. Вавиловский журнал генетики и селекции. 16, 872–878;
- Половая жизнь хроматина;
- Anat Vivante, Eugene Brozgol, Irena Bronshtein, Yuval Garini. (2017). Genome organization in the nucleus: From dynamic measurements to a functional model. Methods. 123, 128-137;
- Paul J. Flory. (1936). Molecular Size Distribution in Linear Condensation Polymers1. J. Am. Chem. Soc.. 58, 1877-1885;
- Paul J. Flory. (1941). Molecular Size Distribution in Three Dimensional Polymers. I. Gelation1. J. Am. Chem. Soc.. 63, 3083-3090;
- Paul J. Flory. (1949). The Configuration of Real Polymer Chains. The Journal of Chemical Physics. 17, 303-310;
- Распутать узлы. Исследования российских ученых позволяют решать задачи в разных областях. (2013). Сайт МГУ;
- Лифшиц И.М., Гросберг А.Ю., Хохлов А.Р. (1979). Объемные взаимодействия в статистической физике полимерной макромолекулы. Успехи физических наук. 127, 353–389;
- A. Yu. Grosberg, S.K. Nechaev, E.I. Shakhnovich. (1988). The role of topological constraints in the kinetics of collapse of macromolecules. J. Phys. France. 49, 2095-2100;
- A Grosberg, Y Rabin, S Havlin, A Neer. (1993). Crumpled Globule Model of the Three-Dimensional Structure of DNA. Europhys. Lett.. 23, 373-378;
- SERGEI NECHAEV, OLEG VASILYEV. (2005). ON TOPOLOGICAL CORRELATIONS IN TRIVIAL KNOTS: FROM BROWNIAN BRIDGES TO CRUMPLED GLOBULES. J. Knot Theory Ramifications. 14, 243-263;
- Christian Münkel, Jörg Langowski. (1998). Chromosome structure predicted by a polymer model. Phys. Rev. E. 57, 5888-5896;
- J. Mateos-Langerak, M. Bohn, W. de Leeuw, O. Giromus, E. M. M. Manders, et. al.. (2009). Spatially confined folding of chromatin in the interphase nucleus. Proceedings of the National Academy of Sciences. 106, 3812-3817;
- Макаров В. (2016). Наглядная 3D-модель организации ДНК внутри ядра. «Популярная механика».