
Нанопоровое секвенирование: на пороге третьей геномной революции
22 июня 2018
Нанопоровое секвенирование: на пороге третьей геномной революции
- 10041
- 0
- 15
Нанопоровое секвенирование является одной из наиболее перспективных современных технологий определения последовательности нуклеиновых кислот, позволяя решать многочисленные задачи, стоящие перед биологами
иллюстрация Елены Беловой
-
Автор
-
Редакторы
-
Иллюстратор
Публикация первого генома человека в 2001 году стала предвестником постгеномной эры — появление технологий секвенирования нового поколения (next-generation sequencing, NGS) позволило поверить в будущее персонифицированной геномики. Сегодня, спустя более 15 лет, коммерциализация приборов, чья работа основана на нанопоровом секвенировании, делает это будущее реальностью. Давайте же обсудим, чем так привлекательна новая технология.
Партнер публикации этой статьи — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Публикация логически продолжает статью «Секвенирование нуклеиновых кислот» из цикла «12 биологических методов в картинках», также поддержанного «Диаэм».
Секвенирование ДНК — рутинная процедура, позволяющая определять нуклеотидную последовательность этой макромолекулы. Современные методы полногеномного секвенирования делают возможным считывать миллиарды последовательностей ДНК за считанные часы — буква за буквой, ген за геном и хромосома за хромосомой. Уже отсеквенированы десятки тысяч геномов, начиная от вирусных и заканчивая геномами человека, полиплоидных и даже вымерших видов. Всё это происходит благодаря новейшим полногеномным технологиям, о которых я написал и опубликовал на «Биомолекуле» большой обзор: «12 методов в картинках: секвенирование нуклеиновых кислот» [1]. Поэтому, если вы желаете поглубже окунуться в дебри современного, и не очень, секвенирования, рекомендую обратиться сначала к нему.
Но время, впрочем как и развитие технологий, не стоит на месте, и в жизни каждого из нас нередко встает проблема, для решения которой необходимо перелопатить десятки публикаций и книг. Совсем недавно с подобными сложностями столкнулись я и мои коллеги из лаборатории палео- и этногенетики НИЦ «Курчатовский институт» в попытке впервые в мире собрать полиплоидный геном одного, находящегося на грани вымирания, организма, который ни в какую не хотел объединяться в единое целое. Знающие люди посоветовали нам присмотреться к нанопоровому секвенированию — решению, позволяющему прочитывать длинные молекулы ДНК за приемлемые деньги. Недолго думая, мы решили разобраться, какие же преимущества сулит эта относительно новая технология, и пробежались по уже опубликованным работам, оценив весь спектр предложений геномных секвенаторов Oxford Nanopore Technologies, поставляемых на российский рынок компанией «Диаэм».
Нанопоровое секвенирование — история и современное состояние. Плюсы и минусы
Знакомясь с историей появления нанопорового секвенирования, можно еще раз убедиться в необходимости как разумного государственного финансирования фундаментальной науки, так и венчурных инвестиций в идеи и прототипы, неожиданно прорезающиеся посреди суровых формул, запутанных биохимических реакций и сложных молекулярных структур.
Первые шаги к созданию технологии нанопорового секвенирования были сделаны более двадцати лет назад, когда группа американских ученых, используя напряжение электрического поля, научилась «протаскивать» молекулы ДНК и РНК — как нитку через игольное ушко — сквозь ионный канал диаметром 2,6 нанометра, закрепленный в бислойной липидной мембране. Наверное, на переломе XX и XXI веков эта работа могла бы показаться обычным физико-химическим методом изучения макромолекул, но уже тогда исследователи сумели различать молекулы ДНК и РНК, а также оценивать их длину [2]. В 2001 году, используя этот подход, стало возможным отличать друг от друга короткие одноцепочечные ДНК, содержащие отдельные нуклеотидные замены [3], а уже в 2009 году нанопоровое секвенирование стало явью — сотрудники Оксфордского университета научились различать все четыре нуклеотида в одноцепочечной ДНК по мере их прохождения через пору [4], что предвосхитило появление первого секвенатора от компании Oxford Nanopore Technologies в 2014 году.
Технология, предложенная британской компанией и внедренная в её секвенирующие устройства, достаточно проста. Молекула ДНК или РНК, несущая на себе существенный отрицательный заряд, под воздействием электрического поля «протискивается» через белковую пору, помещенную в бислойную мембрану, что вызывает уменьшение силы электрического тока из-за уменьшения сечения отверстия (рис. 1 и видео). Считывая изменение силы тока, нанопоровый секвенатор определяет нуклеотид, проходящий через пору в конкретный отрезок времени: оказывается, что каждый «кирпичик» в нуклеиновой кислоте имеет свое собственное «сечение» (и, соответственно, это влияет на силу тока).
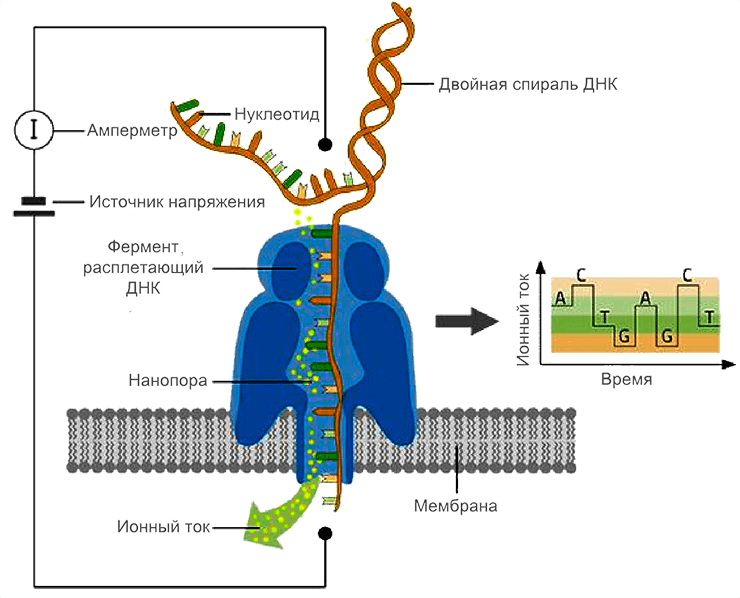
Рисунок 1. Белковая нанопора, закрепленная в бислойной мембране, позволяет считывать десятки тысяч «букв» за один запуск секвенатора
Видео. Принцип работы нанопорового секвенатора
Методика, реализованная в устройствах MinION, GridION X5, PromethION и SmidgION, как и любая другая, имеет и плюсы, и минусы. В первую очередь, от нее не следует ждать такой же производительности, как от последних версий геномных «фабрик», выведенных на биотехнологический рынок компанией Illumina: ведь степень параллелизма в нанопоровом секвенировании гораздо ниже, да и само «протискивание» через нанопору — процесс пока не настолько производительный. Однако современные приборы от Oxford Nanopore имеют и сильные стороны.
В первую очередь это длина прочтений, которая может достигать 100 тысяч нуклеотидов и представляется недостижимой для других технологических платформ. Так, многие коммерциализированные к настоящему времени методики секвенирования «нового поколения» основываются на многократном и одновременном прочтении коротких последовательностей ДНК (до 300 нуклеотидов), что позволяет с одной стороны значительно ускорить сложный ферментативный процесс, а с другой — значительно увеличить копийность (глубину покрытия генома). Данные, которые исследователи получают в результате такого секвенирования, больше походят на «геномный паззл», а не на разложенный по хромосомам геном.
Длина прочтения в десятки тысяч нуклеотидов, предлагаемая нанопоровым секвенированием, позволяет не только значительно упростить и ускорить сборку ранее не опубликованных геномов, но и улучшить уже существующие сборки. Следует особо отметить, что такие секвенаторы не испытывают «аллергии» на участки с повышенным содержанием гомополимеров и на GC-богатые области генома, в отличие от многих альтернативных технологий.
Не секрет, что не все геномы собраны настолько же идеально, как, допустим, человеческий или мышиный. Большинство из них, по сути, представляют собой великое множество длинных фрагментов (контигов), расположение которых на хромосоме неизвестно, поэтому доступность длинных прочтений значительно упростит процесс de novo сборки генома, который состоит из сложения единичных фрагментов в цельную последовательность [5].
В то же время, вместе с плюсом длинных прочтений, методику нанопорового секвенирования неизбежно характеризует бóльшее (относительно других современных платформ) число ошибок секвенирования, хотя эта проблема, по всей видимости, активно решается. В 2015 году предварительно сообщали о почти сорока процентах ошибок в прочтениях секвенатора MinION [6], а по последним данным оно сократилось до почти приемлемых 10% [7]. Но и это еще не всё — компания Oxford Nanopore Technologies приготовила специальный набор для пробоподготовки 1D2, который в итоге позволяет считывать обе, комплементарные друг другу, цепи ДНК, что увеличивает точность секвенирования почти до 99%.
Кроме того, нанопоровое секвенирование при достаточном количестве предварительно экстрагированного из биологического образца генетического материала позволяет отказаться от ПЦР : отсутствие стадии амплификации ДНК при пробоподготовке помогает избежать выборочного обогащения определенных участков генома, а также устраняет необходимость длительной и не самой простой пробоподготовки, например, при изучении модификаций нуклеотидов в геноме [8]. Отсутствие стадии ПЦР и упрощение используемых реагентов делают исследователя более мобильным и значительно удешевляет процесс пробоподготовки и секвенирования. Размер нанопоровых секвенаторов делает их крайне утилитарными устройствами, позволяя проводить исследования в самых неожиданных местах — Экваториальной Африке, Антарктиде или в космосе, — буквально, «на коленке»: маркетологи не преминули воспользоваться этим преимуществом, именуя самый миниатюрный из них (MinION) секвенатором-флешкой (рис. 2).
Что кроется за аббревиатурой «ПЦР», вы можете узнать из нашего обзора «Полимеразная цепная реакция», входящего в цикл «12 биологических методов в картинках» [9]. — Ред.

Рисунок 2. Нанесение пробы на проточную ячейку секвенатора MinION
Неоспоримым преимуществом нанопорового секвенирования является также анализ полученных данных в режиме реального времени — исследователь в любой момент запуска может понять, что процесс идет не совсем так, как хотелось бы, остановить секвенирование и нанести на проточную ячейку секвенатора новый образец.
Оценивая плюсы и минусы платформы, можно смело сказать, что у нанопорового секвенирования неплохое будущее на мировом биотехнологическом рынке, где оно может уже сегодня найти применение и как дополнительная, и как самостоятельная секвенирующая платформа. Несмотря на очевидно высокий процент ошибок, небольшая цена приборов и реагентов к ним, простота использования, а также уникально длинные прочтения сделают этот девайс безусловным лидером продаж, а также подтолкнут компанию Oxford Nanopore к дальнейшему развитию линейки.
Современные приборы нанопорового секвенирования Oxford Nanopore Technologies
Технология нанопорового секвенирования, реализованная в приборах, поставляемых британской компанией Oxford Nanopore Technologies (официальный дистрибьютор в России и СНГ — компания «Диаэм»), по своему принципу кардинально отличается от уже существующих платформ, а также славится своей универсальностью, компактностью и простотой использования.
Так, простейший секвенатор MinION весит каких-то сто граммов и работает от USB 3.0 настольного компьютера, позволяя, например, секвенировать за один запуск геном человека с шестикратным покрытием.
Для большей производительности Oxford Nanopore Technologies разработал приборы, в которые можно параллельно установить сразу несколько проточных ячеек (продуктовая линейка изображена на рисунке 3):
- GridION имеет модуль для пяти проточных ячеек и поставляется со специальным компьютером для обработки получаемых данных;
- PromethION скоро появится на рынке и будет иметь модуль для 48 проточных ячеек, каждая из которых несет на себе три тысячи нанопор;
- SmigdIOn — прибор, готовящийся к выпуску, призванный быть наименьшим из приборов для нанопорового секвенирования и работать в паре со смартфоном.

Рисунок 3. Линейка геномных секвенаторов Oxford Nanopore Technologies, поставляемых на российский рынок компанией «Диаэм»
Кроме самих секвенаторов при работе со значительным количеством образцов компания Oxford Nanopore Technologies рекомендует дополнительно приобрести автоматизированную станцию для пробоподготовки VolTRAX, позволяющую полностью отстраниться от этого процесса. VolTRAX берет на себя все заботы по приготовлению биологических образцов к секвенированию, начиная от выделения ДНК или РНК из ткани и заканчивая приготовлением ДНК-библиотек.
Опытные геномщики, конечно, зададутся вопросом обсчета данных, но и тут ожидает приятный сюрприз — с 2018 года им не придется подбирать ноутбук с необходимыми параметрами или по крупицам собирать мощный настольный компьютер; для удобства своих пользователей Oxford Nanopore Technologies анонсировал продажу специального миниатюрного (менее 300 граммов), но крайне производительного портативного компьютера MinIT, позволяющего обрабатывать данные в любой точке мира.
Применение нанопорового секвенирования
Продолжая свое знакомство с нанопоровым секвенированием и приборами, использующими эту технологию, я не преминул оценить возможные перспективы внедрения приборов компании Oxford Nanopore Technologies в научную работу, одним из направлений которой является сборка геномов видов животных, находящихся на грани уничтожения. Для этого мы пробежались по статьям, использующим эту технологическую платформу.
В погоне за патогеном
В первую очередь новой платформой заинтересовались исследователи, занимающиеся геномикой микроорганизмов. Лишенные длинных прочтений секвенаторами GS FLX и 454 [10] производства Roche (поддержка платформы остановилась в 2016 году), они выбирают длинные фрагменты, генерируемые нанопоровыми секвенаторами, которые при многократном прочтении и использовании специального биоинформатического инструментария позволяют снизить процент ошибок (хотя и с некоторым допущениями) до оптимального, в том числе при комбинации этой технологической платформы с Illumina [11], [12].
В то же время понятно, что приборы Oxford Nanopore Technologies имеют огромный потенциал и уже сейчас могут применяться для быстрой идентификации вирусных и бактериальных патогенов [13], [14], а также для анализа в реальном времени сложных метагеномных образцов из окружающей среды, причем в самых необычных условиях — на антарктической станции и даже в космосе [15], [16].
Нанопора и альтернативный сплайсинг
Первоначально, в силу своей относительно невысокой производительности, казалось, что нанопоровое секвенирование найдёт скромную нишу лишь в геномике вирусов и микроорганизмов (ведь их геномы невелики, в отличие от геномов эукариот), однако, как выяснилось позднее, длинные фрагменты ДНК или РНК, пусть и не всегда идеально прочитанные, необходимы во многих других приложениях современной молекулярной биологии. Так, длинные прочтения позволяют упростить изучение альтернативного сплайсинга , биоинформатический анализ которого при использовании стандартных NGS-методов требует значительных трудозатрат. Использование приборов Oxford Nanopore Technologies в этой задаче незаменимо, поскольку позволяет обнаруживать сотни ранее неизвестных альтернативных сплайс-вариантов генов, в том числе на уровне отдельных клеток [17], что с развитием технологии сулит новые открытия, которые, вероятно, перепишут современные неидеальные подходы анализа экспрессии генов.
Процесс «дозревания» молекул РНК, в результате которого из пре-мРНК (предшественника мРНК) образуется несколько зрелых мРНК. — Прим. авт.
Преимущества для de novo сборки
Нельзя также не отметить возможность использования длинных прочтений при работе с de novo собранными геномами или транскриптомами, и пусть до похромосомной сборки еще далековато, но комбинация длинных прочтений с данными традиционных NGS-платформ позволяет увеличивать параметры сборки на порядки [18–20]. Нанопоровое секвенирование проникает и в Россию: так, исследователи биологического факультета Санкт-Петербургского государственного университета уже заявили о значительном улучшении качества и размера сборки генома пресноводной мшанки Cristatella mucedo при комбинировании данных стандартного NGS и нанопорового секвенирования.
Медицина и криминалистика
Быстрое и относительно продуктивное секвенирование в реальном времени сулит замечательные прорывы не только в фундаментальных исследованиях, но и в медицине и биотехнологии, поскольку позволяют оперативно решать насущные задачи — например, проводить предимплантационную генетическую диагностику эмбрионов [21] или судебно-криминалистическую экспертизу [22].
Загадки эпигенома
Кроме собственно определения последовательности нуклеиновых кислот, нанопоровое секвенирование способно оценивать степень модификации нуклеотидов в геноме, выявляя метилирование ДНК — присоединение метильной группы (—CH3) к цитозину в составе CpG-динуклеотида. В частности, приборы от Oxford Nanopore Technologies позволяют идентифицировать метилированные азотистые основания 5-метилцитозин и 5-гидроксиметилцитозин, таким образом значительно облегчая эпигеномные исследования [23].
Один из способов регуляции работы генома без изменения нуклеотидной последовательности ДНК. Больше об эпигенетике вы узнаете из статей «Эпигеном: параллельная реальность внутри клетки» и «Эпигенетика: невидимый командир генома» на «Биомолекуле» [24], [25]. — Ред.
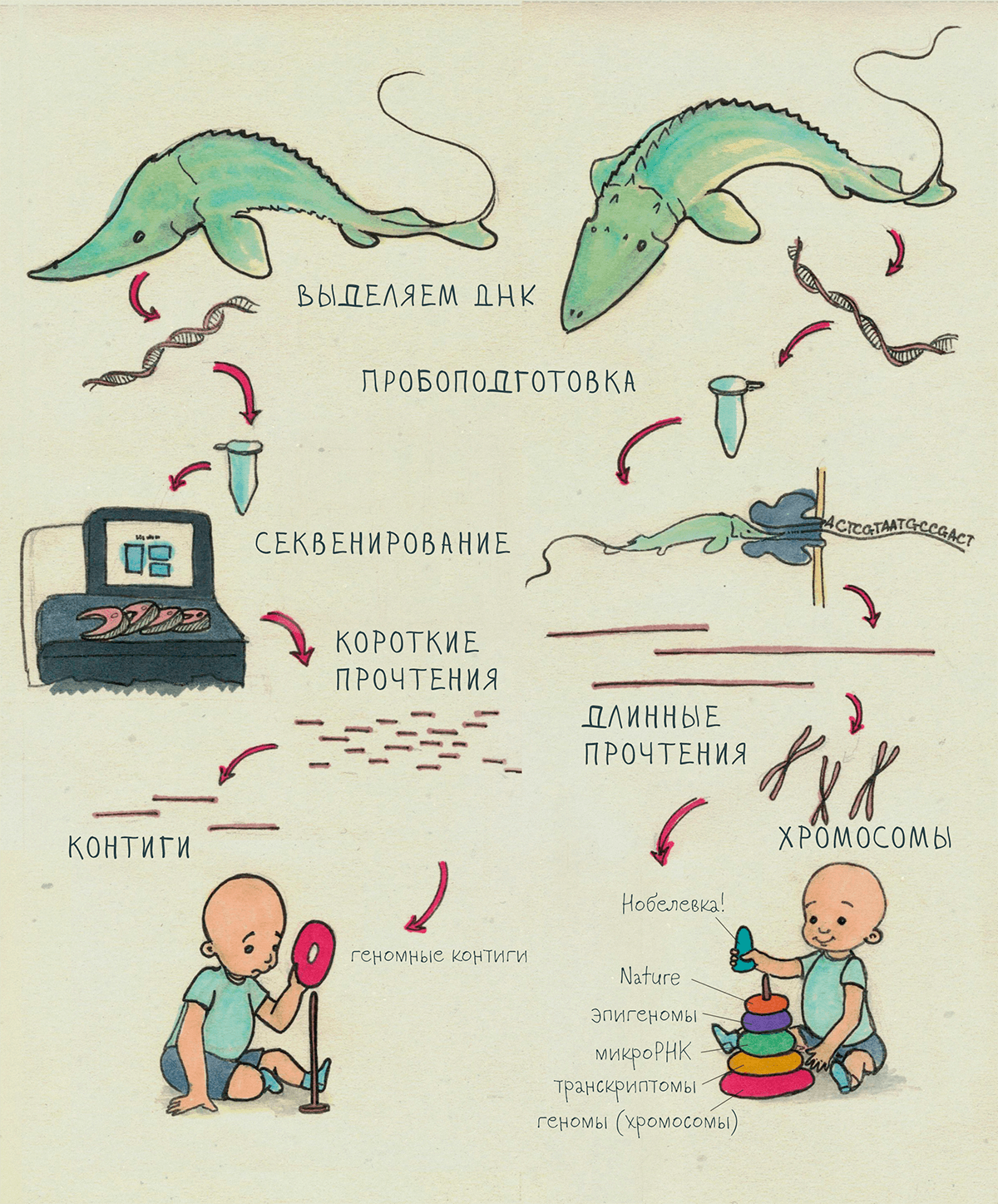
Рисунок 4. Метафорическое сравнение «обычного» NGS-секвенирования (слева) и нанопорового секвенирования «третьего поколения» (справа). Главное преимущество «нанопоры» — длина прочтений, дающая исследователю что-то более-менее похожее на «готовый» геном, нежели «мозаику» контигов. Это сулит возможность «сборки» геномов тех организмов, до которых раньше просто не удавалось добраться (например, полиплоидные осетровые, занесенные в списки охраняемых видов, СИТЕС).
иллюстрация Елены Беловой
Будущее нанопорового секвенирования
Сегодня нанопоровое секвенирование — одна из наиболее перспективных современных технологий определения последовательности нуклеиновых кислот и имеет ряд преимуществ перед традиционными секвенирующими платформами, прочно укоренившимися на биотехнологическом рынке. Упрощенная оценка эпигенетических особенностей генома, длинные прочтения и оценка сложно читаемых участков генома позволяют с оптимизмом смотреть на её дальнейшее развитие.
Имеющиеся недостатки (невысокое качество прочтения генетической информации и относительно невысокий объем данных, получаемых за один запуск прибора), по-видимому, в ближайшие годы будут решены, и тогда будущее действительно станет реальностью.
Взвесив все «за» и «против», оценив все плюсы и минусы нанопорового секвенирования, наша команда также решила влиться в число активных пользователей новой технологии, о результатах использования которой вы узнаете первыми со страниц научных журналов и, конечно же, любимой всеми нами «Биомолекулы».
Литература
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- J. J. Kasianowicz, E. Brandin, D. Branton, D. W. Deamer. (1996). Characterization of individual polynucleotide molecules using a membrane channel. Proceedings of the National Academy of Sciences. 93, 13770-13773;
- Stefan Howorka, Stephen Cheley, Hagan Bayley. (2001). Sequence-specific detection of individual DNA strands using engineered nanopores. Nat Biotechnol. 19, 636-639;
- D. Stoddart, A. J. Heron, E. Mikhailova, G. Maglia, H. Bayley. (2009). Single-nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore. Proceedings of the National Academy of Sciences. 106, 7702-7707;
- Код жизни: прочесть не значит понять;
- T. Laver, J. Harrison, P.A. O’Neill, K. Moore, A. Farbos, et. al.. (2015). Assessing the performance of the Oxford Nanopore Technologies MinION. Biomolecular Detection and Quantification. 3, 1-8;
- Miten Jain, Sergey Koren, Karen H Miga, Josh Quick, Arthur C Rand, et. al.. (2018). Nanopore sequencing and assembly of a human genome with ultra-long reads. Nat Biotechnol. 36, 338-345;
- Michael C Schatz. (2017). Nanopore sequencing meets epigenetics. Nat Methods. 14, 347-348;
- 12 методов в картинках: полимеразная цепная реакция;
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Nicholas J Loman, Joshua Quick, Jared T Simpson. (2015). A complete bacterial genome assembled de novo using only nanopore sequencing data. Nat Methods. 12, 733-735;
- Ryan R. Wick, Louise M. Judd, Claire L. Gorrie, Kathryn E. Holt. (2017). Completing bacterial genome assemblies with multiplex MinION sequencing. Microbial Genomics. 3;
- Alexander L. Greninger, Samia N. Naccache, Scot Federman, Guixia Yu, Placide Mbala, et. al.. (2015). Rapid metagenomic identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis. Genome Med. 7;
- Satomi Mitsuhashi, Kirill Kryukov, So Nakagawa, Junko S. Takeuchi, Yoshiki Shiraishi, et. al.. (2017). A portable system for rapid bacterial composition analysis using a nanopore-based sequencer and laptop computer. Sci Rep. 7;
- J. Goordial, Ianina Altshuler, Katherine Hindson, Kelly Chan-Yam, Evangelos Marcolefas, Lyle G. Whyte. (2017). In Situ Field Sequencing and Life Detection in Remote (79°26′N) Canadian High Arctic Permafrost Ice Wedge Microbial Communities. Front. Microbiol.. 8;
- Sarah L. Castro-Wallace, Charles Y. Chiu, Kristen K. John, Sarah E. Stahl, Kathleen H. Rubins, et. al.. (2017). Nanopore DNA Sequencing and Genome Assembly on the International Space Station. Sci Rep. 7;
- Ashley Byrne, Anna E. Beaudin, Hugh E. Olsen, Miten Jain, Charles Cole, et. al.. (2017). Nanopore long-read RNAseq reveals widespread transcriptional variation among the surface receptors of individual B cells. Nat Comms. 8, 16027;
- Hans J. Jansen, Michael Liem, Susanne A. Jong-Raadsen, Sylvie Dufour, Finn-Arne Weltzien, et. al.. (2017). Rapid de novo assembly of the European eel genome from nanopore sequencing reads. Sci Rep. 7;
- Mun Hua Tan, Christopher M Austin, Michael P Hammer, Yin Peng Lee, Laurence J Croft, Han Ming Gan. (2018). Finding Nemo: hybrid assembly with Oxford Nanopore and Illumina reads greatly improves the clownfish (Amphiprion ocellaris) genome assembly. GigaScience. 7;
- John R. Tyson, Nigel J. O'Neil, Miten Jain, Hugh E. Olsen, Philip Hieter, Terrance P. Snutch. (2018). MinION-based long-read sequencing and assembly extends theCaenorhabditis elegansreference genome. Genome Res.. 28, 266-274;
- Shan Wei, Zachary R Weiss, Pallavi Gaur, Eric Forman, Zev Williams Rapid preimplantation genetic screening (PGS) using a handheld, nanopore-based, DNA sequencer — Cold Spring Harbor Laboratory;
- Senne Cornelis, Yannick Gansemans, Lieselot Deleye, Dieter Deforce, Filip Van Nieuwerburgh. (2017). Forensic SNP Genotyping using Nanopore MinION Sequencing. Sci. Rep.. 7, 41759;
- Arthur C Rand, Miten Jain, Jordan M Eizenga, Audrey Musselman-Brown, Hugh E Olsen, et. al.. (2017). Mapping DNA methylation with high-throughput nanopore sequencing. Nat Methods. 14, 411-413;
- Эпигеном: параллельная реальность внутри клетки;
- Эпигенетика: невидимый командир генома.







