Половая жизнь хроматина
03 апреля 2017
Половая жизнь хроматина
- 1734
- 0
- 10
Отцовское и материнское ядра зиготы, стилизованные под планеты Марс и Венера. Автор рисунка Tibor Kulcsar
-
Авторы
-
Редакторы
Чем отличается хроматин в мужских и женских гаметах? Как он перестраивается при образовании зиготы? Ответы на эти вопросы не только интересны фундаментальной науке, но и могут помочь в перепрограммировании клеток. Интернациональная команда ученых разработала новый метод анализа структуры хроматина в индивидуальных клетках и применила его к ядрам ооцитов и зиготы. В эту работу значительный вклад внесли ученые из России: выпускники МГУ Илья Флямер и Сергей Ульянов, выпускник МФТИ Максим Имакаев. И нашими же соотечественниками возглавляются две из трех лабораторий, в которых выполнялась работа: это лаборатория Сергея Разина в московском Институте биологии гена и лаборатория Леонида Мирного в знаменитом MIT. Полученные результаты были опубликованы в журнале Nature.
Как устроен хроматин
Наша ДНК хранится в ядрах клеток в составе хроматина, формирующего 46 хромосом. Если развернуть все хромосомы из одной клетки до состояния известной всем двойной спирали ДНК и выложить их в линию друг за другом, то общая длина составит примерно два метра! У других эукариот количество хромосом и суммарная длина нитей ДНК могут отличаться, но всё равно это очень длинные молекулы. И чтобы вся эта ДНК уместилась в ядре, средний диаметр которого в наших клетках составляет
Сначала двойная спираль наматывается на специальные белковые катушки, образуя нуклеосомы [1]. Про следующие уровни укладки понятно гораздо меньше. Раньше считалось, что нуклеосомы объединяются в фибриллу толщиной 30 нм, но в последнее время появляется всё больше данных, опровергающих эту модель [2]. Так или иначе участки хроматина объединяются в кластеры — топологически ассоциированные домены (ТАДы). Одни ТАДы содержат ненужные данной клетке гены, а другие собраны из активного хроматина — там идет транскрипция. Если в ядре есть такое четкое разделение на активные и неактивные ТАДы, то говорят, что хроматин поделен на два компартмента: активный и неактивный.
Внутри ТАДов или на их границах могут находиться хроматиновые петли — важный инструмент регуляции активности генов: внутри петель происходит сближение регуляторных элементов с генами, а те регуляторные последовательности, что не попали в петлю, не могут влиять на гены внутри нее. «Биомолекула» уже сообщала, чем чреваты сдвиги границ петель, и как вообще устроен хроматин [3], [4], [5], [6].
Международная коалиция ученых
Хотя наука еще очень далека от четкого понимания устройства хроматина, эта область сейчас активно развивается, и публикуется множество интересных исследований. Не остаются в стороне и российские биологи: совместная работа трех лабораторий в России, Австрии и США внесла серьезный вклад в постижение тайн хроматина. «Мокрые» эксперименты выполнял в основном Илья Флямер в Институте молекулярных биотехнологий в Вене, а наибольший вклад в компьютерную обработку результатов и моделирование внес Максим Имакаев. В результате они разделили первое авторство в статье, опубликованной в журнале Nature: Flyamer I.M., Gassler J., Imakaev M. et al. (2017). Single-nucleus Hi-C reveals unique chromatin reorganization at oocyte-to-zygote transition [7].
Новый метод: как заглянуть в ядро отдельной клетки
Чтобы разобраться в сложных переплетениях ДНК в ядре, ученые строят карты хроматиновых контактов — устанавливают, какие участки ДНК взаимодействуют друг с другом в ядре. Для этого используют методы фиксации конформации хромосом. Начальные этапы всех этих методов одинаковы:
- Клетки обрабатывают формальдегидом, который за счет высокой реакционной способности альдегидной группы «сшивает» между собой крупные молекулы, в том числе ДНК и белки хроматина (то же самое происходит после употребления алкоголя: в организме повышается концентрация ацетальдегида, он реагирует с высокомолекулярными соединениями, и образуются их крупные агрегаты). Теперь участки хромосом, сближенные в ядре, намертво сшиты друг с другом. Вот она, та самая стадия фиксации хромосомной конформации.
- Клетки разрушают, при этом клеточные ядра остаются целыми и невредимыми, и все последующие стадии протекают внутри них [8].
- ДНК разрезают на множество маленьких фрагментов с помощью ферментов эндонуклеаз рестрикции.
- Фрагменты ДНК сшивают друг с другом с помощью фермента ДНК-лигазы. На этом этапе склеиваются друг с другом в одну длинную молекулу те участки ДНК, которые были сближены в пространстве.
Полученные фрагменты ДНК теперь можно анализировать. В зависимости от того, на какой вопрос ученые хотят ответить, они выбирают ту или иную разновидность метода.
- Если нужно узнать, взаимодействуют ли в ядре фрагмент А и фрагмент В, то можно провести ПЦР с праймерами к этим фрагментам. Есть продукт — значит, взаимодействуют. Этот самый простой вариант метода называется 3С.
- Но можно ставить и более сложные вопросы: «с какими фрагментами ДНК взаимодействует фрагмент А?» или «с какими фрагментами ДНК взаимодействует каждый из фрагментов?» Для ответа на последний вопрос применяют метод Hi-C, который заключается в тотальном секвенировании получившихся продуктов.
О ПЦР и секвенировании ДНК можно прочитать в статье «Важнейшие методы молекулярной биологии и генной инженерии» [9].
Второй путь достаточно дорог, но зато можно сразу получить карту всех хроматиновых контактов. Обычно для Hi-C использовали клеточную культуру, но вот беда: оказалось, что даже две соседние клетки печени различаются по устройству хроматина. Как говорит в своих лекциях один из авторов обсуждаемой статьи, Сергей Владимирович Разин, работать с популяцией клеток — всё равно что измерять среднюю температуру по больнице. Поэтому ученые придумывают подходы к работе с индивидуальными клетками и уже научились секвенировать индивидуальные клеточные геномы [10]. Как раз такой метод — snHi-C (single-nucleus Hi-C, то есть Hi-C на индивидуальных ядрах) — и был разработан авторами статьи, опубликованной в Nature. Этим методом (рис. 1) они обнаруживают в 10 раз больше хроматиновых контактов в ядрах, чем другие научные группы [11].
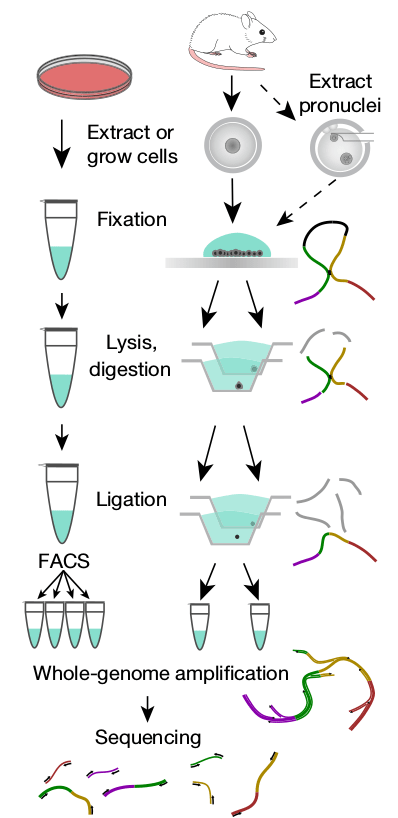
Рисунок 1. Последовательность действий snHi-C для клеточной культуры и ооцитов/зигот: фиксация, лизис и рестрикция, лигирование, полногеномная амплификация и секвенирование
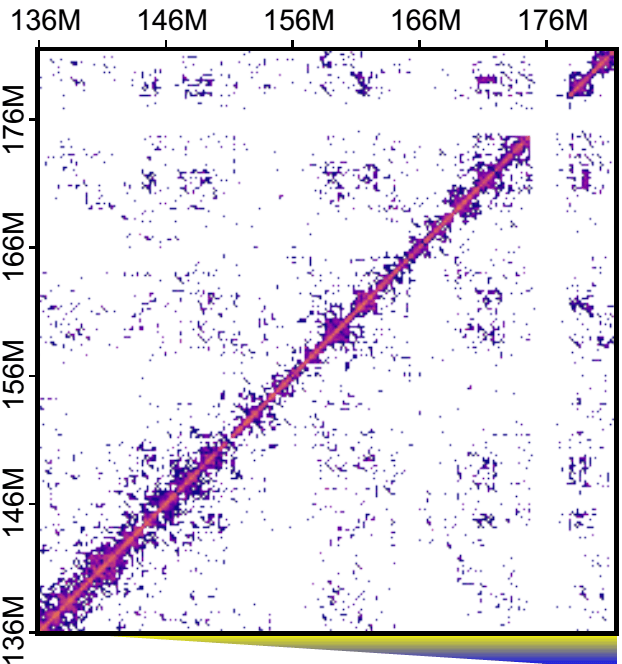
Рисунок 2. Карта контактов участка хромосомы в индивидуальном ооците. Точки — зарегистрированные контакты между участками, отложенными по осям. Диагональ — взаимодействия участков с самими собой
Каждая клетка уникальна
Свой новый метод ученые в первую очередь применили к ядрам ооцитов мышей (рис. 2). Их интересовал вопрос, действительно ли в отдельных ядрах существуют все эти хроматиновые структуры: петли, ТАДы, хроматиновые компартменты? В данной работе решили сконцентрироваться на ТАДах, поскольку их заметить проще всего. Были предложены три альтернативные гипотезы:
- Все ТАДы, известные из экспериментов с популяцией клеток, присутствуют в каждой отдельно взятой клетке в точно таком же виде. Тогда работа с клеточными популяциями — «измерение средней температуры по больнице» — вполне оправдана.
- Все ТАДы можно увидеть только на популяционных данных, а в каждой отдельной клетке некоторые ТАДы могут отсутствовать или сливаться с соседними.
- В каждой отдельной клетке существуют некоторые хроматиновые кластеры, но их границы не соответствуют популяционным ТАДам.
Подтвердилась последняя гипотеза: кластеры контактов в отдельных клетках часто нарушают границы ТАДов. Авторы предполагают, что это отражает процесс создания хроматиновых доменов: такие домены образуются в любом достаточно длинном полимере случайным образом. В клетке эти кластеры формируются точно так же, но есть некоторые чуть более частые положения их границ. Они-то и соответствуют границам популяционных ТАДов. Так в полный хаос живая система вносит немного порядка.
Отработав свой метод на более-менее простых моделях, авторы перешли к сáмой интересной проблеме: чем отличается хроматин в материнском и отцовском ядрах зиготы млекопитающих? Ученые отбирали зиготы мышей на ранних стадиях после оплодотворения, когда генетическая информация от отца и матери еще не перемешалась и находится в отдельных ядрах, и с помощью snHi-C анализировали хроматин этих ядер. С петлями и ТАДами всё было в порядке: они выявлялись в обоих ядрах. А вот результат анализа активных и неактивных компартментов получился неожиданным: в отцовском ядре они есть, хоть и слабо выражены, а в материнском совсем не обнаруживаются! И это первый известный пример ядер млекопитающих, хроматин которых не разделен на компартменты.
Компьютерное моделирование подтверждает предположение авторов
В целом по структуре хроматина материнское и отцовское ядра зиготы заметно отличаются как друг от друга, так и от соматических клеток. Авторы полагают, что на состояние хроматина может влиять предшествующая судьба клетки, а она разная у ооцита и сперматозоида. Это предположение проверили в лаборатории Леонида Мирного с помощью метода молекулярной динамики. Для этого ученые разработали специальный алгоритм, потом ввели данные начального состояния хроматина, и компьютер предсказал, что вероятнее всего будет происходить с хроматином. Компьютерное моделирование подтверждает догадку авторов статьи: если моделировать деконденсацию хроматина из конденсированных хромосом, как в ооците, то получается структура, похожая на хроматин в материнском ядре зиготы. Если же моделировать деконденсацию компактных хроматиновых глобул, как в сперматозоиде, то получается нечто похожее на хроматин в отцовском ядре (рис. 3).
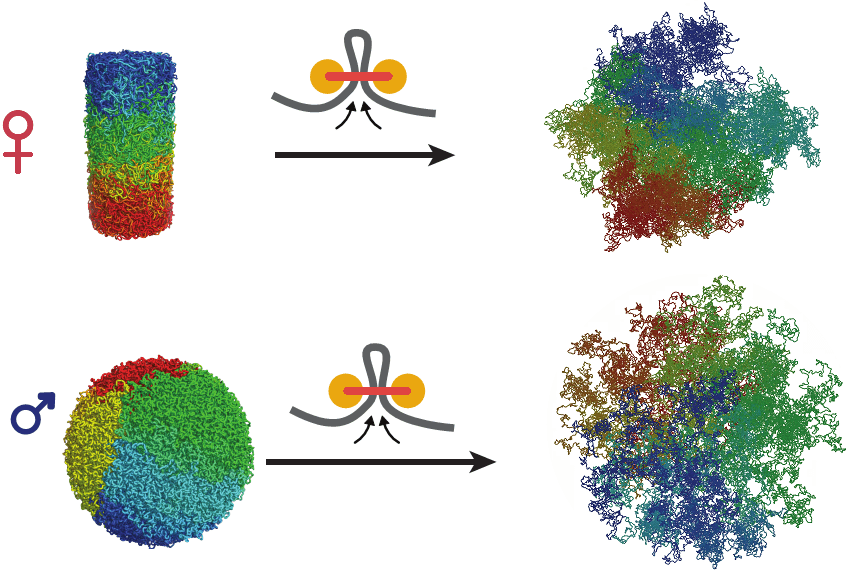
Рисунок 3. Результаты моделирования деконденсации хроматина в ядрах женских (сверху) и мужских (снизу) гамет. Чтобы увидеть рисунок в полном размере, нажмите на него
Итоги и перспективы
Таким образом, в работе были показаны две важные вещи. Во-первых, с помощью single-cell метода ученые убедились, что различные структуры, ранее обнаруженные в хроматине, существуют как популяционные средние, а каждая отдельная клетка уникальна по укладке генома в ядре. Так что вместо измерения средней температуры можно и нужно работать с каждым пациентом! Во-вторых, впервые были получены данные об устройстве хроматина в ядрах ооцитов и зиготы. Именно на этом этапе происходит главное хроматиновое чудо в нашей жизни: хроматин переходит в некоторое базовое состояние, и клетка теряет специализацию — становится тотипотентной, то есть способной к образованию любых органов и тканей нового организма. Если мы разберемся в подробностях этих перестроек, то сможем научиться более эффективно перепрограммировать клетки и создавать индуцированные плюрипотентные стволовые клетки более осознанно (ИПСК «биомолекула» уже не раз уделяла внимание [12], [13], [14]). А там уж недалеко и до органов из пробирок!
***
О том, как выполнялась работа и как обстоят дела в науке о хроматине, «биомолекуле» рассказал один из первых авторов статьи, Илья Флямер (рис. 4).
— Работа велась сразу в трех странах: России, Австрии и США. Как сформировалась такая команда?
— Началось всё с моего интереса к single-cell методам. Учась на 4 курсе биофака МГУ, я познакомился с Максимом Имакаевым из лаборатории Леонида Мирного, находящейся в легендарном MIT. Максим приезжал на школу «Современная биология и биотехнологии будущего» [15]. Там мы с ним много беседовали на эту тему и договорились, что он будет обсчитывать результаты, когда они появятся. После курсовой работы в лаборатории Сергея Владимировича Разина я поехал на летнюю школу в Вену, в лабораторию Кикуе Тачибана-Конвальски (Kikuё Tachibana-Konwalski). Там мои идеи понравились, и я в итоге остался в этой группе (рис. 4) делать диплом. Мы разработали snHi-C и получили приличные данные, но для статьи их еще было недостаточно, а мне уже пора было уезжать на PhD в Эдинбург, где я сейчас и работаю. Я показал протокол PhD студентке Йоханне (Johanna Gassler), и она доделывала значительную часть экспериментов. В итоге мы около года назад подали статью в Nature.
— Раз год назад отправили, а статья опубликована только что, значит, были какие-то проблемы?
— Я бы так не сказал. Это на самом деле еще не очень долго для такого журнала, бывает и гораздо дольше! Рецензии в целом были положительные (или как минимум конструктивные), но попросили еще кое-что доделать. Например, подтвердить все основные выводы методом FISH (это когда мы с помощью флуоресценции напрямую видим в ядре отдельные участки хромосом). Но мы этого ожидали и еще до получения рецензий планировали эксперименты, а потому со всем справились за несколько месяцев. По совпадению, лаборатория, в которой я сейчас работаю, специализируется на этом методе, что нам очень помогло. Йоханне пришлось попросить у другой лаборатории протокол для FISH на зиготах, а также приехать на неделю к нам в Эдинбург, чтобы освоить метод.
— Совсем недавно в том же Nature были опубликованы две статьи про структуру хроматина, в том числе и про single-cell методы [16], [17]. Это случайно так вышло?
— Нет, сейчас это очень модная тема, и многие пытаются опубликоваться в самых лучших журналах, поэтому вдвойне приятно, что нам это удалось. У нас действительно хорошая работа получилась, а метод snHi-C, надеюсь, позволит работать с хроматином редких клеточных типов: стволовых клеток, отдельных раковых клеток и клеток развивающихся эмбрионов. Так что он теперь будет востребован в разных исследованиях.
— Тогда будем ждать новых открытий в этой области совсем скоро!
Литература
- Это FACT;
- Sergey V Razin, Alexey A Gavrilov. (2014). Chromatin without the 30-nm fiber. Epigenetics. 9, 653-657;
- Скрытая угроза: описан новый механизм активации протоонкогенов;
- Загадочное путешествие некодирующей РНК Xist по X-хромосоме;
- Катится, катится к ДНК гистон;
- Транскрипция в хроматине: как проходить сквозь стены;
- Ilya M. Flyamer, Johanna Gassler, Maxim Imakaev, Hugo B. Brandão, Sergey V. Ulianov, Nezar Abdennur, et al.. (2017). Single-nucleus Hi-C reveals unique chromatin reorganization at oocyte-to-zygote transition. Nature. undefined, undefined;
- A. A. Gavrilov, E. S. Gushchanskaya, O. Strelkova, O. Zhironkina, I. I. Kireev, O. V. Iarovaia, et al.. (2013). Disclosure of a structural milieu for the proximity ligation reveals the elusive nature of an active chromatin hub. Nucleic Acids Research. 41, 3563-3575;
- Важнейшие методы молекулярной биологии и генной инженерии;
- Секвенирование единичных клеток (версия — Metazoa);
- Takashi Nagano, Yaniv Lubling, Tim J. Stevens, Stefan Schoenfelder, Eitan Yaffe, Wendy Dean, et al.. (2013). Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64;
- Была клетка простая, стала стволовая;
- Снежный ком проблем с плюрипотентностью;
- Предохранитель ИПСК;
- Зимняя школа «Современная биология и Биотехнологии будущего»: передружить всех между собой!;
- Tim J. Stevens, David Lando, Srinjan Basu, Liam P. Atkinson, Yang Cao, Steven F. Lee, et al.. (2017). 3D structures of individual mammalian genomes studied by single-cell Hi-C. Nature. undefined, undefined;
- Robert A. Beagrie, Antonio Scialdone, Markus Schueler, Dorothee C. A. Kraemer, Mita Chotalia, Sheila Q. Xie, et al.. (2017). Complex multi-enhancer contacts captured by genome architecture mapping. Nature. 543, 519-524.