
Сверим часы
20 января 2008
Сверим часы
- 3060
- 2
- 6
Молекулярные часы — это пока не самый точный прибор, однако со временем он будет неплохо откалиброван
эта и другие иллюстрации к обзору выполнены компанией Visual Science
-
Автор
-
Редакторы
Как научиться определять время, сравнивая молекулы? В настоящее время развитие молекулярной биологии, биоинформатики и геномики позволяет находить новые подходы к изучению центрального вопроса всей биологической науки — проблемы эволюции живых систем. Одним из весомых вкладов этих относительно молодых дисциплин в развитие данной области является метод оценки времени эволюционного расхождения таксонов — так называемый метод «молекулярных часов».
Развитие молекулярной систематики
Идея использовать биомолекулы для определения степени родства между видами, как и многие другие важные идеи в биохимии прошлого века, пришла в голову Лайнусу Полингу (Linus Pauling [1]). Предложенная им и его коллегой Эмилем Цукеркандлем (Emil Zuckerkandl) в 1965 году концепция была достаточно проста и основывалась примерно на тех же принципах, на которых основана систематика морфологическая. Ученые рассудили, что чем больше сходство между биомолекулами, синтезируемыми организмами, тем более филогенетически близки сами организмы, и наоборот. В первых экспериментах, посвященных изучению данного вопроса, Полинг и его коллеги исследовали некоторые биохимические характеристики (такие, например, как молекулярная масса и электрофоретическая подвижность) гемоглобина, выделенного из крови представителей разных таксонов. В результате оказалось, что гемоглобины человека и гориллы отличаются заметно меньше, чем они вместе отличаются от гемоглобинов лошади. Еще дальше от этой группы стояли гемоглобины курицы, ну а самые сильные отличия наблюдались в белках, выделенных из крови рыбы [2]. Несложно заметить, что выводы зарождающейся молекулярной систематики в этом случае полностью совпали с устоявшимися представлениями морфологов. Разумеется, подобный результат вполне удовлетворил исследователей.
Собственно, время расхождения таксонов при таком подходе определяется исходя из двух параметров: примерной скорости накопления изменений в неких биомолекулах и непосредственного количества этих изменений (различий между биомолекулами таксонов, время расхождения которых пытается определить исследователь). Чем раньше виды разошлись, тем больше отличий в последовательностях биополимеров они накопили. Зная количество различий и скорость их появления можно рассчитать время, за которое они образовались. Однако это только теория, а на практике оба этих показателя довольно трудно поддаются точной оценке.
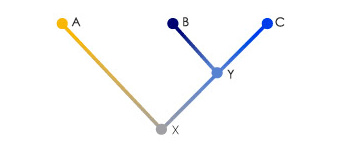
Рисунок 1. Филогенетическое дерево трёх гипотетических видов A, B и C. Эти виды имеют общего предка X. Виды B и C имеют более позднего предка Y.
Одна из первых попыток осуществить калибровку молекулярных часов была предпринята в конце 60-х годов работах Винсента Сэрича (Vincent Sarich) и Алана Уилсона (Alan Wilson), изучавших реципрокное сродство иммуноглобулинов разных родов и видов приматов [3]. Как и Полинг, эти исследователи работали с белками. Сначала выделялись белки из трех разных таксонов. Для простоты назовем их A, B и C (рис. 1). Причем известно, что B и C эволюционно ближе друг к другу, чем к A. К каждому белку были получены антитела, после чего проверялось сродство этих антител к «чужим» белкам. Сначала антитела к белку А тестировались на сродство к белкам В и С, после чего антитела к белкам В и С тестировались на сродство к более эволюционно удаленному белку А. Их целью было выяснить правильность гипотезы о том, что скорость накопления изменений в белковых молекулах является постоянной для изучаемых видов. Данные исследования эту гипотезу не опровергли, поскольку показали, что скорость накопления изменений в линии “В” оказалась такой же, как и скорость накопления изменений в линии “С” с момента их расхождения. После этого ученые предположили, что, зная время расхождения линий “В” и “С” из палеонтологических данных, можно откалибровать получившиеся молекулярные часы и впоследствии датировать время расхождения таксонов, изучая только их биомолекулы. Впрочем, очень быстро выяснилось, что это не так просто, поскольку их попытки осуществить подобные операции не увенчались успехом — молекулярные оценки сильно расходились с данными палеонтологии.
Однако, несмотря ни на что, методика молекулярных часов находила широкое применение на ранних этапах молекулярно-филогенетических исследований. В частности, ее использовали для оценки времени расхождения крупных таксонов. Например, Дикерсон [4] изучал эволюционные изменения гемоглобина и определил, что растения, животные и грибы дивергировали порядка 1—1,2 млрд. лет назад. Используя последовательности тРНК и 5S РНК МакЛафлин и Дэйхофф [5], а также Кимура и Ота [6] оценили время расхождения про- и эукариот, получив результат порядка 2—2,6 млрд. лет назад. Как уже говорилось, все эти оценки характеризуются большими погрешностями, однако они не расходятся принципиально с оценками более точных современных исследований.
Несколько отличный подход к определению степени родства видов молекулярными методами применили Бриттен и Кон в 1968 году [7]. Суть их метода заключалась в сравнении сразу всей ДНК исследуемых видов. Это делалось таким образом: сначала молекулы геномной ДНК подвергались денатурации, после чего одноцепочечные молекулы отжигались друг с другом. Далее исследовался полученный гетеродуплекс. Логика была такова: чем больше энергии придется затратить для того, чтобы осуществить денатурацию полученного дуплекса (чем дуплекс более прочен), тем ближе виды между собой, поскольку очевидно, что стабильность такой гибридной ДНК напрямую зависит от того, насколько похожи две частично комплементарные цепи.
Результаты этих исследований не были очень впечатляющими, поскольку тогда еще не было четкого понимания того, что геном состоит далеко не только из уникальной ДНК. Картину портили всевозможные геномные повторы, количество и размер которых, как оказалось, плохо коррелирует со степенью различия между видами (кстати, данная работа сделала серьезный вклад в их изучение) [8]. Возникновение и исчезновение повторов — это непредсказуемый процесс, который может быть вызван огромным числом самых разных причин. К тому же, предполагать, что число повторов изменяется с постоянной скоростью у всех видов в любой исторический момент, довольно наивно.
К 70-м годам прошлого века стало ясно, что если имевшиеся тогда методы молекулярной систематики и подходят для того, чтобы классифицировать эволюционно далекие объекты, то когда речь заходит о близких видах, сопоставление белков и полных геномов обнаруживает явные неточности и дает ненадежные результаты. Например, оказалось, что биохимические различия между шимпанзе и человеком настолько малы, что, судя только по ним, невозможно будет отделить один вид от другого. Становилось понятно, что для более точных результатов надо искать и сравнивать конкретные последовательности ДНК
ДНК: какие куски лучше сравнивать?
Как уже говорилось, одним из самых тонких мест в методике молекулярных часов является определение скорости накопления изменений, которую довольно проблематично точно оценить. К тому же, в большинстве случаев эта скорость для удобства считается постоянной на всем времени с момента расхождения изучаемых видов, а говорить об этом не всегда правомерно. Классическим эволюционистам идея постоянства скорости накопления мутаций была непонятна совершенно. Ведь согласно общепризнанной в то время синтетической теории эволюции, скорость эволюционного изменения видов определяется факторами среды и интенсивностью естественного отбора, а, следовательно, она просто обязана колебаться, поскольку условия среды меняются с переменной скоростью.
Первые идеи по разрешению сложившегося противоречия были предложены японским биологом Моту Кимура (Motoo Kimura), который сформулировал так называемую «нейтральную теорию» молекулярной эволюции [9]. Предположение заключалось в том, что большинство изменений в последовательности геномной ДНК никак не отражается на фенотипе особей, а, следовательно, не попадает под действие естественного отбора. Интересно, что по этой причине, концепцию Кимуры некоторое время считали противоречащей классическому дарвинизму. Однако сейчас очевидно, что никакого противоречия нет.
Дело в том, что отбор преимущественно действует на уровне строения белковых молекул. Ведь для того, чтобы организм нормально существовал, его белки должны правильно работать, а это невозможно в случае, если белковые молекулы накопят слишком много структурных изменений. Следовательно, организмы, синтезирующие дефектные белки, погибают, а выживают только те, чьи белковые молекулы нормально функционируют и не содержат критического количества перестроек. Но, как оказывается, отсутствие различий в белковых молекулах видов вовсе не говорит о том, что этих различий нет в последовательностях ДНК.
Генетический аппарат клетки устроен таким образом, что непосредственно на структуру белков (а, следовательно, на фенотипические проявления признаков, подвергающиеся действию отбора) влияют далеко не все последовательности ДНК. Сейчас хорошо известно, что в среднем у эукариотических организмов количество структурных генов может колебаться от 10% до 40% от всего генома. Остальные последовательности представлены межгенными спейсерами, регуляторными участками, мобильными элементами и гетерохроматиновыми повторами, мутации в которых далеко не всегда отражаются на фенотипе особи и в массе своей оказываются именно нейтральными.
Разумеется, эти представления появились не сразу. В 30-е годы XX века считалось, что в хромосомах нет ничего, кроме генов, линейно соединенных между собой наподобие бусин на нити. Эти соображения были особенно популярны в период активного изучения политенных хромосом дрозофилы, которым присуща характерная поперечная исчерченность. Поперечные полоски на таких хромосомах, как раз и считали некоторое время генами, наблюдаемыми непосредственно в световой микроскоп. К 60-м годам становится ясно, что между генами содержится множество нуклеотидных последовательностей, которые тогда считались «мусорными», ну а к середине 70-х годов стало ясно, что и сама тонкая структура гена такова, что не все его участки непосредственно влияют на фенотипическое проявление признака (рис. 2). Ведь гены содержат экзоны (кодирующие участки) и интроны (участки, удаляющиеся в ходе процессинга пре-мРНК). Вполне естественно, что скорости изменения функционально разных последовательностей должны отличаться, поскольку важные для нормального существования организма регионы генома естественным образом окажутся более консервативными, а менее значимые участки будут накапливать изменения интенсивнее.
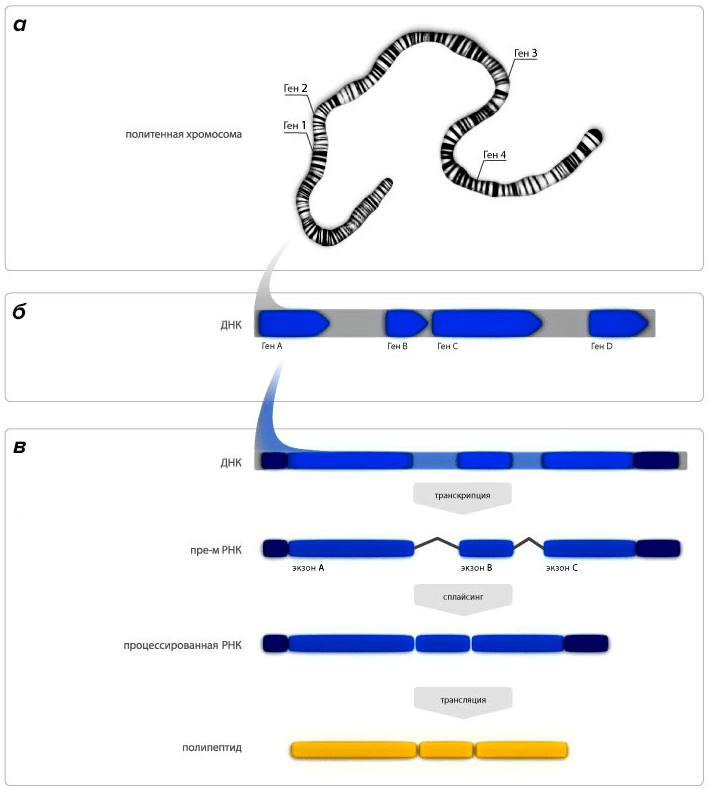
Рисунок 2. Эволюция представлений о строении гена. а — 30-е годы. Хромосомы — это цепочки из генов. б — 60-е годы. Гены в хромосомах разделены спейсерами и гетерохроматиновыми участками. в — Наши дни. Гены содержат интроны, которые удаляются из пре-мРНК в результате сплайсинга.
Молекулярные часы: множество стрелок и все идут с разной скоростью!
В 80-е годы c накоплением данных о ДНК-последовательностях геномов разных организмов число противоречий относительно методики молекулярных часов возросло. В 1986 году в своей работе Бриттен заключил, что разные группы живых организмов могут накапливать молекулярные изменения с разной скоростью [10]. В частности, он пришел к выводу, что грызуны, а именно мыши и крысы, эволюционировали заметно быстрее, чем прочие группы млекопитающих, а, например, человекообразные обезьяны, наоборот, характеризовались низкой скоростью молекулярной эволюции (нейтральные замены накапливались медленнее). Сам Бриттен склонен был объяснять это тем, что у данных групп могли быть различия в системе репарации ДНК, что и привело к расхождению в скорости накопления нейтральных мутаций. Однако эти исследования показывали, что скорость нуклеотидных замен пропорциональна скорее количеству поколений, сменившихся с момента дивергенции исследуемых таксонов, чем абсолютному времени, прошедшему с тех пор, что усложняло применение метода молекулярных часов в исследованиях, поскольку вносило дополнительные неточности.
Примерно в это же время ученые с короткими фамилиями Ву и Ли сделали предположение о том, что в линиях грызунов и человекообразных обезьян могут по каким-то причинам различаться количества синонимичных (не приводящих к замене аминокислоты и, следовательно, не отражающихся на фенотипе) и несинонимичных нуклеотидных замен [11]. Им даже удалось подтвердить это в своей работе, в которой они показали, что число синонимичных замен у грызунов в два раза больше, чем в линии наших предков. Эти данные подтверждали необходимость сравнивать не абсолютные промежутки времени, а количество поколений, прошедших с момента дивергенции.
Однако эти заключения были подвергнуты сомнению в работе Эстила [12], в которой утверждалось, что причиной расхождений в скоростях накопления мутаций была неверная оценка времени дивергенции общих предков линии грызунов и человекоподобных обезьян. Впрочем, в последующих работах Ли было показано, что есть разница в скорости накопления замен в интронах некоторых генов у обезьян Старого света и их человекоподобных родственников [13].
Сравнение скоростей молекулярной эволюции различных локусов проводилось и на других объектах. В частности, было показано, что ген алкогольдегидрогеназы у гавайских представителей рода Drosophila изменялся заметно быстрее в сравнении с таким же геном у D. pseudoobscura [14]. Или, например, известно, что структурные гены митохондрий по каким-то причинам изменяются заметно медленнее у рыб, чем у млекопитающих [15]. А недавно с помощью молекулярно-филогенитического анализа удалось не только оценить время, прошедшее с момента дивергенции опсинов, отвечающих за зрение в различных таксонах животного мира, но и доказать сам факт возникновения зрения [16], насолив тем самым противникам идеи биологической эволюции, считающим, что возникновение новых функций в процессе естественного отбора невозможно по причине нежизнеспособности переходных форм.
Все эти результаты свидетельствуют о том, что в разработке метода молекулярных часов есть еще множество неразрешенных вопросов. И по сей день не ясно, чем вызывается разница в скорости накопления мутации и каков вклад в этот процесс систем репарации ДНК и внешних воздействий со стороны среды обитания. Также не всегда понятно, какие именно участки ДНК лучше применять в тех или иных исследованиях. Ясно только то, что эта методика может стать очень удобной и действенной по мере накопления большего количества данных о составе последовательностей ДНК разных видов.
Литература
- На заре молекулярной графики;
- Emile Zuckerkandl, Linus Pauling. (1965). Molecules as documents of evolutionary history. Journal of Theoretical Biology. 8, 357-366;
- Sarich V.M. and Wilson A.C. (1967). Rates of albumin evolution in primates. Proc. Natl. Acad. Sci. U.S.A. 58, 142–148;
- Richard E. Dickerson. (1971). The structure of cytochromec and the rates of molecular evolution. J Mol Evol. 1, 26-45;
- P. J. McLaughln, M. O. Dayhoff. (1970). ukaryotes versus Prokaryotes: An Estimate of Evolutionary Distance. Science. 168, 1469-1471;
- MOTOO KIMURA, TOMOKO OHTA. (1973). Eukaryotes-Prokaryotes Divergence estimated by 5S Ribosomal RNA Sequences. Nature New Biology. 243, 199-200;
- R. J. Britten, D. E. Kohne. (1968). Repeated Sequences in DNA. Science. 161, 529-540;
- Геном человека: полезная книга, или глянцевый журнал?;
- MOTOO KIMURA. (1968). Evolutionary Rate at the Molecular Level. Nature. 217, 624-626;
- R. Britten. (1986). Rates of DNA sequence evolution differ between taxonomic groups. Science. 231, 1393-1398;
- C. I. Wu, W. H. Li. (1985). Evidence for higher rates of nucleotide substitution in rodents than in man.. Proceedings of the National Academy of Sciences. 82, 1741-1745;
- Easteal S. (1985). Generation time and the rate of molecular evolution.. Molecular Biology and Evolution;
- Li W.H. and Wu C.I. (1987). Rates of nucleotide substitution are evidently higher in rodents than in man.. Molecular Biology and Evolution;
- Russo C.A., Takezaki N., Nei M. (1995). Molecular phylogeny and divergence times of drosophilid species.. Molecular Biology and Evolution;
- Andrew P. Martin, Gavin J. P. Naylor, Stephen R. Palumbi. (1992). Rates of mitochondrial DNA evolution in sharks are slow compared with mammals. Nature. 357, 153-155;
- Откуда пошло зрение.






