Выделяем нуклеиновые кислоты: эволюция методов
28 октября 2022
Выделяем нуклеиновые кислоты: эволюция методов
- 19062
- 1
- 27
Выделение нуклеиновых кислот — ключевой этап работы с геномом организмов, который позволил приблизиться к разгадке многих тайн эволюции, развить медицину и изучить индивидуальные особенности представителей разных форм жизни. Рисунок в полном размере.
рисунок Элины Стояновой
-
Автор
-
Редакторы
-
Иллюстратор
Пожалуй, самым распространенным «мокрым» методом в биологии является выделение нуклеиновых кислот. Именно от этого первого шага нередко зависит и результат многостадийного эксперимента — скажем, секвенирования важнейшего генома. Сколь разнообразны представители жизни на планете, столь же обширны и подходы к извлечению ДНК и РНК из клеток. Об этом — новая статья цикла «Ультрасовременные методы».
Ультрасовременные методы
Методы, которыми оперирует современная наука, постоянно совершенствуются. Некоторые области развиваются настолько стремительно, что порой даже специалистам сложно уследить за новейшими приборами и модификациями методик. Так происходит, например, в геномике. Другие же области (такие как гистология и связанные с ней световая и флуоресцентная микроскопия) развиваются куда медленнее, но всё равно расширяются и обзаводятся более высокопроизводительными методами. В статьях нового спецпроекта (он продолжает наш хит — «12 биологических методов в картинках») мы хотим рассказать, какие методы помогают ученым совершать новые открытия сегодня, и чего нам ждать в будущем.

Партнер этой статьи — «СкайДжин».
Пять причин выбрать SkyGen:
- Доступ к высококачественной продукции для молекулярной биологии.
- Высококвалифицированная научная поддержка.
- Быстрая логистика и складская программа.
- Удобное и взаимовыгодное сотрудничество.
- Адекватные цены.
Сотрудников лабораторий разных профилей объединяет необходимость работы с нуклеиновыми кислотами. Например, генетику из медицинской клиники потребуется извлечь ДНК из клеток крови, чтобы провести тестирование на некий синдром. В это же время на другом конце планеты лаборант в НИИ, включив погромче музыку в наушниках, старается как можно аккуратнее выделить ДНК из клеток консервированного мяса. Такую работу институту подкинули местные надзорные органы, заподозрившие производителя в недостаточном контроле качества продуктов.
Перенесемся в горы Перу, где в недавней экспедиции собрали пробы воды в озере Титикака. Теперь предстоит сделать посев и определить микробиом, который обитает в одном из самых высокогорных замкнутых водоемов мира. Это тоже требует выделения нуклеиновых кислот из образцов. А вот бедолаге из криминалистической лаборатории Франции повезло меньше: его объект, из которого требуется извлечь ДНК, — полупрозрачный обрезок ногтя предполагаемого преступника . Стоп-стоп-стоп, скажете вы, есть еще более трудные случаи — извлечение древней ДНК из окаменелостей . Здесь стандартным набором не отделаешься, придется выложиться на полную.
Подробнее о генетической экспертизе в криминалистике мы писали в статье «Криминалистика. Молекулярно-генетическая экспертиза» [1]. А о древней ДНК — в статье «Древняя ДНК: привет из прошлого» [2]. И об актуальном — в статье «Крестный отец палеогеномики — лауреат Нобелевской премии по физиологии/медицине (2022)» [3].
Какой бы ни была цель каждого научного сотрудника, первым этапом работы всегда окажется один из наиболее рутинных — выделение нуклеиновых кислот. Экстракция ДНК или РНК имеет решающее значение для успеха многих экспериментов. От чистоты и выхода нуклеиновой кислоты будет зависеть результат последующих этапов работы — например, ПЦР . А что уж говорить о секвенировании ! Оно требует ДНК и РНК отличного качества: чем лучше, тем выше вероятность получить достоверный результат.
Об этих важнейших методах мы подробно писали в статьях спецпроекта «12 биологических методов в картинках»: «Полимеразная цепная реакция» [4] и «Секвенирование нуклеиновых кислот» [5].
Начинали мы говорить об экстракции, в том числе и нуклеиновых кислот, в статье «12 методов в картинках: очистка молекул и разделение смесей» [6]. Однако прежде чем сейчас, уже в рамках спецпроекта «Ультрасовременные методы», нырнуть в суть технологии с головой, давайте немного вспомним историю.
История выделения нуклеиновых кислот
Важнейшим событием в истории изучения ДНК стало открытие ее структуры Уотсоном и Криком в 1953 году [7]. Однако на самом деле история отношений ученых и нуклеиновых кислот началась гораздо раньше — в 1869 году, когда молодой швейцарский врач Фридрих Мишер (рис. 1), только получив образование, занялся научными изысканиями в лаборатории биохимика Феликса Хоппе-Зейлера (Тюбинген, Германия). Целью исследований был поиск и изучение так называемых «строительных блоков жизни» — белков.

Рисунок 1. Иоганн Фридрих Мишер
В ходе опытов с клетками крови — лейкоцитами — Фридрих Мишер обратил внимание на вещество, которое коренным образом отличалось от белков по свойствам. Оно выпадало в осадок из раствора при добавлении кислоты и снова растворялось при добавлении щелочи. Обнаруженные молекулы, случайно извлеченные из ядер лейкоцитов, Мишер назвал «нуклеинами» [8].
Мишер выделил нуклеин из клеток гноя с хирургических повязок. Сперва он отделил лейкоциты от сыворотки при помощи разбавленного раствора сульфата натрия. Затем, оставив промывочный раствор на 1–2 часа, дождался осаждения клеток и изучил полученные лейкоциты под микроскопом на предмет разрушения.
Теперь нужно было отделить ядра от цитоплазмы, для чего Мишер разработал собственную методику: он промывал клетки 6–10 раз раствором разбавленной соляной кислоты в течение нескольких недель при низкой температуре. Затем Мишер убедился, что в растворе не осталось цитоплазмы, используя раствор йода, которым окрашивались клеточные компоненты. После ученый добавил к раствору с ядрами водный раствор эфира и долго встряхивал пробирку. Оставшиеся липиды растворились, часть ядер «сгруппировалась» в поверхностную пленку, а часть осталась в растворе. Отфильтровав раствор, Мишер исследовал ядра под микроскопом, после чего подверг их воздействию щелочного раствора и увидел, что они становятся полупрозрачными и набухают. Последующее добавление уксусной или соляной кислоты в избытке привело к получению хлопьевидного осадка — нуклеиновых кислот [9].
Вот так немецкий ученый впервые в истории выделил нуклеиновые кислоты. После Мишер усовершенствовал свою методику, но, даже глядя на ее первую версию, те, кто хоть раз выделял ДНК, заметят явное сходство и основные принципы, которые лягут в основу всех подходов к выделению, появившихся позднее (рис. 2). Позже ученик Мишера Ричард Альтман даст новому веществу привычное для нас название «нуклеиновая кислота».
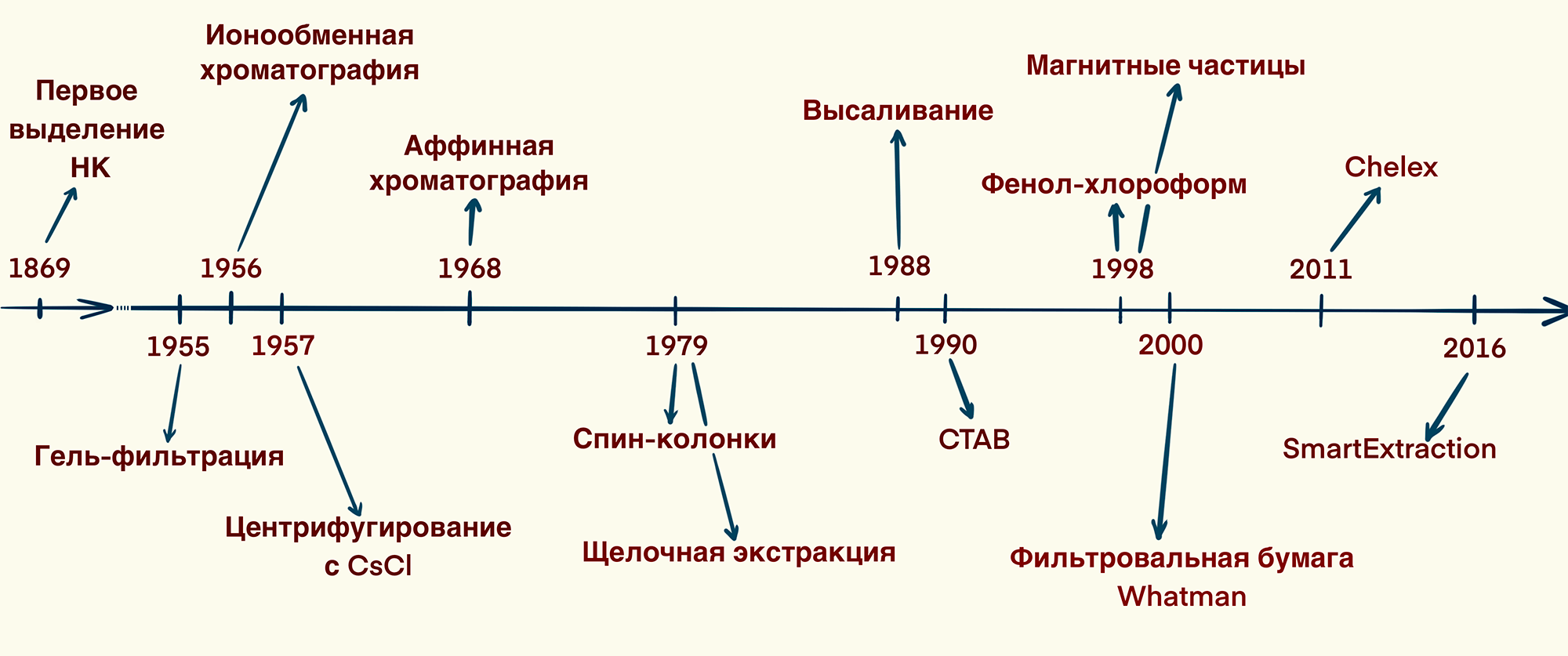
Рисунок 2. Хронология развития различных методов выделения нуклеиновых кислот
рисунок Элины Стояновой по [10]
В 1957 году Мезельсон и Сталь разработали иной протокол для извлечения ДНК, в котором для выделения ДНК из бактерий E. coli использовалось центрифугирование в градиенте плотности. В 1988 году Миллер с коллегами применили для экстракции ДНК протеиназу K — фермент, который расщепляет пептидные связи в белках и способствует их отделению от нуклеиновых кислот. А еще позже Сэмбрук и Рассел предложили один из самых популярных методов экстракции ДНК при помощи фенол-хлороформа и изоамилового спирта.
Так к середине XX века были разработаны основные принципы работы с нуклеиновыми кислотами.
Основные принципы выделения
Цели экспериментов могут быть разными, но все исследователи скорее будут отдавать предпочтение тем методам, которые:
- Специфичны и подходят под особенности объекта, из тканей которого осуществляется экстракция.
- Экономичны по времени.
- Просты и не требуют высокоспециализированного оборудования.
- Включают в себя использование безопасных реактивов.
- Могут быть модифицированы под методы, с которыми предстоит дальнейшая работа.
- Максимально защищены от загрязнения (контаминации).
Выделение нуклеиновых кислот делят на три основных этапа (рис. 3):
- Лизис клеток — физический или химический. Так, если мы выделяем нуклеиновые кислоты из листьев растения, то сперва необходимо их протереть (физически нарушить целостность клеток), а потом использовать специальный буфер. Возможно, также понадобится нагревание (для ускорения лизиса) или центрифугирование образцов (для разделения на фракции).
В качестве химического агента для лизиса используют специальные буферы. Они могут содержать буферные соли (например Tris-HCl) и ионные соли (например NaCl) для регулирования pH и осмолярности лизата. Иногда добавляют детергенты (такие как Triton X-100 или SDS), чтобы разрушить клеточную мембрану. - Очистка. После разрушения клеточной и ядерной мембран возникает необходимость удалить примеси — другие вещества, из которых состоят клетки. Добавление концентрированного солевого раствора приводит к выпадению осадка, в котором содержатся белки, липиды и сахара. Помним, что если они останутся вместе с ДНК, то изрядно навредят при ПЦР и/или секвенировании. Нуклеиновые кислоты остаются в растворе, лучшему отделению их от осадка способствует центрифугирование. Удалению белков также помогает добавление протеазы, которая их расщепляет и позволяет отделять от нуклеиновых кислот. Так можно избавиться, например, от белков-гистонов, на которые «намотана» ДНК в хромосомах.
- Элюирование. Теперь нуклеиновые кислоты необходимо удалить из раствора. Тут пригодится знание химических свойств интересующих нас кислот — они растворимы в воде, но не в спирте. Выходит, стоит нам добавить этанол или изопропиловый спирт, как появится осадок — это наша ДНК (или РНК). Осадок можно отделить от раствора при помощи центрифугирования. После удаляем спирт, и готово.
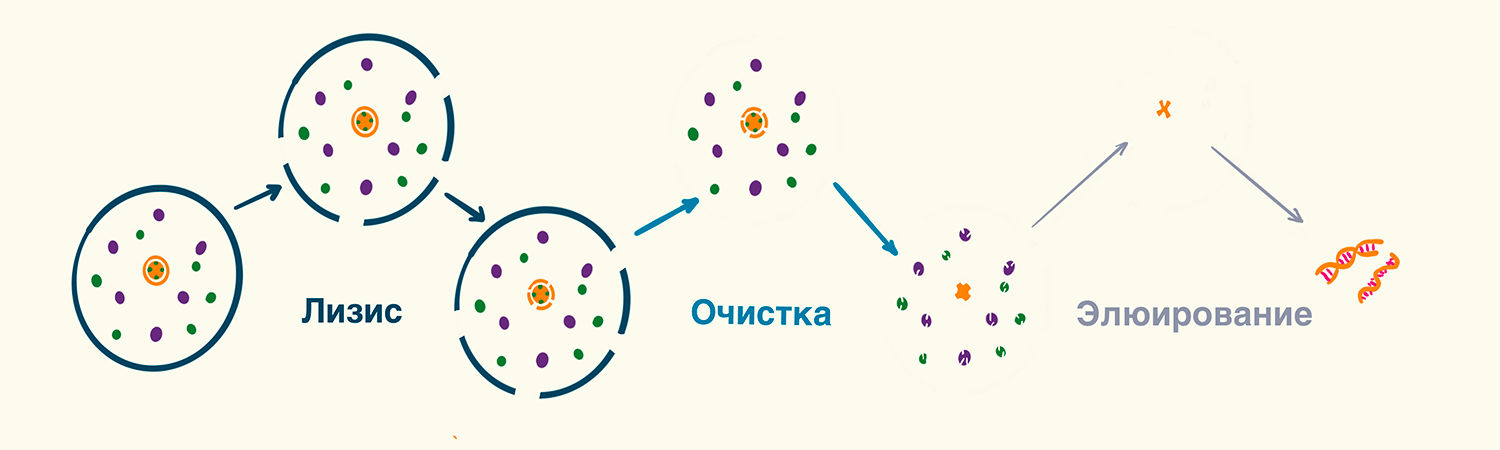
Рисунок 3. Основные этапы выделения нуклеиновых кислот. Сперва происходит лизис клеточных стенок, а затем и ядерных мембран. После осуществляется деградация белков, в том числе гистонов. Затем нуклеиновые кислоты отделяются от других клеточных компонентов.
рисунок Элины Стояновой по [11]
При выделении нуклеиновых кислот также важно отделить ДНК от РНК. Для этого полученный образец нуклеиновых кислот можно обработать рибонуклеазами, приводящими к деградации РНК, — тогда в растворе останется цельная ДНК; или же добавить дезоксирибонуклеазы — так получим РНК.
Выход ДНК можно оценить с помощью различных методов: определения оптической плотности, электрофореза в агарозном геле или с использованием флуоресцентных ДНК-связывающих красителей. Все три метода удобны, но имеют разные требования с точки зрения необходимого оборудования, различаются простотой использования и расчетов.
Оптическую плотность измеряют при 260 нм (A260). При такой длине волны ДНК наиболее сильно поглощает свет, и полученное число позволяет оценить концентрацию раствора. Сильное поглощение около 230 нм может указывать на то, что в очищенной ДНК присутствуют органические соединения или хаотропные соли. Отношение значений, полученных при 260 нм и 230 нм, показывает загрязненность ДНК солями. Концентрацию и выход можно определить после завершения гель-электрофореза путем сравнения интенсивности свечения полосы с ДНК образца с интенсивностью образца стандарта (маркера) [12], [13].
Основные реагенты для выделения
В основе разных методов выделения лежат три базовых принципа, но это не всё, что их объединяет. Многие подходы схожи еще и составом «действующих лиц». Вот наиболее часто встречающиеся:
- трис-буфер — контролирует рН, взаимодействует с липополисахаридами и повышает проницаемость, а также лизирует клеточную мембрану;
- ЭДТА — работает как хелатирующий агент, блокируя потребность в кофакторе фермента ДНКазы, тем самым предотвращая деградацию ДНК;
- SDS — солюбилизирует белки ядерных и клеточных мембран;
- NaCl — нейтрализует отрицательный заряд ДНК, стабилизирует молекулу;
- MgCl2 — агент, который защищает и стабилизирует ДНК, блокируя отрицательный заряд липопротеидов;
- фенол — осаждает белковые примеси.
Методы выделения нуклеиновых кислот
Теперь перейдем непосредственно к методам выделения. Как мы уже рассказали, Мишер определил основные этапы, необходимые для извлечения нуклеиновых кислот. Но время шло, появлялись новые знания о функциях и значении ДНК и РНК — и, конечно, другие методы работы с ними. Всё вместе стало причиной необходимости оптимизации выделения: сокращение времени на выделение, увеличение выхода и качества нуклеиновых кислот. Ученые по всему миру приступили к экспериментам по изменению этапов изначальной методики и за более чем 150 лет значительно ушли вперед (рис. 4).
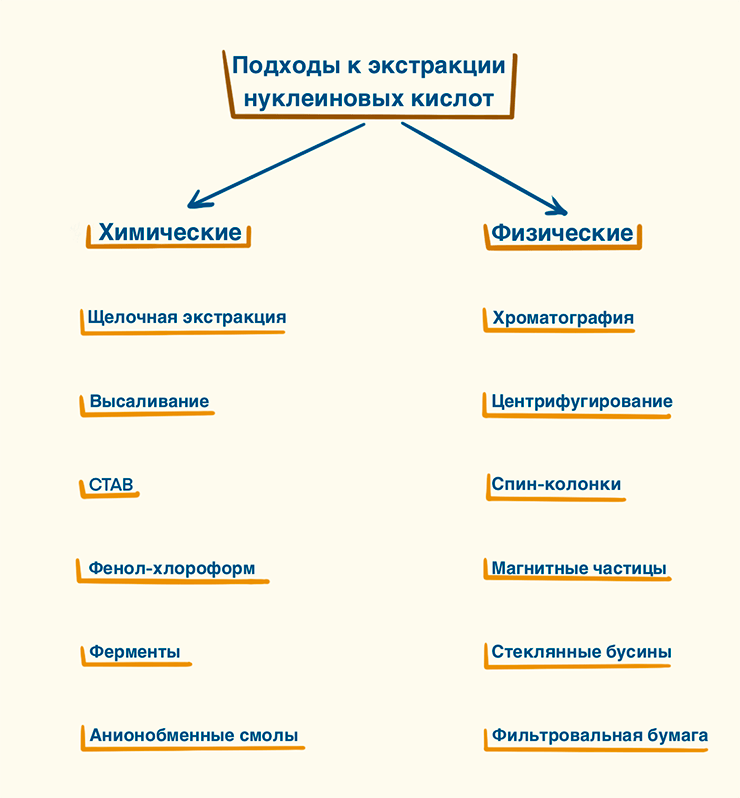
Рисунок 4. Классификация методов выделения нуклеиновых кислот. Условно методы по выделению ДНК можно разделить на химические и физические. В первых задействованы химические свойства веществ, например, фенол-хлороформа или протеиназы K, в основе вторых — разделение согласно с физическими особенностями молекул.
В таблице 1 мы просуммировали основные характеристики методов, о которых говорится в статье. Что выбрать — вопрос к исследователю.
| Метод экстракции | Объекты | Преимущества | Недостатки |
|---|---|---|---|
| Хроматография SEC | Любые | Прост в использовании, относительно быстр | Сравнительно невысокий выход и качество НК. Не может эффективно отделять ДНК от РНК |
| Хроматография IEC | Любые | Прост в использовании, относительно быстр. Хорошо отделяет ДНК от РНК | Сравнительно невысокий выход и качество НК |
| Центрифугирование + EtBr-CsCl | Любые | Может эффективно изолировать плазмиды, а также геномную ДНК бактерий | Токсичные реагенты, дорогостоящий и трудоемкий. Чистота и выход НК относительно невелики |
| Щелочная | Предпочтительно — бактерии | Лучше для выделения плазмидной ДНК | Загрязнение выходного раствора хромосомной ДНК |
| Спин-колонки | Любые | Быстрый, простой и безопасный. Можно менять фильтры и отделять геномную ДНК от плазмидной или РНК. | Дорогой, сравнительно невысокий выход |
| Высаливание | Кровь, культура клеток, гомогенат | Бюджетный, безопасные реагенты | Может занять много времени |
| CTAB | Растения | Эффективный метод для работы с растительными тканями, богатыми полифенолами и т.д. | Трудоемкий метод, возможна комбинация с другими подходами с использованием токсичных реагентов |
| Ферменты | Любые | Прост, безопасен, дает хорошее качество и выход НК | Дороже по расходным материалам, иногда требует длительной инкубации |
| Фенол-хлороформ | Любые | Хороший выход и качество НК | Токсичные реагенты, длительность |
| Магнитные частицы | Любые | Прост, быстр, точен, безопасен | Требуется настройка магнитного прибора |
| Стеклянные бусины | Любые | Можно комбинировать с нетоксичными буферами, хороший выход | Загрязнение итогового элюата бусинами |
| Chelex-100 | Любые | Быстрый, экономичный, безопасный | Не удаляет примеси достаточно для дальнейшего использования с применением многих других методов |
| SmartExtraction | Любые | Нет необходимости в большом количестве оборудования, не токсичен, хороший выход | |
| Ферментативное температурно-зависимое | Любые | Быстрый, недорогой | Для небольшого количества образца |
| Фильтровальная бумага | Любые | Экономичен, прост, безопасен, хороший выход | Не работает с малыми объемами |
Методы-пионеры
Изначально появление методов по выделению нуклеиновых кислот сопровождалось новаторским взглядом ученых, использующих «подручные» средства и методы для покорения нового типа молекул. Но вскоре в экспериментах по выделению стала появляться упорядоченность; уже устоявшиеся подходы модифицировались, что привело к появлению современных и привычных нам методов. О первых настоящих методиках выделения нуклеиновых кислот мы сейчас и поговорим.
Задействуем хроматографию: SEC и IEC
Новые методы, основанные на добавлении дополнительных принципиально иных стадий, появляются во второй половине XX века. И открывают «гонку вооружений» среди молекулярных биологов и генетиков Грант Генри Лате и Колин Рутвен в 1955 году [10], [14]. Они начинают использовать эксклюзионную, или гель-фильтрующую хроматографию (SEC, size-exclusion chromatography) .
Сперва колонку наполняли гелем из крахмала, позже декстрана, а потом черед дошел и до привычных сейчас агарозы и полиакриламида.
Хроматография — чрезвычайно важный метод для разделения самых разных молекул и смесей. Поэтому прежде чем читать дальше, советуем ознакомиться с ее базовыми принципами по статье «12 методов в картинках: очистка молекул и разделение смесей» [6]. — Ред.
Суть метода такова (рис. 5): полую трубку заполняют плотно упакованным и гранулированным полимером (допустим, агарозой). В каждой грануле есть как сквозные отверстия, так и выемки на поверхности, что многократно увеличивает их общую поверхность. Анализируемый раствор добавляется сверху, после чего наступает томительный период ожидания, пока он пройдет сквозь колонку — разделяясь при этом на фракции. Чем меньше молекулы, тем активнее они будут оседать на гранулах, «заблуждаясь» в выемках, и медленнее проходить по колонке. В свою очередь, крупные молекулы просто «проскочат» между гранулами, не заходя внутрь извилистых туннелей, и быстрее доберутся до финиша — противоположного конца трубки. Выходит, что основополагающий принцип метода — зависимость скорости прохождения через колонку от размера молекулы: чем она меньше, тем дольше окажется ее путешествие. Такие молекулы, как мРНК и белки, проникают через маленькие поры и каналы гранул, в то время как длинные фрагменты ДНК даже не заходят в них, а потому вымываются из колонки быстрее [15].
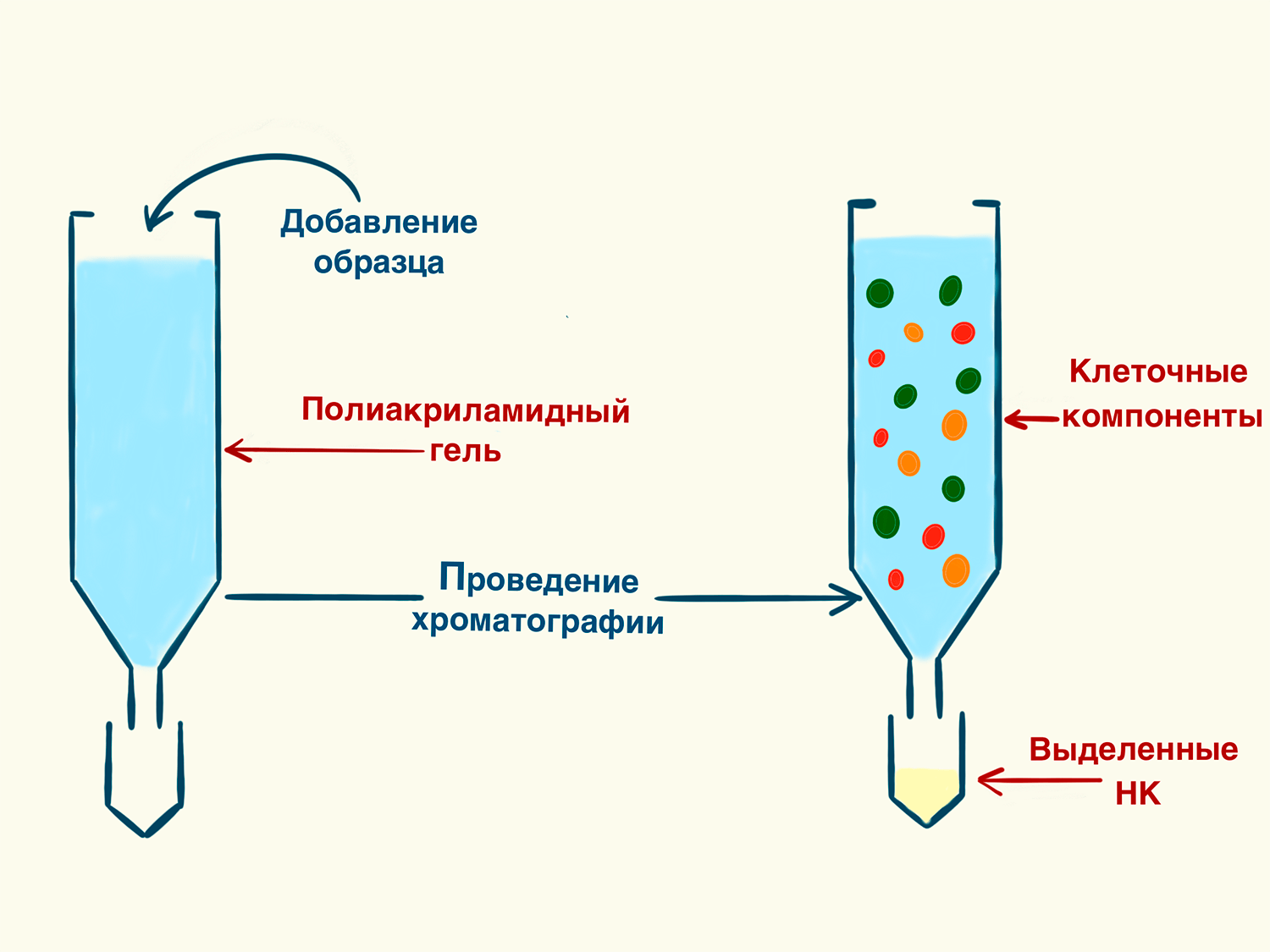
Рисунок 5. Принцип хроматографического выделения. Образцы помещают в полимер, например, полиакриламидный гель. По мере прохождения через него клеточные компоненты разделяются на фракции, в зависимости от размера молекул. Так, ДНК проходит через полимер быстрее, нежели мРНК или белки, продлевающие свой путь «путешествием» через поры в гранулах геля.
рисунок Элины Стояновой по [11]
Следующим методом стала ионообменная хроматография (IEC). В 1956 году Э.А. Петерсон и Г.А. Собер опубликовали статью, в которой рассказали о применении метода для работы с белками. В качестве матрицы внутри колонки использовали диэтиламиноэтилцелюллозу, которая связывается с молекулами ДНК и удерживает их в течение длительного времени, в то время как белки, липиды, метаболиты и РНК вымываются солевым буфером [16].
Качество и количество нуклеиновых кислот, которые удавалось получить после проведения хроматографии, были относительно невысоки. Необходимо было пробовать и искать другие подходы, и исследователи задействовали центрифугирование, ведь разница в плотности разных групп клеточных компонентов позволяла смело обратиться к новому для работы с нуклеиновыми кислотами подходу.
Прибегаем к центрифугированию в градиенте хлористого цезия
Пятидесятые годы XX века оказались богаты на новые подходы к выделению нуклеиновых кислот. Следующим стало центрифугирование, которое стали применять после добавления к клеточному лизату хлористого цезия (CsCl).
При центрифугировании молекулы в пробирке разделяются на слои по градиенту плотности, который возникает в результате осаждения из-за действия центробежных сил (седиментация) и перемещения молекул из областей с высокой концентрацией в области с низкой (диффузия), что также становится причиной разделения на фракции (рис. 6). Добавление CsCl приводит к тому, что молекулы ДНК осаждаются только до того положения в пробирке, при котором плотность градиента окажется равной ее собственной плотности, и остаются там, образуя полосу. После образования полосы ДНК ее удаляют из центрифужной пробирки. Сам CsCl можно удалить из образца путем осаждения ДНК этанолом. Основным преимуществом этого метода является возможность отделения плазмидной ДНК от геномной за счет их различий в компактизации нуклеиновой кислоты. При визуализации с добавлением бромистого этидия плазмидную ДНК легко отличить от геномной [17].
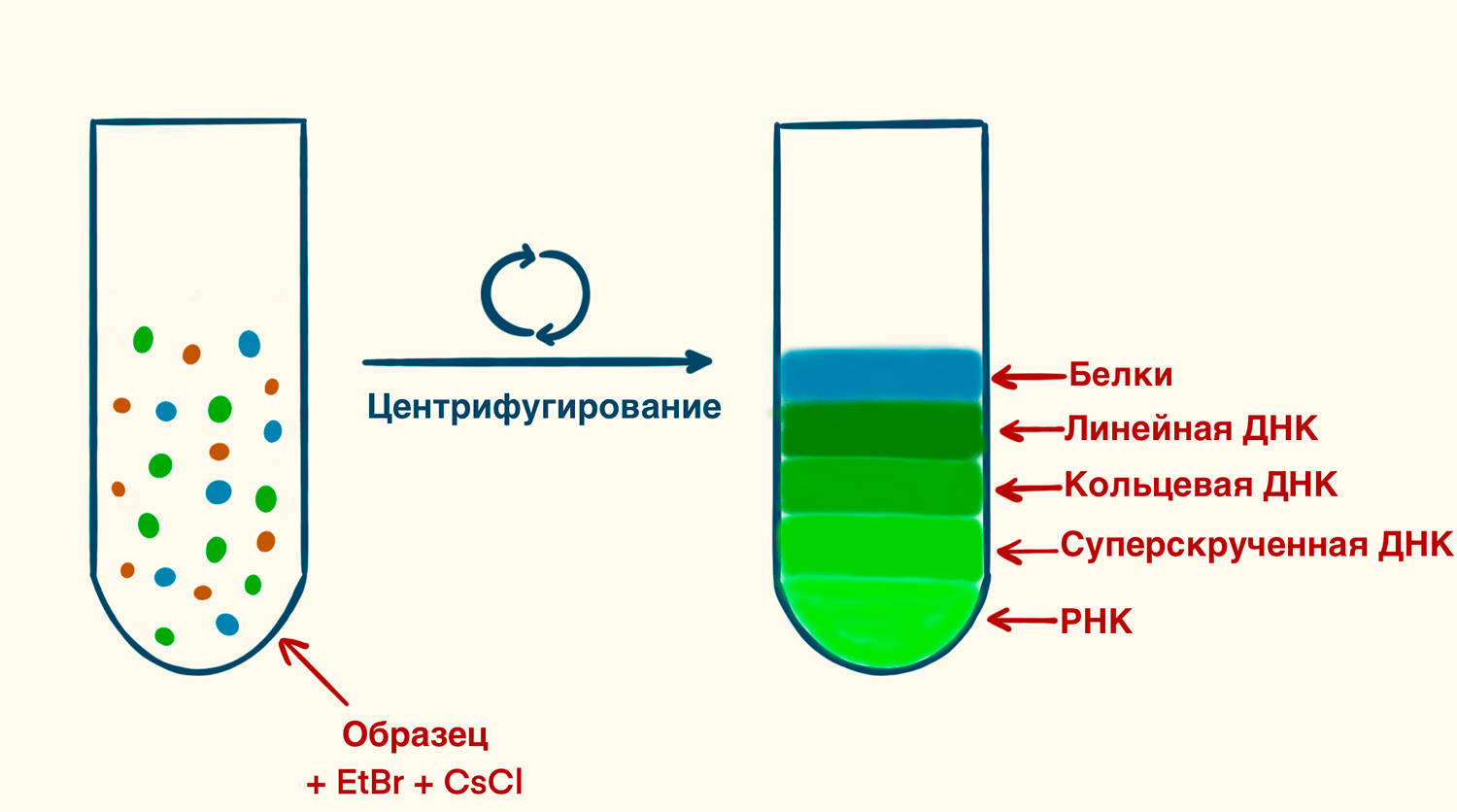
Рисунок 6. Разделение клеточных компонентов, в том числе и разных типов нуклеиновых кислот, при помощи центрифугирования. Так, седиментация и диффузия приводят к разделению молекул в растворе на фракции по градиенту плотности.
рисунок Элины Стояновой по [11] и [17]
Скорость центрифугирования в этом методе достаточно высока, и от нее зависит время, необходимое для эксперимента. При использовании высокопроизводительных центрифуг со скоростью 200 000 g разделение занимает около 16 часов. Согласитесь, несколько «черепаший» метод!
Хроматография и центрифугирование были основаны в первую очередь на физических свойствах молекул нуклеиновых кислот. Другим взглядом на проблему экстракции стали попытки применить химические агенты для отделения нуклеиновых кислот от других компонентов. И первым среди них стала щелочь.
Используем щелочную экстракцию
В 1979 году в журнале Nucleic Acids Research Дж. Доли и Х.Ч. Бирнбойм опубликовали статью с описанием щелочной экстракции нуклеиновых кислот [18]. Особенно актуален метод оказался для работы с плазмидной ДНК. Почему? Разбираемся...
Для лизиса к исследуемым клеткам добавляют смесь гидроксида натрия (NaOH) и SDS (натриевую соль лаурилсерной кислоты). При осторожном переворачивании пробирки и инкубации в холоде происходит следующее:
- NaOH нарушает целостность клеточных стенок.
- Моющее средство SDS (будем точны: не моющее, но в мыле, шампунях и зубной пасте оно есть) разрушает клеточные мембраны.
- После этого NaOH разрушает третичную структуру ДНК — в первую очередь, геномной. Она становится линейной, разрушаются водородные связи между цепями, что приводит к образованию одноцепочечных молекул.
Последующее добавление ацетата калия приводит к ренатурации молекул ДНК и случайному образованию длинных сетей, а также совместному их выпадению в осадок вместе с SDS и белками. Казалось бы, если мы хотим выделить ДНК, то его связывание и выпадением в осадок в «общей куче» совершенно ненужный поворот событий. Но оказалось, что плазмидная ДНК восстанавливает целостность без образования сети, и ее можно отделить от осадка центрифугированием. Таким образом метод стали использовать для выделения плазмидной ДНК.
Несмотря на разделение ДНК, некоторая часть геномной ДНК может попадать в супернатант с плазмидной. Если нам необходима чистая плазмидная ДНК, это окажется недостатком метода, ведь плазмиды активно используются в генной инженерии в качестве векторов .
Подробнее о плазмидах и их значимости в генной инженерии мы писали в статьях «Молекулярное клонирование, или как засунуть в клетку чужеродный генетический материал» [19] и «Плазмиды, от которых так просто не избавиться» [20] — Ред.
Выделяем ДНК на спин-колонках
Совершенно иным подходом стало использование фильтров из кремнезема в спин-колонках. Спин-колонки — это модифицированные пробирки, в которых объем пробирки разделен фильтром из силиката. Ранее уже было показано, что ДНК связывается с силикатом в присутствии холотропных солей; этот принцип и был использован в процессе создания спин-колонок. Новый подход задействовал простые и доступные материалы и позволял выделять нуклеиновые кислоты достаточно высокого качества. Впервые использование кремнезема (силиката) при работе с ДНК описали в 1979 г. Б. Фогельштейн и Д. Гиллеспи [21]. Они показали, что в буферном растворе, содержащем NaCl, нуклеиновые кислоты могут связываться с поверхностью кремнезема. Соответственно, «заякоренные» таким образом молекулы ДНК можно отмыть от примесей и дальше «открепить» от носителя при помощи элюирующего буфера или дистиллированной воды (рис. 7).
Суть метода заключается в связывании молекул нуклеиновых кислот с иммобилизованным кремнием в колонке. Образец сперва подвергают лизису (и здесь все стандартно), а после лизат взаимодействует с кремнием в спин-колонке. Для прохождения лизата через кремниевый фильтр пробирку центрифугируют. Затем следуют этапы промывки, далее нуклеиновые кислоты элюируют. Весомым достоинством метода в настоящее время стала возможность использовать разные фильтры, которые способны отделять друг от друга геномную и плазмидную ДНК, а также РНК.
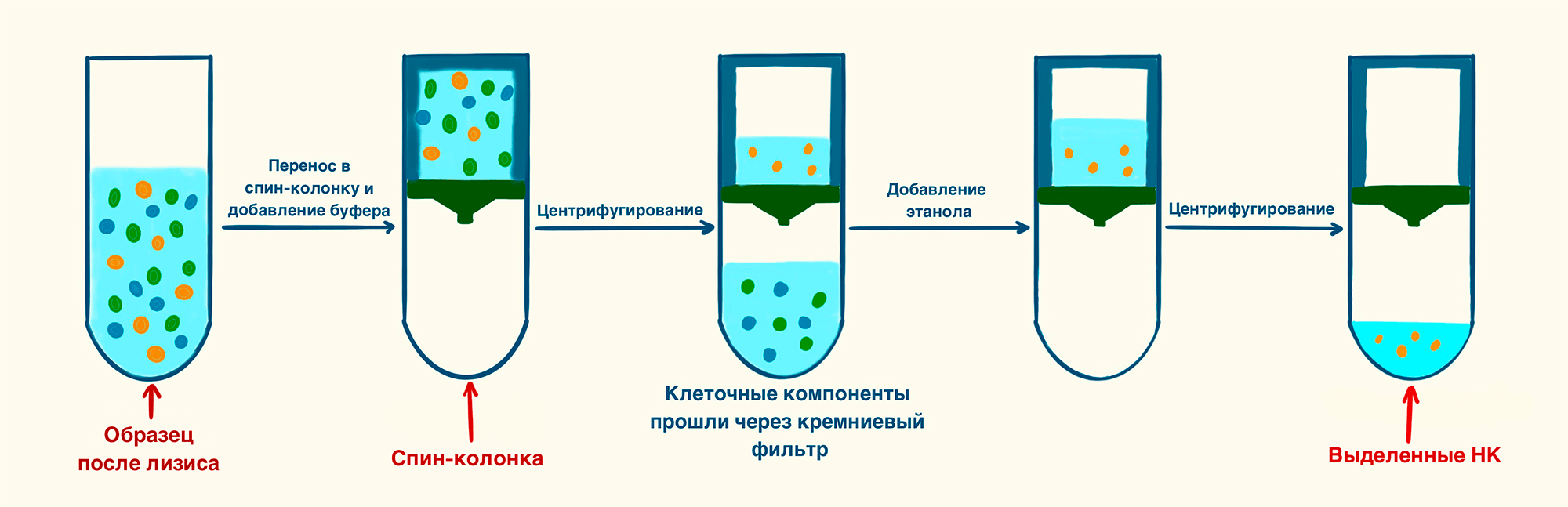
Рисунок 7. Последовательность выделения при помощи спин-колонок. После лизиса образец переносят в спин-колонку. Во время центрифугирования лизат частично проходит через кремниевый фильтр, при этом нуклеиновые кислоты остаются в спин-колонке и к ним добавляют этанол для осаждения и очищения. Последующее центрифугирование c буфером для элюции приводит к прохождению раствора с нуклеиновыми кислотами через спин-колонку.
рисунок Элины Стояновой по [11]
Метод кремнеземных матриц прост, быстр в выполнении, экономичен, позволяет получить ДНК высокого качества. Основное ограничение — одноразовость кремнеземной матрицы (хотя есть уже примеры и многократного применения [22]).
Высаливаем нуклеиновые кислоты
В 1988 году С.А. Миллер, Д.Д. Дайкс и Х.Ф. Полески описали нетоксичный подход к выделению ДНК — метод высаливания (рис. 8). В нем не задействованы ни хлороформ, ни этидиум бромид, которые ядовиты для человека и требуют дополнительных мер предосторожности при работе. Образец инкубируют в солевом буфере, способствующем осаждению белковых молекул из раствора. Последующее центрифугирование позволяет отделить белковый осадок от ДНК в растворе. Метод используют при работе с кровью, культурой клеток, гомогенатом ткани. Он прост, дешев и безопасен. Однако поскольку эффективное удаление белковых молекул требует длительной инкубации — скажем, целую ночь, — для быстрой работы он не подходит.
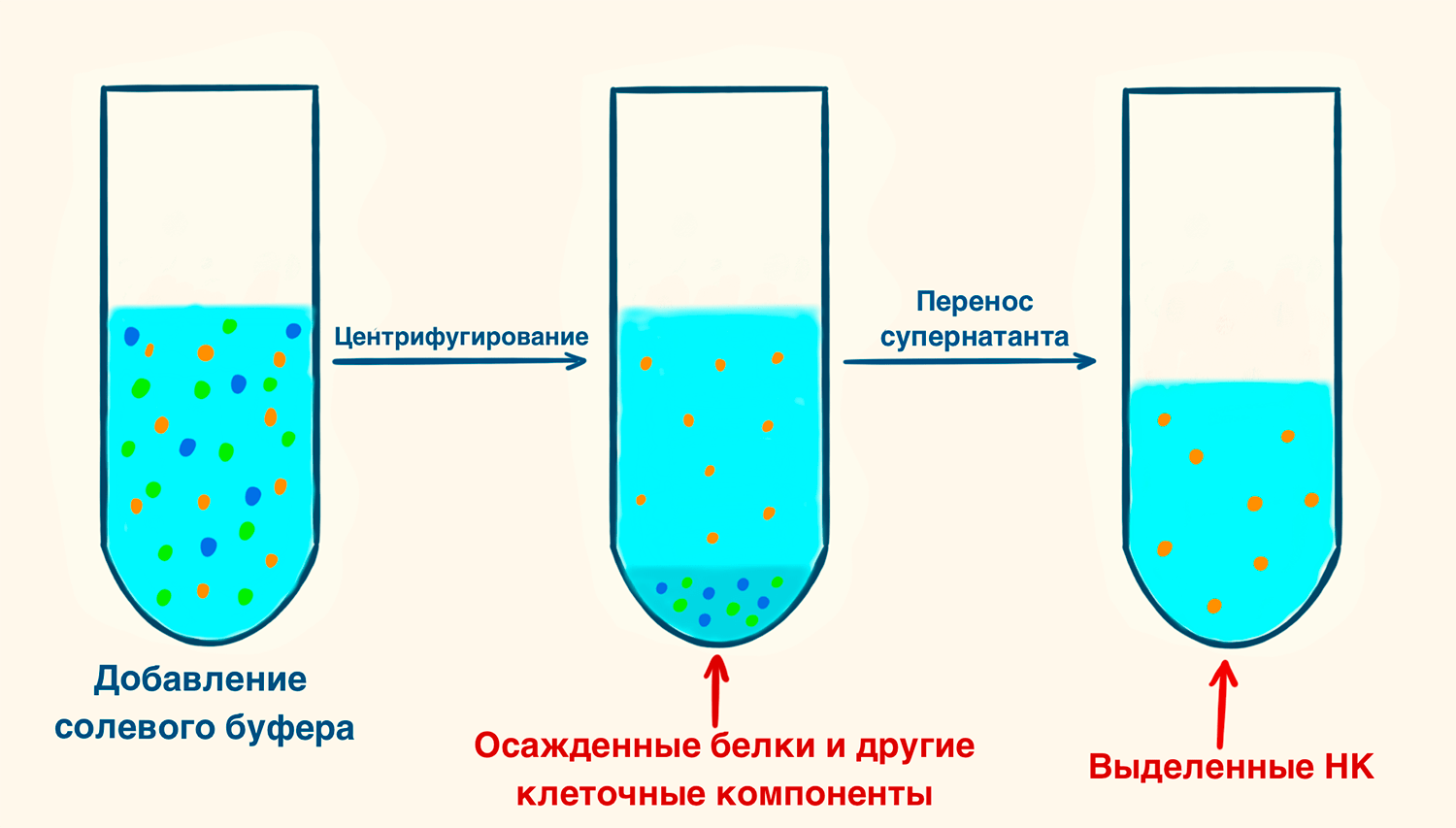
Рисунок 8. Выделение нуклеиновых кислот при помощи высаливания. Добавление к клеточному лизату солевого буфера приводит к осаждению белков и других клеточных компонентов после центрифугирования. Далее полученный супернатант с нуклеиновыми кислотами переносят в новую пробирку.
рисунок Элины Стояновой по [23]
Выделяем растительную ДНК
Следующий метод, описанный в 1987 году, до сих пор остается одним из самых используемых при работе с растительной ДНК [24]. Работа с ДНК растений необходима для исследований в области генной инженерии с целью создания новых сортов важных сельскохозяйственных культур. Также изучение растительной ДНК позволяет ученым изучать особенности лекарственных свойств растений, а еще проводить мероприятия как по сохранению редких и исчезающих видов, так и по поддержанию биоразнообразия в целом.
Ключевой особенностью метода стало использование CTAB-буфера (раствор бромида цетримониума), который отлично справлялся с очищением растительной ДНК от примесей полифенолов и полисахаридов (рис. 8). Для многих видов растений только этот метод дает достаточный для проведения ПЦР и секвенирования выход качественной ДНК. Также метод можно использовать и для работы с бактериальной ДНК.
К образцу добавляется CTAB-буфер, который осаждает нуклеиновые кислоты и некоторые полисахариды. Затем при помощи солевого раствора ДНК отделяется от полисахаридов (они выпадают в осадок) и отмывается от оставшихся примесей при помощи органических растворителей — фенола и хлороформа.
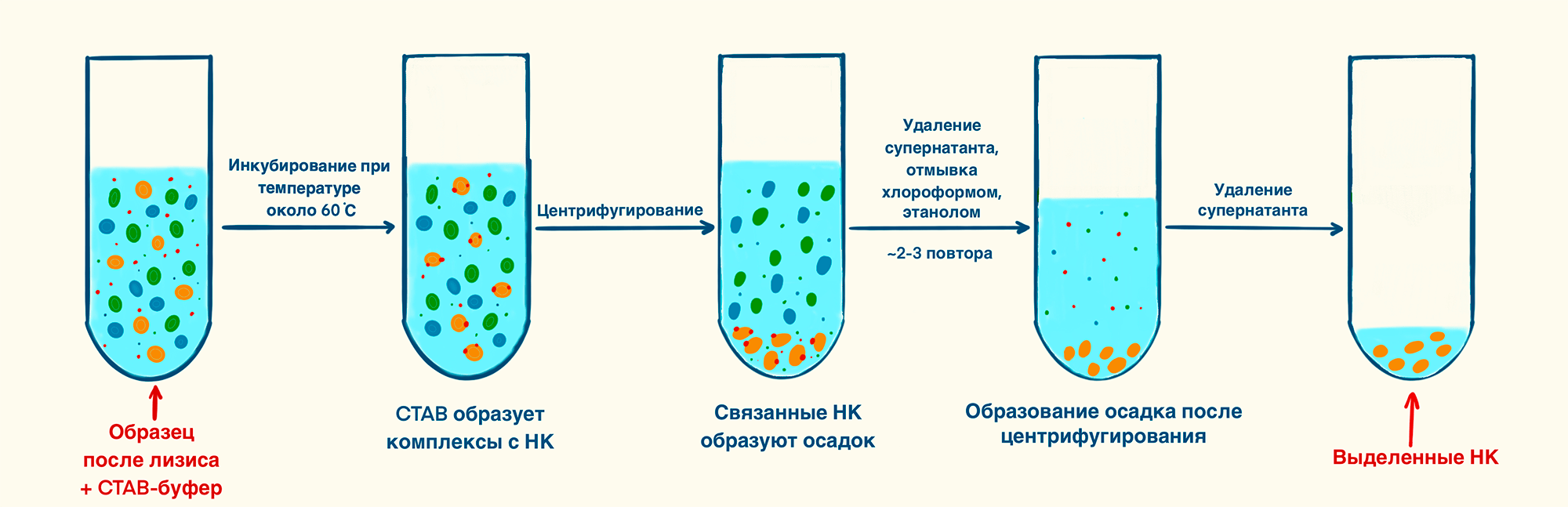
Рисунок 9. Выделение ДНК из растительного материала при помощи CTAB-буфера. К образцу после лизиса добавляют CTAB-буфер, который после инкубации образует комплексы с нуклеиновыми кислотами. После центрифугирования связанные нуклеиновые кислоты осаждаются, и можно удалить супернатант с остальными клеточными компонентами. Затем следуют этапы отмывки при помощи хлороформа и этанола. Выделенные нуклеиновые кислоты образуют видимый осадок на дне пробирки, супернатант удаляют.
рисунок Элины Стояновой по [25]
Приглашаем на работу ферменты
Одной из особенностей пространственной организации генетического материала является его тесное взаимодействие с белками. ДНК буквально намотана на белки-гистоны, и это — одно из препятствий при выделении. Для удаления белков-гистонов и нуклеаз (ферментов, расщепляющих нуклеиновые кислоты), используют протеиназу K, впервые выделенную из гриба Engyodontium album в 1974 году (рис. 10) — фермент, который умеет расщеплять пептидную связь, прилегающую к карбоксильной группе алифатических и ароматических аминокислот с блокированными альфа-аминогруппами: как раз то, что нужно, чтобы избавится от гистонов и нуклеаз.
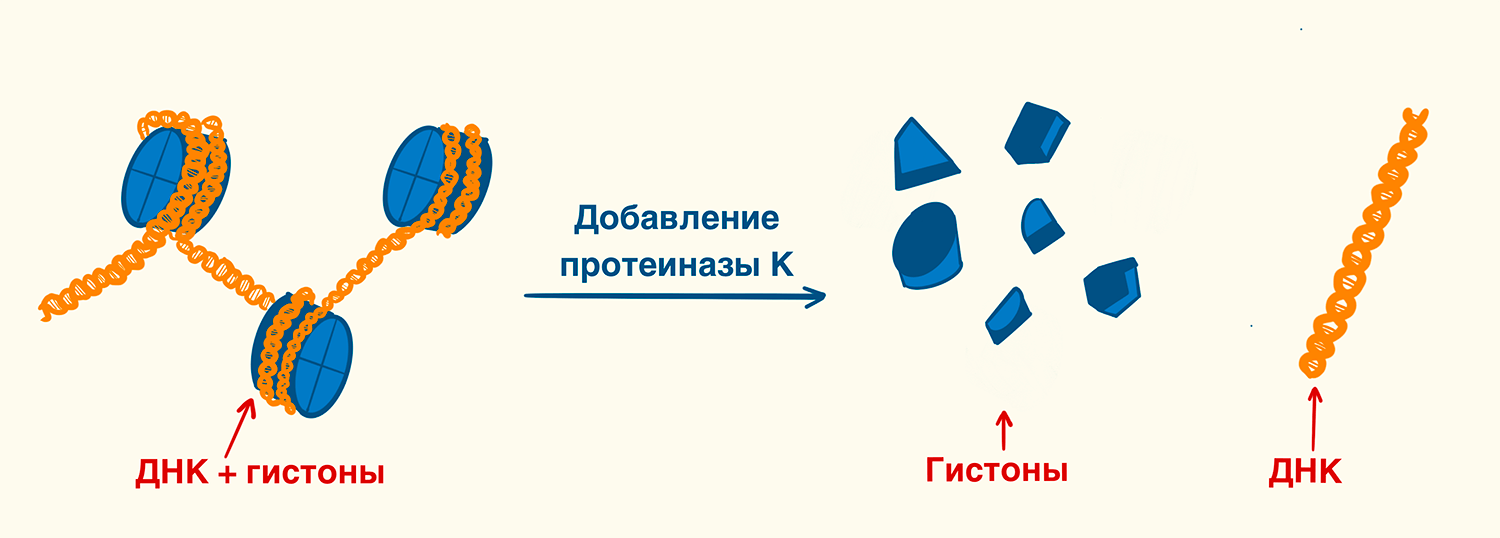
Рисунок 10. Действие протеиназы K во время выделения ДНК. Фермент расщепляет пептидную связь, что позволяет отделить нуклеиновые кислоты от белков.
рисунок Элины Стояновой по [26]
Помогает протеиназе К додецилсульфат натрия (SDS), нарушающий целостность клеточной мембраны и ядерной оболочки. После обработки пробы протеиназой К используются уже описанные методы очистки ДНК с использованием фенол-хлороформа (об этом позже) или высаливания.
Современные методы: усовершенствованные и востребованные
Параллельно с подходами к выделению нуклеиновых кислот, развивались и другие направления арсенала молекулярной биологии, такие как секвенирование, ПЦР и электрофорез. Становилось всё более понятно, что для качественных результатов необходим чистый исходный материал, и эволюция методов выделения нуклеиновых кислот продолжалась. Первые методы не ушли в тень, они модифицировались, усовершенствовались и лишь для некоторых специфических задач уступают более современным в чистоте итогового продукта и производительности.
Выделение при помощи фенол-хлороформа
Выделение ДНК фенол-хлороформом известно еще с 1998 года. Метод оказался чрезвычайно полезен при работе со «сложными» вариантами начального материала, например, тканями тропических растений, в которых много разных органических соединений. Так, многие лекарственные растения, например, широко распространенный тысячелистник обыкновенный, содержат дубильные вещества и смолы, поэтому «классический» подход к выделению в этом случае не сработает.
Принцип метода достаточно прост (рис. 11): после лизирования к образцу добавляют фенол-хлороформ-изоамиловый спирт. Фенол разрушает белки, изоамиловый спирт помогает в осаждении ДНК, а хлороформ увеличивает плотность органической примеси и предотвращает переход фенола в водную фазу, где он может нарушить целостность молекул ДНК. При перемешивании на дно пробирки оседают в первую очередь гидрофобные клеточные компоненты, например, белки. ДНК в водной части переносится в другую пробирку для дальнейшего использования или, например, концентрирования переосаждением в спирте [15].
При выделении РНК метод остается прежним с одним отличием: в нем используется фенол с кислым pH (например, 4), в то время как при выделении смеси ДНК и РНК используют фенол с pH=8. При значении pH=4 ДНК остается на границе водной и фенольной фаз, а РНК — в водной фазе [27].
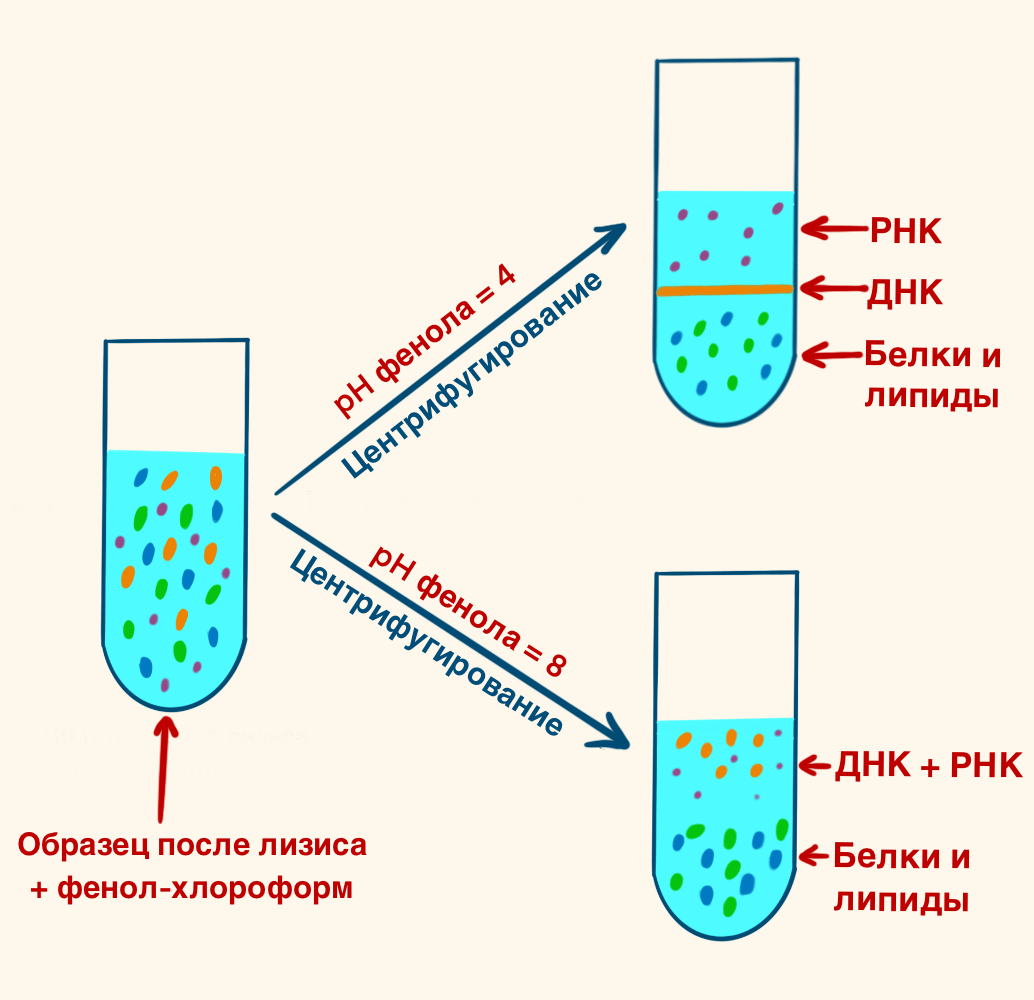
Рисунок 11. Выделение нуклеиновых кислот при помощи фенол-хлороформа. К клеточному лизату добавляют фенол-хлороформ. Фенол образует отдельную фазу, в которой находятся растворимые белки, в то время как нуклеиновые кислоты остаются в водной фазе. После центрифугирования смеси эти две фазы четко разделяются: фенольная фаза оказывается внизу, в то время как ДНК в водной фазе остается сверху. Теперь супернатант можно отделить, а ДНК промыть этанолом. Также использование фенола при разном pH раствора позволяет отделять ДНК от РНК. Так, при pH=4, они образуют отдельные фракции, в то время как при pH=8 только одну общую.
рисунок Элины Стояновой по [28]
Фенол-хлороформный метод является золотым стандартом для выделения ДНК. Он обеспечивает высокий выход и является относительно недорогим. Пожалуй, единственный недостаток — токсичность реагентов (фенола и хлороформа) и, соответственно, необходимость работы в вытяжном шкафу.
Магнитные частицы
К началу нового тысячелетия был запатентован новый подход к выделению нуклеиновых кислот, ключевым нововведением которого стало использование магнитных частиц, покрытых ДНК-связывающим агентом для взаимодействия с целевой ДНК (рис. 12).
В основе магнитных частиц — магнетит или маггемит: железосодержащие минералы, на поверхность которых нанесено кремнеземное покрытие (могут быть задействованы функциональные группы — например, гидроксильные). Нуклеиновые кислоты связываются с кремниевым покрытием в присутствии хаотропных солей, например хлорида гуанидина. Он нарушает целостность молекулы ДНК, что приводит к потере гидрофобности и связыванию нуклеиновых кислот. Магнитные частицы с нуклеиновыми кислотами на поверхности отделяются от клеточного лизата путем создания магнитного поля внешним магнитом со стороны дна пробирки. Таким образом, частицы собираются на дне, а остальной раствор можно легко удалить. После промывки и инкубации при 65 °C нуклеиновые кислоты отделяются от магнитных частиц и могут быть использованы в дальнейших экспериментах. Метод отличается быстротой и достаточно высоким выходом нуклеиновых кислот надлежащего качества. Он хорош для автоматизации высокопроизводительных процессов, поскольку не требует центрифугирования и большого количества оборудования , [29].
Об этом мы рассказывали в статье «Автоматизация омиксных технологий» [30].
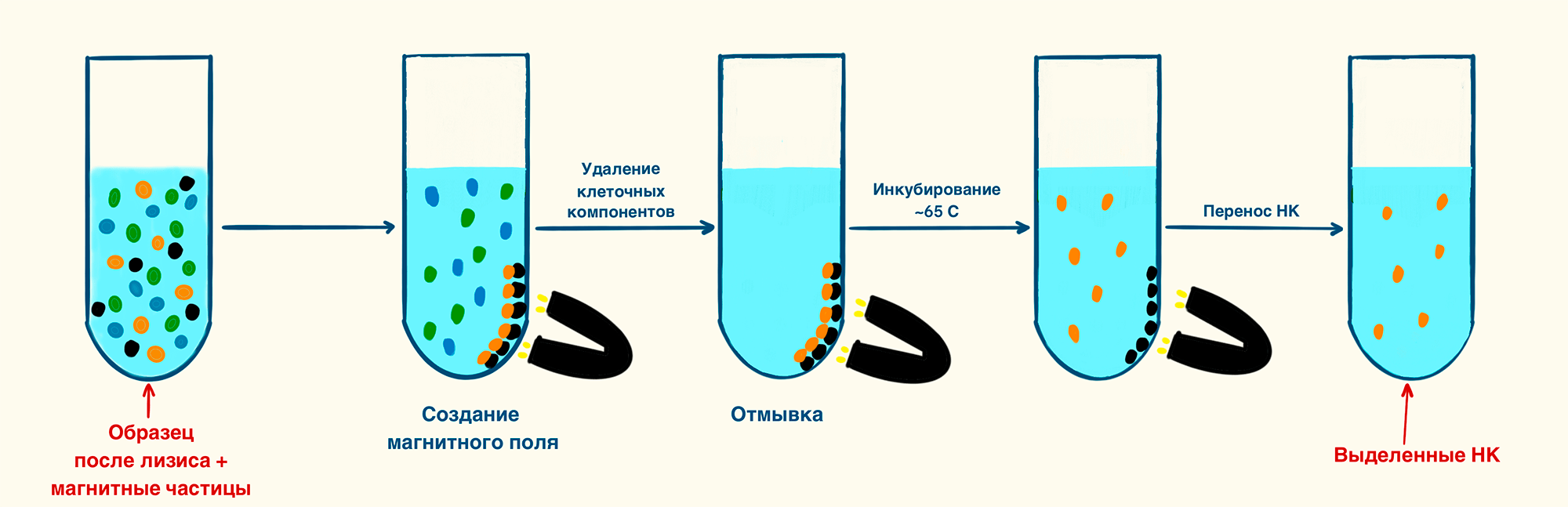
Рисунок 12. Выделение нуклеиновых кислот при помощи магнитных частиц. К клеточному лизату добавляют магнитные частицы, на поверхности которых адсорбируются нуклеиновые кислоты. Создание магнитного поля внешним источником приводит к концентрации магнитных частиц на дне пробирки, после чего остальной лизат удаляют. После отмывки и инкубирования нуклеиновые кислоты отделяются от магнитных частиц и могут быть перенесены в другую пробирку.
рисунок Элины Стояновой по DNA Purification Magnetic Beads
Использование стеклянных бусин для преципитации
Несмотря на богатый арсенал методов выделения, одной из проблем остается получение длинных неповрежденных фрагментов ДНК. Одним из подходов, позволяющих минимизировать повреждения, является использование стеклянных бусин (рис. 13). Они добавляются в образец после лизиса одновременно с добавлением спирта. Геномная ДНК прикрепляется к ним, как и в случае со спин-колонками, в присутствии хаотропных солей, вот только гладкая поверхность бусин позволяет линейной ДНК оборачиваться вокруг, сохраняя свою целостность.
Захват ДНК на поверхности стеклянных шариков упрощает этапы промывки и элюирования, устраняя необходимость в длительных этапах сушки. Держатель не дает шарикам соскользнуть, тогда как растворы быстро и эффективно удаляются из образца при вращении пробирки. После окончания всех этапов промывки ДНК удаляется с поверхности шариков нагреванием до 56 °C.
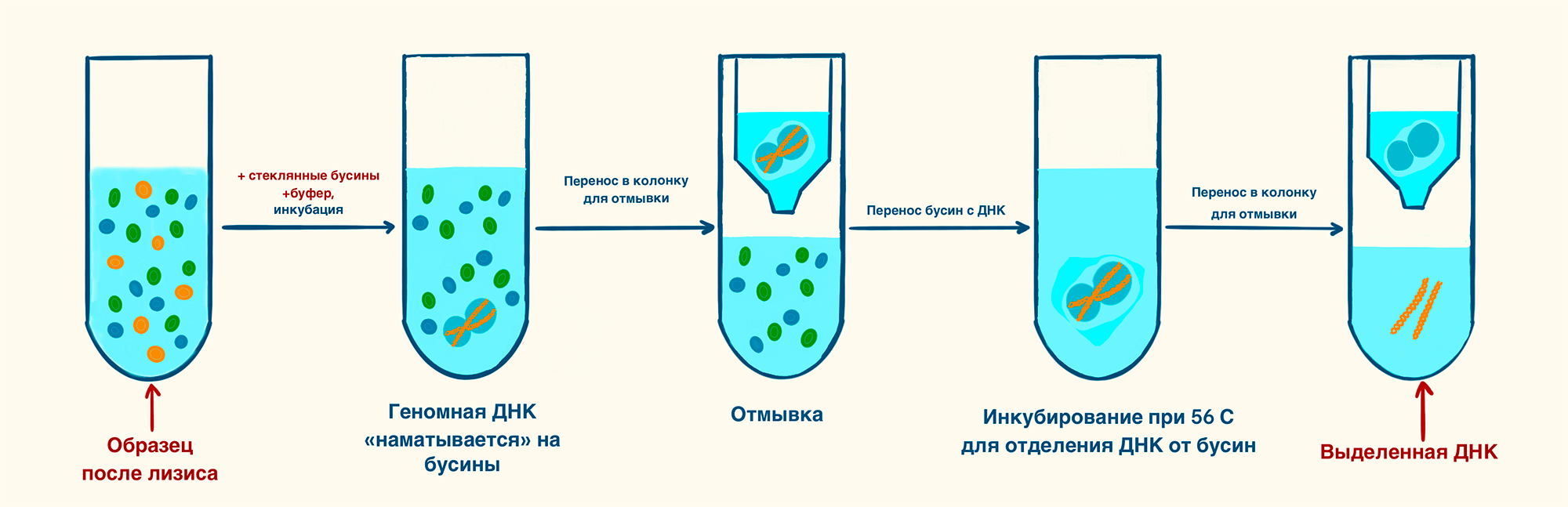
Рисунок 13. Выделение ДНК при помощи стеклянных бусин. К клеточному лизату добавляют стеклянные бусины и буфер с хаотропными солями, который способствует «наматыванию» нуклеиновых кислот на бусины во время инкубирования. После бусины переносят в колонку для отмывки от клеточного лизата. Затем бусины перемещают в другую пробирку, где после инкубирования нуклеиновые кислоты отделяются от них. Еще один этап отмывки позволяет извлечь нуклеиновые кислоты.
рисунок Элины Стояновой по [31]
Анионобменные смолы
Впервые анионобменные смолы были использованы при выделении ДНК из образцов судебно-медицинской экспертизы — спермы и пятен засохшей крови. Представленный подход оказался прост и быстр, не требовал многократного переноса образцов из пробирки в пробирку и, соответственно, снижал вероятность потери ценного биоматериала.
Chelex 100 — сополимер стирола с дивинилбензолом, в качестве хелатных групп для связывания ионов поливалентных металлов содержащий иминодиацетат. Принцип действия заключается в способности анионобменной смолы связывать заряженные клеточные компоненты (а также ферменты, способные разрушить нуклеиновую кислоту или мешающие дальнейшему прохождению ПЦР), оставляя нуклеиновые кислоты в растворе (рис. 14). Всё! Таким образом, типичный протокол получения ДНК или РНК включает: добавление суспензии смолы к образцу, кипячение для высвобождения нуклеиновых кислот, отделение раствора нуклеиновых кислот от смолы [32].
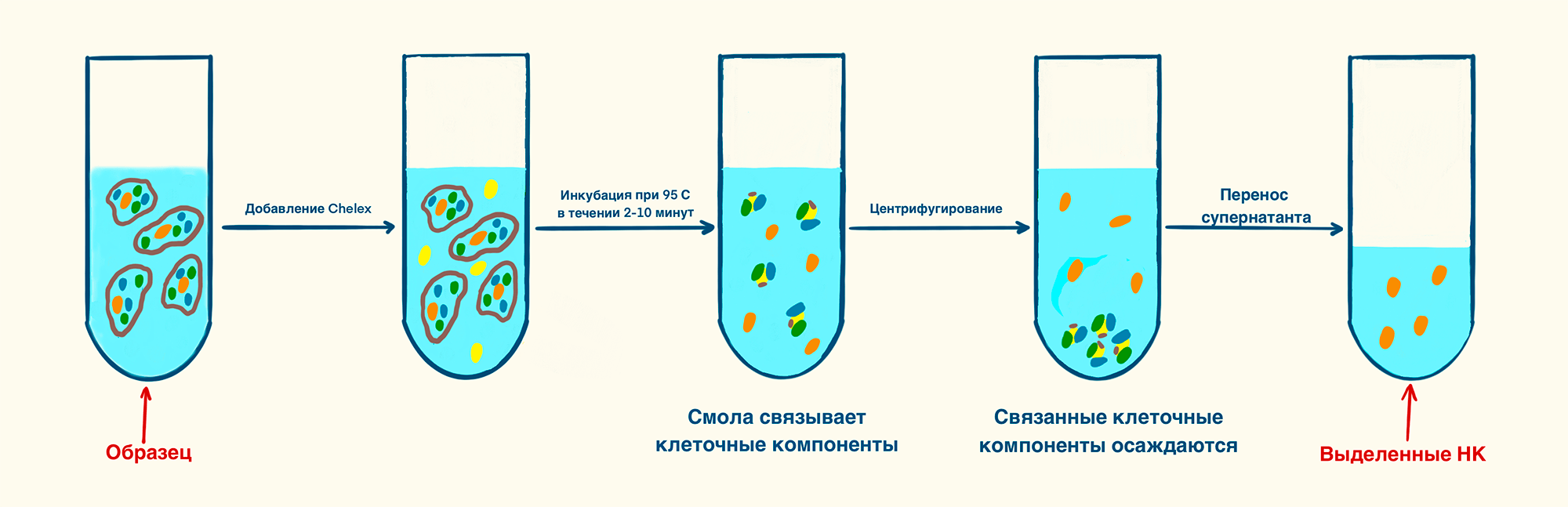
Рисунок 14. Выделение нуклеиновых кислот при помощи Chelex 100. После добавления Chelex 100 к образцу, его нагревают до 95 °C. Клеточные стенки разрушаются, а смола связывает компоненты лизата. В результате центрифугирования клеточные компоненты осаждаются, а супернатант с выделенными нуклеиновыми кислотами переносят в новую пробирку.
Полногеномные исследования, конечно же, включавшие и выделение нуклеиновых кислот на ранних этапах подготовки образца, требовали повышенного качества и количества ДНК и, желательно, удешевления методов выделения. Встречаем ультрасовременные методы!
Ультрасовременные методы
После объявления об окончании проекта «Геном человека» в 2003 году начался следующий этап эволюции уже существующих методов выделения ДНК, поскольку запрос на чистоту образца в связи с повсеместным использованием NGS вырос многократно [33]. Всё чаще новые методы разрабатывались в лабораториях компаний, которые снабжают оборудованием ученых по всему миру. Давайте посмотрим, что они предложили!
SmartExtraction
Новаторский метод выделения нуклеиновых кислот разработали сотрудники компании Analytik Jena. Для его использования нет необходимости прибегать к центрифугированию: нужны только термошейкер, магнитный штатив и, собственно, набор для выделения SmartExtraction. Выделенная ДНК обладает высокой чистотой и длиной — до пятисот тысяч пар нуклеотидов.
Сперва в пробирку добавляют образец и лизирующий буфер, содержащий протеиназу K. Клеточный лизат отбирают в наконечник, в котором расположены магнитные частицы с «умной» поверхностью (Smart Modified Surfaces). Для связывания с ними ДНК необходим оптимизатор связывания, его добавляют и инкубируют. Для отделения ДНК от остальных клеточных компонентов образец пипетируется, а магнитные частицы удаляются при помощи магнитного штатива. Чистый образец ДНК высокого качества элюируют специальным буфером (рис. 15).
В качестве буферов используют растворы хаотропных и нехаотропных солей с низкой ионной силой. Для автоматизированного выделения есть системы обработки жидкостей InnuPure C16 touch и CyBio FeliX [34].
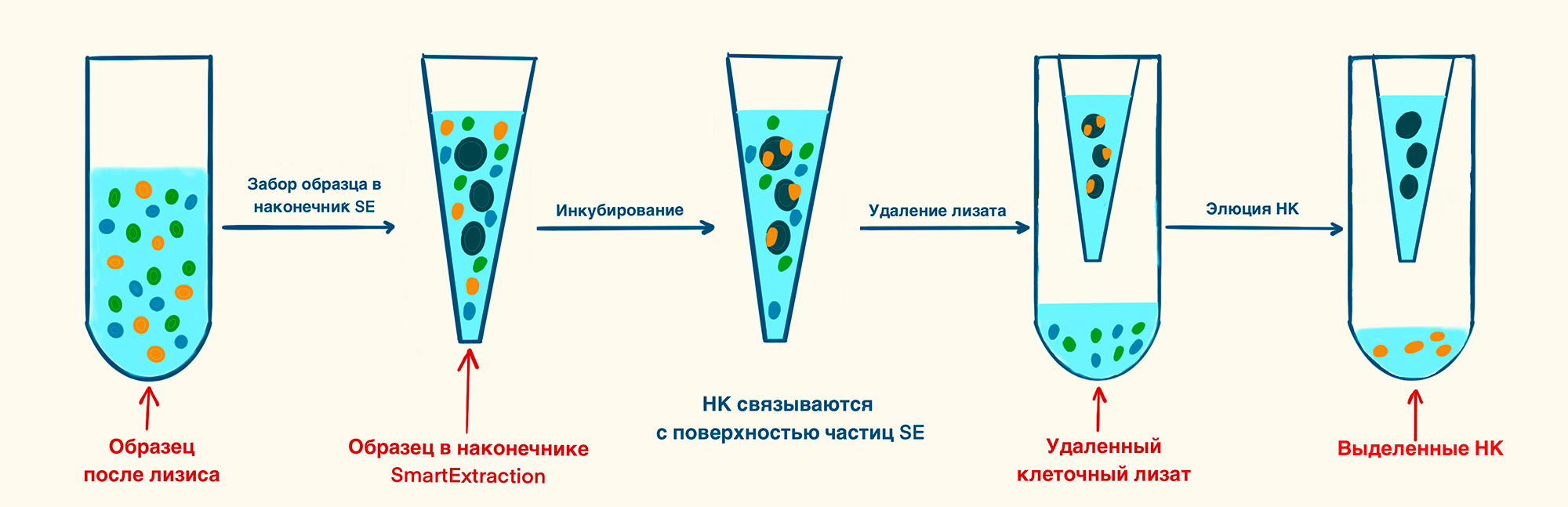
Рисунок 15. Выделение нуклеиновых кислот при помощи SmartExtraction. После стандартного лизиса ДНК вместе с лизатом забирают в специально модифицированный наконечник с частицами, на которым ДНК «оседает» после добавления оптимизатора связывания и инкубирования. Далее при использовании магнитного штатива происходит отмывка для удаления клеточных компонентов и элюирование полученной ДНК.
рисунок Элины Стояновой по [35] и smart DNA prep
Ферментативное температурно-зависимое выделение
Компания MicroGEM (Новая Зеландия) изобрела подход к выделению нуклеиновых кислот, в котором основным игроком является смесь ферментов, выделенных из мезофильных и экстремофильных организмов.
Образец вносят в разработанный компанией MicroGEM экстрактор вместе с ферментным коктейлем и помещают в термостат (рис. 16). Нагревание необходимо для активации ферментов: они начинают последовательно разрушать клеточные стенки (35–52 °C), а затем и расщеплять белки (75 °C). Дальнейшее повышение температуры приводит к сжатию стенок пробирки (и дезактивации ферментов), благодаря чему лизат вытесняется в полимер, который фильтрует ДНК и удаляет ингибиторы, такие как полифенолы и углеводы, а также ферменты, используемые для экстракции [36].
У метода относительно небольшая стоимость и высокая скорость выделения — от 7 до 20 минут. Также он отлично подходит для автоматизации.
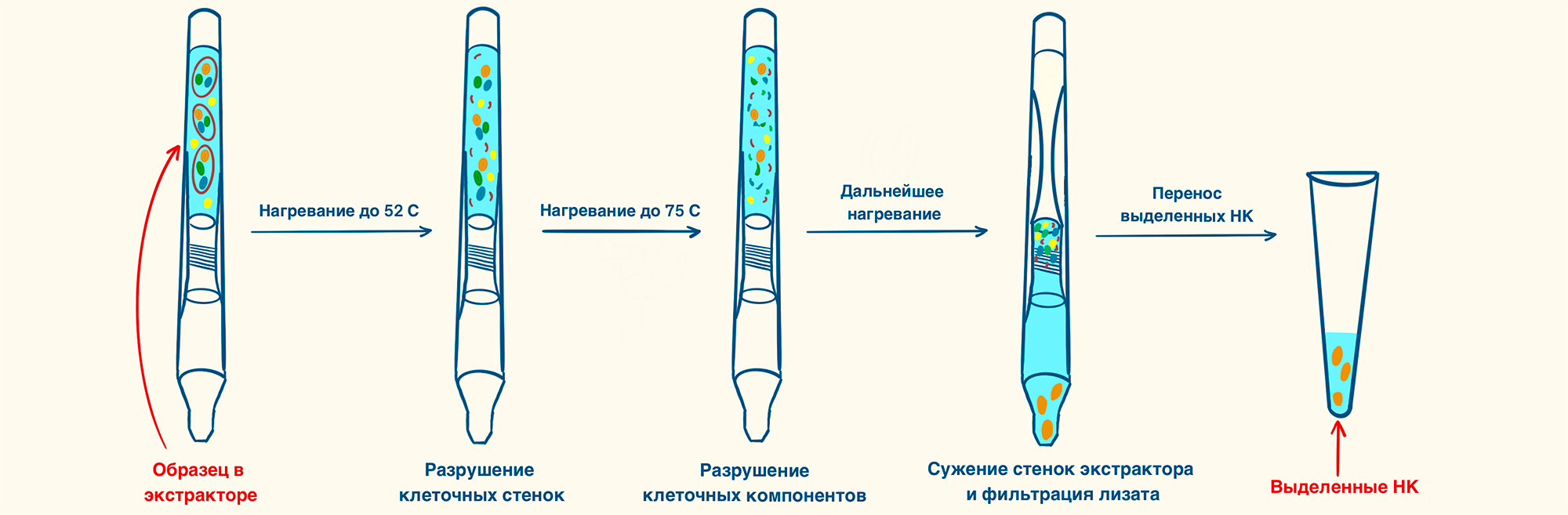
Рисунок 16. Ферментативное температурно-зависимое выделение нуклеиновых кислот. Образец забирают в экстрактор, куда также добавляют ферменты. Экстрактор помещают в термостат. Нагревание до 52 °C приводит к активации ферментов и лизису клеточных стенок, последующее поднятие температуры, вплоть до 75 °C, вызывает дальнейшую деградацию клеточных компонентов. Продолжение повышения температуры становится причиной сжатия емкости с лизатом, вследствии чего он проходит через специальный фильтр. Нуклеиновые кислоты оказываются в отдельной пробирке, а остальной клеточный лизат остается в экстракторе.
рисунок Элины Стояновой по [35]
Заключение
Любой подход к выделению включает в себя три основных пункта — разрушение клеточных и ядерных мембран, деградацию белков и отделение нуклеиновых кислот от остального клеточного содержимого. Постепенное появление и развитие ПЦР, секвенирования и других методов работы с нуклеиновыми кислотами привели к закономерной эволюции и подходов к выделению. Так или иначе, они были направлены на сокращение расходов и трудозатрат на выделение, использование нетоксичных реагентов и увеличение качества и количества выхода нуклеиновых кислот.
Появление новых методов — вопрос времени. Да, выделение нуклеиновых кислот — рутина для любой генетической лаборатории. Некоторые методы уже сейчас настолько автоматизированы, что практически не требуют участия человека. Но разнообразие биологических материалов, форм жизни и исследовательских задач позволит творчеству всегда оставаться частью процесса. Ведь к каждому организму, ткани и органу может понадобиться свой уникальный подход.
Литература
- Криминалистика. Молекулярно-генетическая экспертиза;
- Древняя ДНК: привет из прошлого;
- Крестный отец палеогеномики — лауреат Нобелевской премии по физиологии/медицине (2022);
- 12 методов в картинках: полимеразная цепная реакция;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- 12 методов в картинках: очистка молекул и разделение смесей;
- J. D. WATSON, F. H. C. CRICK. (1953). Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. Nature. 171, 737-738;
- Ralf Dahm. (2005). Friedrich Miescher and the discovery of DNA. Developmental Biology. 278, 274-288;
- Miescher F. Letter I; to Wilhelm His; Tübingen, february 26th, 1869 // Die histochemischen und physiologischen Arbeiten von Friedrich Miescher. 1897. P. 33–38;
- G. H. Lathe, C. R. J. Ruthven. (1956). The separation of substances and estimation of their relative molecular sizes by the use of columns of starch in water. Biochemical Journal. 62, 665-674;
- Chauhan T. (2018). 10 different types of DNA extraction methods (updated). Genetic Education;
- Gaikwad A.B. DNA quality: electrophoresis, spectrophotometry and fluorometry. ICAR;
- Quantification of DNA. QIAGEN;
- Lathe G.H. and Ruthven C.R. (1955). The separation of substances on the basis of their molecular weights, using columns of starch and water. The Biochemical journal. 60 (4), xxxiv;
- Preetha J Shetty. (2020). The Evolution of DNA Extraction Methods. AJBSR. 8, 39-45;
- Introduction to affinity chromatography. Bio-Rad;
- Mitchell F. (2022). DNA Purification. RR School Of Nursing;
- H.C. Birnboim, J. Doly. (1979). A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Research. 7, 1513-1523;
- Молекулярное клонирование, или как засунуть в клетку чужеродный генетический материал;
- Плазмиды, от которых так просто не избавиться;
- B Vogelstein, D Gillespie. (1979). Preparative and analytical purification of DNA from agarose.. Proc. Natl. Acad. Sci. U.S.A.. 76, 615-619;
- Karl-Heinz Esser, Wolfram H Marx, Thomas Lisowsky. (2006). maxXbond: first regeneration system for DNA binding silica matrices. Nat Methods. 3, i-ii;
- Tara C. Schmitz, Aysegul Dede Eren, Janne Spierings, Jan Boer, Keita Ito, Jasper Foolen. (2021). Solid‐phase silica‐based extraction leads to underestimation of residual DNA in decellularized tissues. Xenotransplantation. 28;
- Doyle J.J. and Doyle J.L. (1987). A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical bulletin. 19, 11–15;
- Chauhan T. (2019). Plant DNA extraction using CTAB buffer. Genetic Education;
- Proteinase K. Microbiologie clinique;
- 5 PRIME phase lock gel. (2016). GeneTargetSolutions;
- Mészáros E. (2021). Essential equipment for nucleic acid extraction. INTEGRA Biosciences AG;
- Efficient and reproducible magnetic bead DNA purification. (2019). INTEGRA Biosciences AG;
- Автоматизация омиксных технологий;
- New England Biolabs broadens its Monarch portfolio with high molecular weight DNA extraction kits for long-read sequencing. (2020). BioTechniques;
- Walsh P.S., Metzger D.A., Higuchi R. (1991). Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material. BioTechniques. 10, 506–513;
- International Human Genome Sequencing Consortium. (2004). Finishing the euchromatic sequence of the human genome. Nature. 431, 931-945;
- Sample preparation made easy. (2021). Analytik Jena;
- Андреева А. (2019). Топ-5 современных методик выделения нуклеиновых кислот. PCR News;
- A peek behind the paper — David Saul and Jo Stanton on the new method for DNA extraction. (2019). BioTechniques;
- Rui Shi, Dilip R. Panthee. (2017). A novel plant DNA extraction method using filter paper-based 96-well spin plate. Planta. 246, 579-584.