Анализ индивидуальных репертуаров Т-клеточных рецепторов
11 декабря 2015
Анализ индивидуальных репертуаров Т-клеточных рецепторов
- 5311
- 1
- 16
«Коллега, я не заслуживаю такой гомологии». Каждый Т-лимфоцит несет на поверхности молекулы своего Т-клеточного рецептора, способные распознать чужеродный антиген и защитить организм от инфекции или онкологии. Что они такое — эти рецепторы — и как в них разобраться? Насколько индивидуален их «репертуар»? О чем он может нам рассказать?
рисунок Виталины Киргизовой
-
Автор
-
Редактор
Статья на конкурс «био/мол/текст»: В организме взрослого человека насчитывается порядка триллиона Т-лимфоцитов, задача которых — защитить нас от инфекций и онкологических заболеваний. Задача эта невероятно сложная, так как различных вирусов, бактерий и других паразитов очень много, равно как и мутаций, которые могут привести к развитию онкологических заболеваний. Кроме того, паразиты умело эволюционируют, меняют хозяев — постоянно появляются новые угрозы. Таким образом, заложить в наш организм защитную реакцию к каждому отдельному патогену оказывается невозможно, да и не хватило бы для этого емкости генома. Здесь мы расскажем о том, как адаптивный иммунитет решает эту непростую задачу, а также о том, как мы можем за этим наблюдать.
Конкурс «био/мол/текст»-2015
Эта работа опубликована в номинации «Лучшая статья по иммунологии» конкурса «био/мол/текст»-2015.
Спонсором номинации «Лучшая статья о механизмах старения и долголетия» является фонд «Наука за продление жизни». Спонсором приза зрительских симпатий выступила фирма Helicon.
Спонсоры конкурса: Лаборатория биотехнологических исследований 3D Bioprinting Solutions и Студия научной графики, анимации и моделирования Visual Science.
Адаптивный иммунитет решает свою задачу — противостояние широкому и изменчивому спектру патогенов вокруг и внутри нас — пожалуй, наиболее изящным способом. Он генерирует огромное разнообразие случайных последовательностей, кодирующих антитела и Т-клеточные рецепторы (T cell receptors, TCR), уникальные (точнее, почти уникальные) для каждого клона B- и Т-клеток соответственно.
Надеемся, что слово «клон» должно быть интуитивно понятно всем, кто смотрел «Звездные войны», ну то есть практически всем :-) Применительно к Т- и B-лимфоцитам это условно точные копии клеток, несущие те же самые TCR либо продуцирующие те же самые антитела. — Авт.
В течение жизни (но преимущественно в молодом возрасте) в нашем организме образуется великое множество Т-лимфоцитов с разными TCR, каждый из которых потенциально способен распознать «свою» молекулу антигена (частичку белка чужеродного происхождения) в составе комплекса MHC (major histocompatibility complex; существует также обозначение HLA — human leukocyte antigen), презентирующего этот антиген на поверхности зараженных или опухолевых клеток (рис. 1).
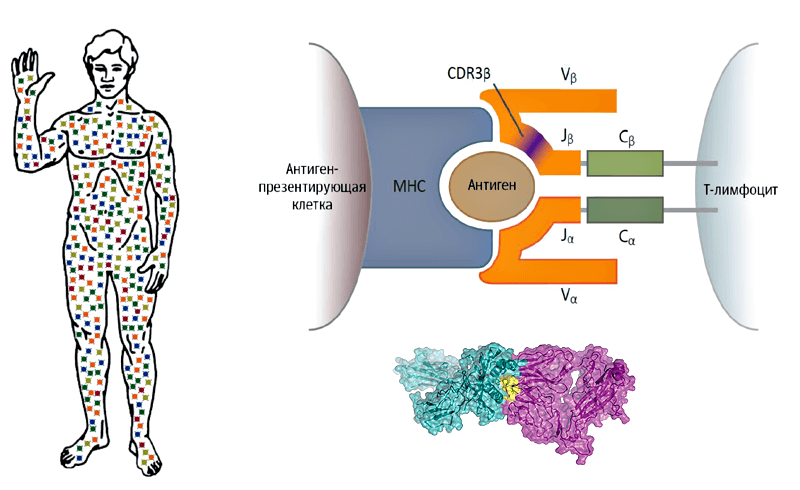
Рисунок 1. В каждом из нас — триллион Т-лимфоцитов. У них разные функции, но каждый из них несет на поверхности молекулы своего Т-клеточного рецептора, способного распознать «свою» молекулу антигена в составе комплекса MHC и защитить нас от инфекции или онкологии.
коллаж автора статьи
До того как Т-лимфоцит узнает молекулу патогена, он называется наивным Т-лимфоцитом. Такие наивные Т-лимфоциты чрезвычайно разнообразны: по современным оценкам, у каждого человека можно насчитать более 100 миллионов уникальных вариантов TCR.
Может показаться парадоксальным, но бóльшая часть этого колоссального разнообразия TCR никогда в жизни нам не понадобится. Однако именно это разнообразие является залогом того, что для новой инфекции или опухоли найдутся специфичные Т-лимфоциты, распознающие антигены, характерные для данного патогена или патологических клеток. Здесь можно провести аналогию с огромной коллекцией ключей, из которых для (почти) любой новой двери найдется подходящий вариант.
Таким образом, разнообразие TCR для каждого организма в значительной степени определяет, сколько разных антигенов он способен распознать. Чем оно выше, тем выше шансы выработать эффективный иммунный ответ при встрече с новым патогеном.
Наивные Т-лимфоциты, распознавшие свой «замόк» — антиген, — становятся эффекторными. Они активно размножаются и атакуют зараженные или злокачественные клетки, а также инструктируют другие клетки иммунной системы.
В то же время Т-клетки памяти — клональные копии эффекторных Т-лимфоцитов, участвовавших в иммунном ответе — сохраняют высокую численность на многие годы, а то и на десятки лет. Они защищают нас от повторного заболевания, наряду и во взаимодействии с B-лимфоцитами, производящими антиген-специфичные антитела и также образующими клональные популяции клеток памяти. Именно на феномене памяти адаптивного иммунитета основана практика вакцинации .
Вакцинация — одно из величайших изобретений биологии и медицины. О том, как она была открыта, как действует и как применяется в наши дни — читайте в статьях «Вакцины в вопросах и ответах» [1], «Что такое ДНК-вакцины и с чем их едят?» [2] и «Вакцинация в контексте “нано”» [3]. — Ред.
Текущий индивидуальный репертуар TCR определяет эффективность иммунной защиты, диапазон распознаваемых антигенов, особенности патологических состояний иммунитета. Более того, репертуар TCR, подобно библиотеке, содержит в себе потенциально читаемую информацию о многих инфекционных, онкологических и аутоиммунных заболеваниях человека (см. также близкую тему про репертуары антител «Следствие ведет ВироСкан. Новый подход выявляет большинство вирусов, с которыми сталкивался человек» [4]), а также об эффективности прошедшей вакцинации и последствиях проведенной терапии [5], [6].
Сегодня с помощью технологий массированного секвенирования мы научились читать «книги» в этой «библиотеке» — идентифицировать геномные последовательности для сотен тысяч вариантов TCR в образце крови или исследуемой ткани [7–9] (рис. 2).
Современные технологии определения последовательности ДНК (секвенирование) принесли с собой фантастические возможности по установлению эволюционных путей организмов, установлению причины заболеваний и поиску способов их лечения, и так далее (см. «454-секвенирование (высокопроизводительное пиросеквенирование ДНК)» [10]). Можно даже читать геномы и транскриптомы единичных клеток, что открывает захватывающие перспективы в молекулярной иммунологии и персонализованной медицине (см. «Секвенирование единичных клеток (версия — Metazoa)» [11]), однако мы еще только учимся использовать эти данные в полной мере: «Код жизни: прочесть не значит понять» [12]. — Ред.

Рисунок 2. Мы научились читать «книги» в «библиотеке» индивидуальных репертуаров Т-клеточных рецепторов
коллаж автора статьи
Правда, значения большинства «слов» — специфичности всех этих вариантов к конкретным антигенам — нам пока неизвестны. Однако мы уже можем исследовать «структуру текста» — сравнивать, сколько раз в той или иной «книге» встречается слово «любовь», как структура «повествования» меняется с возрастом, в ответ на проводимую терапию, после иммуносупрессии, трансплантации клеток крови, вакцинации; как различается состав и структура репертуара TCR для различных функциональных субпопуляций Т-лимфоцитов. Уже сегодня этот подход может рассказать нам очень многое о фундаментальной природе адаптивного иммунитета и находит все большее применение в клинических исследованиях и практических медицинских приложениях.
Попробуем немного рассказать об этом.
Формирование исходного репертуара TCR на этапе рекомбинации — из какого сора...
Огромное разнообразие вариантов Т-клеточных рецепторов, как и разнообразие антител, формируется в результате относительно случайной «сборки» — событий рекомбинации, в ходе которых из имеющегося в геноме набора так называемых V-, D-, и J-сегментов выбирается по одному варианту.
Произвольная комбинация этих сегментов собирается в новый ген, а на стыке сегментов — в местах «сварки» — происходит дополнительное случайное удаление и добавление нуклеотидов. Таким образом, место стыка сегментов V и J (D-сегмент совсем короткий, и его фактически «заваривает» в середину) характеризуется наибольшей гетерогенностью. И, как правило, именно этот гипервариабельный участок в дальнейшем играет ключевую роль в распознавании антигена — является основной «бородкой» «ключа».
Однако после первичной сборки TCR большая часть (95–98%) Т-лимфоцитов погибает в тимусе в результате позитивной и негативной селекции, направленной на то, чтобы отобрать из множества случайно генерируемых последовательностей TCR работоспособные и безопасные для организма варианты. Эту жесткую селекцию пройдут только те TCR, которые, с одной стороны, принципиально способны распознавать антигены в контексте MHC, а с другой — не проявляют сильного взаимодействия с MHC, несущими собственные пептиды организма. Такой отбор необходим для того, чтобы избежать аутоиммунных реакций — атаки Т-клеток на собственные клетки хозяина.
Интересно, что существенная часть зрелых Т-лимфоцитов помимо генов функциональных TCR несет на второй (гомологичной) хромосоме также и последовательности нефункциональных цепей TCR со сбитой рамкой считывания либо стоп-кодоном, нарушающими синтез белка. Такие TCR не экспонируются на поверхности клеток, а значит и не проходят позитивную и негативную селекцию в тимусе. Таким образом, эти бесполезные на первый взгляд последовательности могут нам многое рассказать о процессе рекомбинационных событий, в ходе которых рождается пре-селекционный репертуар TCR [13–17]. Сюда можно отнести и частоты используемых генных сегментов, из которых происходит не вполне случайная сборка TCR, и число случайно добавленных и «отгрызенных» нуклеотидов в местах соединения сегментов, и некоторые другие характеристики, определяющие исходную структуру репертуара TCR для каждого человека и в популяции.
На основе данных по репертуарам нефункциональных вариантов TCR была построена вероятностная модель сборки [16] и установлено нуклеотидное разнообразие уже для рабочих, функциональных вариантов пре-селекционного репертуара β-цепей TCR, составившее у людей (как популяции) порядка 1013, что существенно ниже, чем предсказания более простых моделей, считающих процессы выбора сегментов и каждый случайно вставленный нуклеотид независимыми событиями [13] (рис. 3). Создание такой модели позволяет исследователям генерировать искусственные репертуары TCR на компьютере, практически точно копируя поведение реальной рекомбинационной машинерии человека.
Интересно, что пре-селекционный репертуар TCR однояйцевых (генетически идентичных) близнецов характеризуется практически идентичными частотами выбора генных сегментов [17], что указывает на решающее влияние индивидуальных генетических особенностей на статистику событий рекомбинации. В то же время, например, в парах мама / свой ребенок основные характеристики пре-селекционного репертуара TCR не ближе, чем таковые для пар мама / чужой ребенок [15].
Ребенок — гость в твоем доме: накорми, воспитай и отпусти.
Индейская поговорка
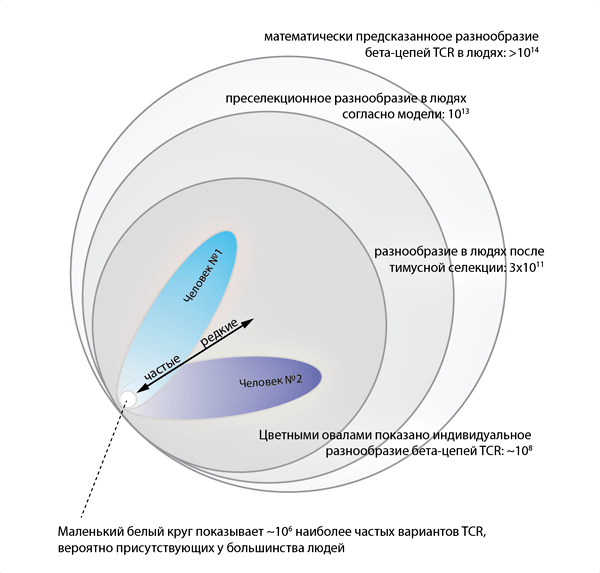
Рисунок 3. Популяционное и индивидуальное разнообразие β-цепей TCR человека
коллаж автора статьи
Селекция Т-клеточного репертуара в тимусе — прокрустово ложе
Продолжая эту логику, сравнение репертуаров нефункциональных TCR и функциональных TCR наивных Т-лимфоцитов позволяет достаточно точно проследить последствия давления отбора в тимусе. Такие исследования показывают, что тимусная селекция сопровождается выраженным изменением частот встречаемости генных сегментов, из которых собраны TCR. Однако, в отличие от событий рекомбинации, направленность этих изменений в целом сходна как у близнецов и родственных индивидуумов, так и у неродственных людей, указывая на то, что общепопуляционные эволюционные факторы доминируют здесь над изменчивостью MHC [15], [16]. Подтверждая предыдущие работы [18], [19], такой анализ показывает, что селекция в тимусе оставляет преимущественно более короткие варианты TCR, с низким числом случайно встроенных нуклеотидов [13], [15], [16], и демонстрирует определенные аминокислотные предпочтения в специфических позициях [16], что в целом приводит к существенному упрощению постселекционного репертуара. Тем не менее оставшееся разнообразие достаточно велико, чтобы охватить практически весь спектр возможных угроз.
Все должно быть настолько простым, насколько возможно, но не проще.
Альберт Эйнштейн
Индивидуальные репертуары функциональных TCR — кто больше?
Рисунок 4. Т-лимфоцит несет на себе половинки «ключа» — α- и β-цепи Т-клеточного рецептора.
рисунок Виталины Киргизовой
В 1998–1999 годах на основании экстраполяции данных секвенирования буквально нескольких десятков последовательностей TCR [20], а также с использованием метода предельного разбавления [21], индивидуальное разнообразие β-цепей TCR в одном взрослом человеке было с удивительной точностью оценено в 106–107 вариантов.
В 2009 году индивидуальное разнообразие вариантов β-цепей TCR впервые оценили на основании данных массированного секвенирования. Нижняя оценка составила ~3 × 106 нуклеотидных вариантов [7], позднее эта цифра была увеличена до ~9 × 106 [22]. Наша текущая оценка достигает 1,2 × 107 для здоровых детей и, возможно, еще вырастет в результате использования более глубокого секвенирования репертуаров TCR.
Теоретически, учитывая, что общее число Т-клеток в индивидууме достигает триллиона, а также учитывая клональность наивных лимфоцитов ввиду их экспансии в тимусе, верхнюю границу возможного разнообразия β-цепей TCR можно оценить как ~108 нуклеотидных вариантов (рис. 3). Однако каждый вариант β-цепи TCR может сочетаться с несколькими вариантами α-цепей — вместе они образуют две половинки «ключа» (рис. 4) [20]. Таким образом, максимальное индивидуальное нуклеотидное разнообразие α-β TCR может составлять ~109.
Здесь следует отметить, что при анализе данных массированного секвенирования репертуаров TCR, как и вообще при анализе данных массированного секвенирования, нужно учитывать реальный размер анализируемого образца и ограничения в точности ПЦР и самого секвенирования [23].
Действительно, если последовательность нуклеотидов TCR будет прочитана с ошибками, мы увидим вместо одного исходного клона несколько похожих друг на друга. С другой стороны, высокогомологичные варианты TCR действительно присутствуют в реальных образцах. Таким образом, для точной оценки разнообразия нужно уметь видеть и исправлять ошибки ПЦР и секвенирования, при этом сохраняя реальное разнообразие библиотеки. В противном случае мы рискуем либо во много раз переоценить реальное разнообразие, либо, напротив, потерять реально существующие варианты последовательностей TCR [23], [24].
Кроме того, технически возможно провести анализ репертуара TCR лишь для нескольких миллионов Т-лимфоцитов [22] — а это лишь верхушка айсберга из триллиона Т-клеток человека. Верхушка, обогащенная наиболее вероятными (по результатам рекомбинации и селекции в тимусе), а значит часто встречающимися вариантами TCR. Таким образом, характеристики анализируемой части репертуара не вполне точно отражают характеристики всего репертуара Т-лимфоцитов, что особенно ярко видно из следующей главы.
Пересечения между индивидуальными репертуарами: все люди — братья
Если мы случайно зачерпнем два раза по 108 вариантов β-цепей TCR из общего пула вариантов ~3 × 1011 [16], то отобранные репертуары не будут значительно пересекаться между этими двумя воображаемыми людьми.
Однако сравнение индивидуальных репертуаров TCR не только подтвердило предыдущие выводы о широкой представленности в человеческой популяции определенных вариантов TCR Т-клеток памяти [25–27], но и показало на удивление высокий уровень межиндивидуальных совпадений общего, в первую очередь наивного, репертуара TCR у неродственных людей [13], [15], [28].
Причиной возникновения множества подобных «общественных» (public) вариантов TCR являются часто повторяющиеся, или конвергентные (разные, но ведущие к тому же результату), рекомбинационные события, которые продуцируют идентичные нуклеотидные последовательности TCR. Определенные варианты TCR генерируются, проходят селекцию в тимусе и встречаются с большей вероятностью, чем остальные [7], [16], [29], [30]. Таким образом, репертуары TCR имеют выраженную иерархическую структуру: есть варианты TCR, которые встречаются у большинства людей (если не у всех), есть варианты, распространенные в популяции, есть редкие варианты, и наконец, есть уникальные рецепторы, вероятно, присущие только одному конкретному человеку (правда, ему трудновато было бы это доказать).
Кроме того, конвергентная рекомбинация также продуцирует варианты TCR с разными нуклеотидными последовательностями, но с одинаковой последовательностью аминокислот [27], [29]. Это существенно сокращает функциональное разнообразие репертуара TCR и увеличивает межиндивидуальное пересечение репертуаров на аминокислотном уровне. Непосредственно наблюдаемое пересечение аминокислотных репертуаров β-цепей TCR между двумя индивидуумами уже превысило 105 общих вариантов [28]. Более того, экстраполяция данных глубокого секвенирования на общее разнообразие рецепторов предполагает примерно 40-процентное совпадение общих аминокислотных репертуаров β-цепей TCR между двумя неродственными людьми [28].
Наиболее распространены те последовательности TCR, которые близки к структуре исходно используемых генных сегментов (характеризуются минимальным числом случайно удаленных и добавленных нуклеотидов). Вероятно, такие TCR являют собой эволюционно выработанные варианты, важные для эффективного ответа иммунной системы против наиболее распространенных инфекций, а также, возможно, выполнения определенных регуляторных функций [31]. Этот же эволюционный механизм, по всей видимости, определяет низкое разнообразие репертуаров TCR у NKT (natural killer T cells) и MAIT (mucosal-associated T cells) клеток, находящихся функционально где-то между врожденным и адаптивным иммунитетом [32], [33].
Как ни странно, общее пересечение репертуаров TCR практически не различается у неродственных людей, гаплоидентичных и HLA-идентичных родственников [13], в парах мама / родной ребенок [15] и между генетически идентичными близнецами [17]. Это показывает, что влияние наследственных факторов на формирование репертуаров TCR наивных Т-лимфоцитов не выходит далеко за рамки использования схожих частот генных сегментов в ходе рекомбинации.
Получается, что «иммунόм» (так можно обозначить совокупность генов TCR и антител человека) гораздо более индивидуален, чем генόм или транскриптóм . Действительно, геномы и даже транcкриптомы близнецов практически идентичны, а вот репертуар TCR индивидуален.
Значения этих загадочных слов, а также смысл вторжения в «классическую» биологию концепции Big Data можно узнать из статьи «“Омики” — эпоха большой биологии» [34]. — Ред.
И тем не менее, если посмотреть на самую верхушку айсберга — несколько тысяч самых встречаемых в периферической крови вариантов TCR, — окажется, что пересечение этой части репертуаров для однояйцевых близнецов значительно выше, чем для неродственных индивидуумов [17]. Наиболее вероятным объяснением этого является частая антиген-зависимая селекция Т-лимфоцитов, несущих идентичные варианты TCR, в одинаковых генетических, инфекционных и внешних условиях.
Пересечения репертуаров между функциональными субпопуляциями — бывает ли у Т-лимфоцитов Юрьев день?
В зависимости от выполняемых функций и молекулярных маркеров Т-лимфоциты подразделяют на большое количество субпопуляций. Несмотря на ключевую роль в формировании иммунного ответа и развитии патологических состояний, взаимодействие этих субпопуляций, степень их родства, а также способности Т-лимфоцитов к временному или необратимому переходу из одной функциональной субпопуляции в другую до сих пор плохо изучены.
Современное программное обеспечение для анализа данных секвенирования репертуаров TCR позволяет проводить сравнение интересующих функциональных или тканеспецифичных субпопуляций Т-лимфоцитов по широкому спектру характеристик [35], [36]. Так, относительное разнообразие репертуара TCR и процент популяции, занимаемый высокоамплифицированными клонами, могут отражать статус и степень вовлеченности субпопуляции в активный иммунный ответ. В то же время, пересечения репертуаров Т-клеточных рецепторов позволяют количественно оценить возможные пути перехода клонов Т-лимфоцитов между функциональными субпопуляциями, этиологическую близость субпопуляций, влияние того или иного воздействия или мутации на формирование субпопуляционного разнообразия TCR.
Этот подход уже был использован в целом ряде работ [37–41] и позволил ответить на ряд запутанных вопросов. Однако по большому счету его потенциал остается практически не раскрытым современной иммунологией.
А дальше? Анализ репертуаров TCR в медицине
Массированное секвенирование репертуаров TCR и антител уже применяется для регистрации злокачественного клона (минимальной остаточной болезни, МОБ) после проведенной противоопухолевой терапии у пациентов с лимфоидными опухолями. Дело в том, что, если сами опухолевые клетки имеют Т- либо B-клеточное происхождение, они могут нести уникальную перестройку гена TCR или антитела. Таким образом, уникальная последовательность гена иммунного рецептора может быть использована в качестве генетического маркера для слежения за злокачественными клетками [42–46]. Измерение уровня МОБ позволяет определить эффективность химиотерапии — удалось ли полностью избавиться от опухоли? — и сделать прогноз вероятности рецидива, что крайне важно для выбора стратегии дальнейшей терапии [46–49], такой как химиотерапия или аллогенная (от другого донора) трансплантация гемопоэтических стволовых клеток (ТГСК). Первый диагностический набор для оценки минимальной остаточной болезни в качестве прогностического маркера при множественной миеломе должен получить одобрение Food and Drug Administration (организации, обеспечивающей надзор за качеством лекарственных средств и медицинских приборов в США) уже в 2016 году.
Оценка последствий аллогенной ТГСК с точки зрения динамики последующего восстановления Т-клеточного иммунитета (а эта задача оптимально решается как раз слежением за восстановлением репертуара TCR) и возможных реакций «трансплантат против хозяина» (когда пересаженные Т-клетки атакуют организм больного) также очень важна для рационального совершенствования терапевтических протоколов и в ближайшем будущем может начать применяться для мониторинга лечения каждого пациента [50].
Другой тип трансплантации гемопоэтических стволовых клеток — аутологичная (пересадка небольшой части предварительно забранных собственных клеток пациента) — используется в сочетании с химиотерапией для лечения тяжелых аутоиммунных заболеваний [51], [52]. В этой области большой интерес представляет посттрансплантационная судьба клональных популяций Т-лимфоцитов [53]. Неожиданно для исследователей, анализ репертуаров TCR показал, что очень большое число клонов Т-лимфоцитов (как минимум сотни тысяч) переживает процедуру трансплантации, но при этом происходят выраженные изменения в их относительной представленности [54], [55]. При этом популяция CD4 (так называемые Т-хелперы) все же существенно обновляется, а вот популяция CD8 (так называемые Т-киллеры) формируется преимущественно из активно размножающихся клонов памяти, регистрируемых до терапии [56]. На основании всех этих данных сегодня ведется усовершенствование методов трансплантационной терапии аутоиммунных заболеваний и заболеваний крови.
Еще одним практическим применением анализа иммунных репертуаров является количественное определение клональности и состава проникающих в солидную опухоль Т-лимфоцитов (tumor infiltrating lymphocytes, TILs). Так, недавние исследования показали, что репертуары TIL являются относительно гомогенными, если сравнивать первичные и метастатические опухоли, но при этом отличаются от репертуаров периферической крови [57]. Анализ репертуаров TCR также потенциально позволяет предсказать ответ на иммунотерапию. Предварительные данные для различных типов рака, включая колоректальный рак, рак яичников и глиобластому, показывают, что выраженное присутствие размноженных клонов Т-лимфоцитов в опухоли может коррелировать с лучшим прогнозом для пациента.
Мониторинг изменений Т-клеточного репертуара в периферической крови также представляет интерес с точки зрения иммунологии опухолей. Так, исследования, оценивающие изменения репертуара TCR, вызванные анти-CTLA-4-терапией, показали, что лучший прогноз реакции на терапию связан со стабильностью Т-клеточного репертуара [58].
Важно, что последовательности антиген-специфичных TCR часто совпадают у разных людей. TCR предпочтительно распознают определенные мотивы [59], [60], и в контексте ограниченного разнообразия иммуногенных пептидов это ведет к возможности использования анализа индивидуальных репертуаров TCR для диагностических целей. По мере накопления информации о специфичности различных TCR и встречаемости конкретных вариантов TCR («слов») и их паттернов («предложений») при различных заболеваниях, возможности для диагностического использования информации о последовательностях TCR при инфекционных, онкологических и аутоиммунных заболеваниях должны существенно расшириться.
Накопленные сведения об антигенной специфичности конкретных TCR (а в перспективе, возможно, и алгоритмы предсказания их специфичности in silico) также могут быть применены для терапии аутологичными T-клетками или генной терапии с использованием TCR для лечения онкологических [61–63] и инфекционных [64] заболеваний. В то же время слежение за судьбой введенных клонов Т-лимфоцитов с помощью анализа репертуаров TCR может помочь в разработке более эффективных терапевтических протоколов.
Потенциально очень интересный подход к лечению аутоиммунных заболеваний может быть основан на том, что идентифицированные по последовательности аутоиммунные клоны Т-клеток могут быть избирательно элиминированы наряду с ~1/30 общего количества Т-клеток с использованием антител, специфичных к конкретному сегменту TCR, либо с использованием геноспецифичных подходов, таких как морфолины или CRISPR-технологии [65]. Анализ репертуаров TCR может быть полезен для разработки и усовершенствования методов вакцинации, а также для оценки эффективности ранее проведенной вакцинации — с использованием мониторинга динамики индуцированного Т-клеточного ответа.
Наконец, анализ репертуаров TCR позволяет точно оценить «степень старения» адаптивного иммунитета [22] и последствия проведенной химиотерапии, полученного облучения, действия факторов окружающей среды, применения медикаментов (в том числе иммуномодуляторов и иммуностимуляторов). Такой анализ может быть важен для системной оценки применяемых методов лечения, разработки более эффективных и щадящих для адаптивного иммунитета вариантов терапии и медикаментов, а также методов поддержания функции адаптивного иммунитета в пожилом возрасте.
Одним словом, современные методы анализа репертуаров Т-клеточных рецепторов, а равно и антител (о которых мы здесь не успели рассказать [66]) — это целое новое направление современной науки. Кому стало интересно — добро пожаловать в пабмед. Особенно рекомендуется следить за публикациями Georgiou G. по антителам и Robins H. по TCR.
Ну и наши поглядите :)
По правилам редакции, нам пришлось поставить в авторы одного человека, то есть меня. На деле же в работе над текстом прямо и косвенно принимали участие многие сотрудники лабораторий Геномики адаптивного иммунитета и Сравнительной и функциональной геномики ИБХ РАН: Виталина Киргизова, Екатерина Путинцева, Михаил Шугай, Михаил Погорелый и еще многие другие.
Литература
- Вакцины в вопросах и ответах;
- Что такое ДНК-вакцины и с чем их едят?;
- Вакцинация в контексте «нано»;
- Следствие ведет ВироСкан. Новый подход выявляет большинство вирусов, с которыми сталкивался человек;
- Venturi V., Rudd B.D., Davenport M.P. (2013). Specificity, promiscuity, and precursor frequency in immunoreceptors. Curr. Opin. Immunol. 25, 639–645;
- Woodsworth D.J., Castellarin M., Holt R.A. (2013). Sequence analysis of T-cell repertoires in health and disease. Genome Med. 5, 98;
- Robins H.S., Campregher P.V., Srivastava S.K., Wacher A., Turtle C.J., Kahsai O. et al. (2009). Comprehensive assessment of T-cell receptor beta-chain diversity in alphabeta T cells. Blood. 114, 4099–4107;
- Freeman J.D., Warren R.L., Webb J.R., Nelson B.H., Holt R.A. (2009). Profiling the T-cell receptor beta-chain repertoire by massively parallel sequencing. Genome Res. 19, 1817–1824;
- Mamedov I.Z., Britanova O.V., Zvyagin I.V., Turchaninova M.A., Bolotin D.A., Putintseva E.V. et al. (2013). Preparing unbiased T-cell receptor and antibody cDNA libraries for the deep next generation sequencing profiling. Front. Immunol. 4, 456;
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Секвенирование единичных клеток (версия — Metazoa);
- Код жизни: прочесть не значит понять;
- Robins H.S., Srivastava S.K., Campregher P.V., Turtle C.J., Andriesen J., Riddell S.R. et al. (2010). Overlap and effective size of the human CD8+ T cell receptor repertoire. Sci. Transl. Med. 2, 47ra64;
- Murugan A., Mora T., Walczak A.M., Callan C.G. Jr. (2012). Statistical inference of the generation probability of T-cell receptors from sequence repertoires. Proc. Natl. Acad. Sci. USA. 109, 16161–16166;
- Putintseva E.V., Britanova O.V., Staroverov D.B., Merzlyak E.M., Turchaninova M.A., Shugay M. et al. (2013). Mother and child T cell receptor repertoires: deep profiling study. Front. Immunol. 4, 463;
- Elhanati Y., Murugan A., Callan C.G. Jr., Mora T., Walczak A.M. (2014). Quantifying selection in immune receptor repertoires. Proc. Natl. Acad. Sci. USA. 111, 9875–9880;
- Zvyagin I.V., Pogorelyy M.V., Ivanova M.E., Komech E.A., Shugay M., Bolotin D.A. et al. (2014). Distinctive properties of identical twins’ TCR repertoires revealed by high-throughput sequencing. Proc. Natl. Acad. Sci. USA. 111, 5980–5985;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- In vivo — in vitro — in silico;
- Флагеллин защитит от поражения радиацией;
- Wagner U.G., Koetz K., Weyand C.M., Goronzy J.J. (1998). Perturbation of the T cell repertoire in rheumatoid arthritis. Proc. Natl. Acad. Sci. USA. 95, 14447–14452;
- Откуда пошло зрение;
- Bolotin D.A., Mamedov I.Z., Britanova O.V., Zvyagin I.V., Shagin D., Ustyugova S.V. et al. (2012). Next generation sequencing for TCR repertoire profiling: platform-specific features and correction algorithms. Eur. J. Immunol. 42, 3073–3083;
- Shugay M., Britanova O.V., Merzlyak E.M., Turchaninova M.A., Mamedov I.Z., Tuganbaev T.R. et al. (2014). Towards error-free profiling of immune repertoires. Nat. Methods. 11, 653–655;
- Turner S.J., Doherty P.C., McCluskey J., Rossjohn J. (2006). Structural determinants of T-cell receptor bias in immunity. Nat. Rev. Immunol. 6, 883–894;
- Venturi V., Kedzierska K., Price D.A., Doherty P.C., Douek D.C., Turner S.J. et al. (2006). Sharing of T cell receptors in antigen-specific responses is driven by convergent recombination. Proc. Natl. Acad. Sci. USA. 103, 18691–18696;
- Venturi V., Price D.A., Douek D.C., Davenport M.P. (2008). The molecular basis for public T-cell responses? Nat. Rev. Immunol. 8, 231–238;
- Shugay M., Bolotin D.A., Putintseva E.V., Pogorelyy M.V., Mamedov I.Z., Chudakov D.M. et al. (2013). Huge overlap of individual TCR beta repertoires. Front. Immunol. 4, 466;
- Quigley M.F., Greenaway H.Y., Venturi V., Lindsay R., Quinn K.M., Seder R.A. et al. (2010). Convergent recombination shapes the clonotypic landscape of the naive T-cell repertoire. Proc. Natl. Acad. Sci. USA. 107, 19414–19419;
- Новые успехи в предсказании пространственной структуры белков;
- Wallace M.E., Bryden M., Cose S.C., Coles R.M., Schumacher T.N., Brooks A., Carbone F.R. (2000). Junctional biases in the naive TCR repertoire control the CTL response to an immunodominant determinant of HSV-1. Immunity. 12, 547–556;
- Greenaway H.Y., Ng B., Price D.A., Douek D.C., Davenport M.P., Venturi V. (2013). NKT and MAIT invariant TCRalpha sequences can be produced efficiently by VJ gene recombination. Immunobiology. 218, 213–224;
- Lepore M., Kalinichenko A., Colone A., Paleja B., Singhal A., Tschumi A. et al. (2014). Parallel T-cell cloning and deep sequencing of human MAIT cells reveal stable oligoclonal TCRbeta repertoire. Nat. Commun. 5, 3866;
- «Омики» — эпоха большой биологии;
- Shugay M., Bagaev D.V., Turchaninova M.A., Bolotin D.A., Britanova O.V., Putintseva E.V. et al. (2015). VDJtools: unifying post-analysis of T cell receptor repertoires. PLOS Comput. Biol. 11, e1004503;
- Nazarov V.I., Pogorelyy M.V., Komech E.A., Zvyagin I.V., Bolotin D.A., Shugay M. et al. (2015). tcR: an R package for T cell receptor repertoire advanced data analysis. BMC Bioinformatics. 16, 175;
- Новое в изучении «прыгающих генов»;
- Henderson L.A., Volpi S., Frugoni F., Kim S., Janssen E., Sundel R.P. et al. (2014). A119: deep sequencing analysis of the T regulatory and T effector repertoire in juvenile idiopathic arthritis. Arthritis & Rheumatology. 66, S156;
- Föhse L., Suffner J., Suhre K., Wahl B., Lindner C., Lee C.W. et al. (2011). High TCR diversity ensures optimal function and homeostasis of Foxp3+ regulatory T cells. Eur. J. Immunol. 41, 3101–3113;
- Ye L., Goodall J.C., Zhang L., Putintseva E.V., Lam B., Jiang L. et al. (2015). TCR usage, gene expression and function of two distinct FOXP3+Treg subsets within CD4CD25 T cells identified by expression of CD39 and CD45RO. Immunol. Cell Biol. doi: 10.1038/icb.2015.90;
- Feng Y., van der Veeken J., Shugay M., Putintseva E.V., Osmanbeyoglu H.U., Dikiy S. et al. (2015). A mechanism for expansion of regulatory T cell repertoire and its role in self tolerance. Nature. 528, 132–136;
- Faham M., Zheng J., Moorhead M., Carlton V.E., Stow P., Coustan-Smith E. et al. (2012). Deep-sequencing approach for minimal residual disease detection in acute lymphoblastic leukemia. Blood. 120, 5173–5180;
- Wu D., Sherwood A., Fromm J.R., Winter S.S., Dunsmore K.P., Loh M.L. et al. (2012). High-throughput sequencing detects minimal residual disease in acute T lymphoblastic leukemia. Sci. Transl. Med. 4, 134ra163;
- Martinez-Lopez J., Lahuerta J.J., Pepin F., González M., Barrio S., Ayala R. et al. (2014). Prognostic value of deep sequencing method for minimal residual disease detection in multiple myeloma. Blood. 123, 3073–3079;
- Ladetto M., Brüggemann M., Monitillo L., Ferrero S., Pepin F., Drandi D. et al. (2014). Next-generation sequencing and real-time quantitative PCR for minimal residual disease detection in B-cell disorders. Leukemia. 28, 1299–1307;
- Logan A.C., Vashi N., Faham M., Carlton V., Kong K., Buño I. et al. (2014). Immunoglobulin and T cell receptor gene high-throughput sequencing quantifies minimal residual disease in acute lymphoblastic leukemia and predicts post-transplantation relapse and survival. Biol. Blood Marrow Transplant. 20, 1307–1313;
- Logan A.C., Gao H., Wang C., Sahaf B., Jones C.D., Marshall E.L. et al. (2011). High-throughput VDJ sequencing for quantification of minimal residual disease in chronic lymphocytic leukemia and immune reconstitution assessment. Proc. Natl. Acad. Sci. USA. 108, 21194–21199;
- Weng W.K., Armstrong R., Arai S., Desmarais C., Hoppe R., Kim Y.H. (2013). Minimal residual disease monitoring with high-throughput sequencing of T cell receptors in cutaneous T cell lymphoma. Sci. Transl. Med. 5, 214ra171;
- Logan A.C., Zhang B., Narasimhan B., Carlton V., Zheng J., Moorhead M. et al. (2013). Minimal residual disease quantification using consensus primers and high-throughput IGH sequencing predicts post-transplant relapse in chronic lymphocytic leukemia. Leukemia. 27, 1659–1665;
- van Heijst J.W., Ceberio I., Lipuma L.B., Samilo D.W., Wasilewski G.D., Gonzales A.M. et al. (2013). Quantitative assessment of T cell repertoire recovery after hematopoietic stem cell transplantation. Nat. Med. 19, 372–377;
- Farge D., Labopin M., Tyndall A., Fassas A., Mancardi G.L., Van Laar J. et al. (2010). Autologous hematopoietic stem cell transplantation for autoimmune diseases: an observational study on 12 years’ experience from the European Group for Blood and Marrow Transplantation Working Party on Autoimmune Diseases. Haematologica. 95, 284–292;
- Snowden J.A., Saccardi R., Allez M., Ardizzone S., Arnold R., Cervera R. et al. (2012). Haematopoietic SCT in severe autoimmune diseases: updated guidelines of the European Group for Blood and Marrow Transplantation. Bone Marrow Transplant. 47, 770–790;
- Muraro P.A. and Douek D.C. (2006). Renewing the T cell repertoire to arrest autoimmune aggression. Trends Immunol. 27, 61–67;
- Mamedov I.Z., Britanova O.V., Bolotin D.A., Chkalina A.V., Staroverov D.B., Zvyagin I.V. et al. (2011). Quantitative tracking of T cell clones after haematopoietic stem cell transplantation. EMBO Mol. Med. 3, 201–207;
- Britanova O.V., Bochkova A.G., Staroverov D.B., Fedorenko D.A., Bolotin D.A., Mamedov I.Z. et al. (2012). First autologous hematopoietic SCT for ankylosing spondylitis: a case report and clues to understanding the therapy. Bone Marrow Transplant. 47, 1479–1481;
- Muraro P.A., Robins H., Malhotra S., Howell M., Phippard D., Desmarais C. et al. (2014). T cell repertoire following autologous stem cell transplantation for multiple sclerosis. J. Clin. Invest. 124, 1168–1172;
- Emerson R.O., Sherwood A.M., Rieder M.J., Guenthoer J., Williamson D.W., Carlson C.S. et al. (2013). High-throughput sequencing of T-cell receptors reveals a homogeneous repertoire of tumour-infiltrating lymphocytes in ovarian cancer. J. Pathol. 231, 433–440;
- Cha E., Klinger M., Hou Y., Cummings C., Ribas A., Faham M., Fong L. (2014). Improved survival with T cell clonotype stability after anti-CTLA-4 treatment in cancer patients. Sci. Transl. Med. 6, 238ra270;
- Wooldridge L., Ekeruche-Makinde J., van den Berg H.A., Skowera A., Miles J.J., Tan M.P. et al. (2012). A single autoimmune T cell receptor recognizes more than a million different peptides. J. Biol. Chem. 287, 1168–1177;
- Birnbaum M.E., Mendoza J.L., Sethi D.K., Dong S., Glanville J., Dobbins J. et al. (2014). Deconstructing the peptide-MHC specificity of T cell recognition. Cell. 157, 1073–1087;
- Dudley M.E., Wunderlich J.R., Robbins P.F., Yang J.C., Hwu P., Schwartzentruber D.J. et al. (2002). Cancer regression and autoimmunity in patients after clonal repopulation with antitumor lymphocytes. Science. 298, 850–854;
- Linnemann C., Mezzadra R., Schumacher T.N. (2014). TCR repertoires of intratumoral T-cell subsets. Immunol. Rev. 257, 72–82;
- Gros A., Robbins P.F., Yao X., Li Y.F., Turcotte S., Tran E. et al. (2014). PD-1 identifies the patient-specific CD8(+) tumor-reactive repertoire infiltrating human tumors. J. Clin. Invest. 124, 2246–2259;
- Lam S. and Bollard C. (2013). T-cell therapies for HIV. Immunotherapy. 5, 407–414;
- CRISPR-системы: иммунизация прокариот;
- Моноклональные антитела.