
Автостопом по биоинформатике
09 февраля 2022
Автостопом по биоинформатике
- 4551
- 0
- 20
-
Авторы
-
Редакторы
Темы
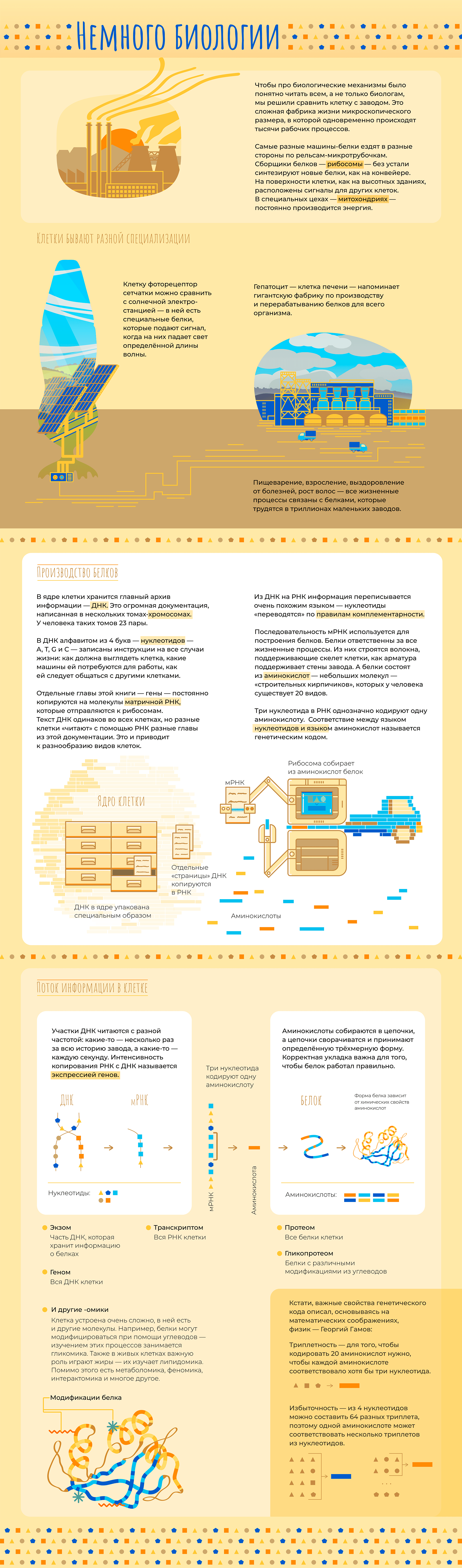
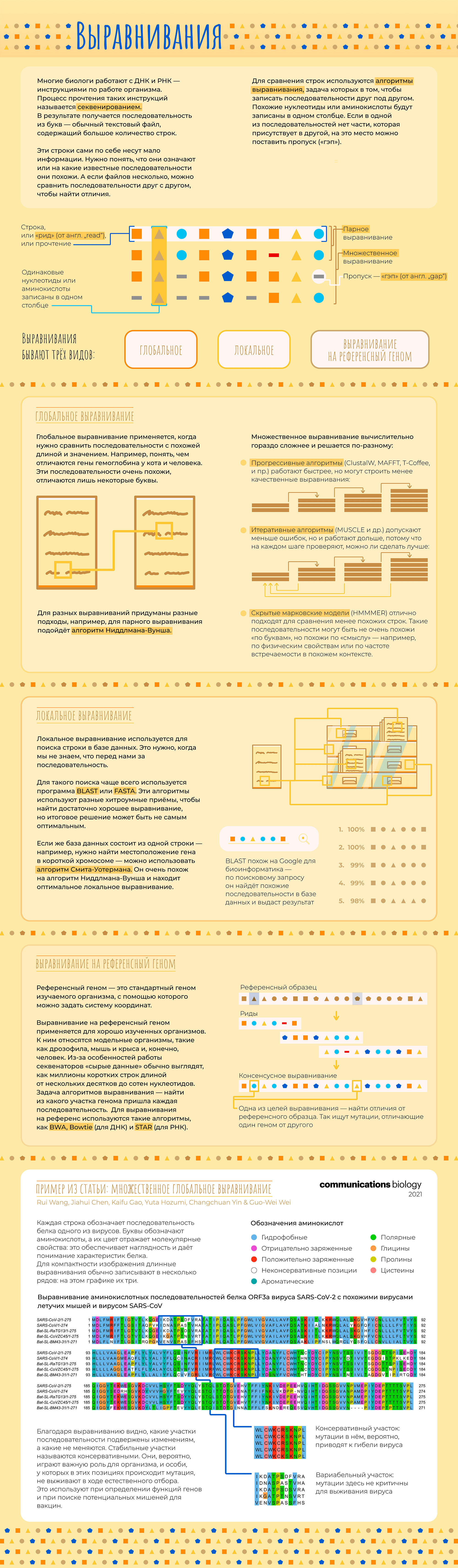
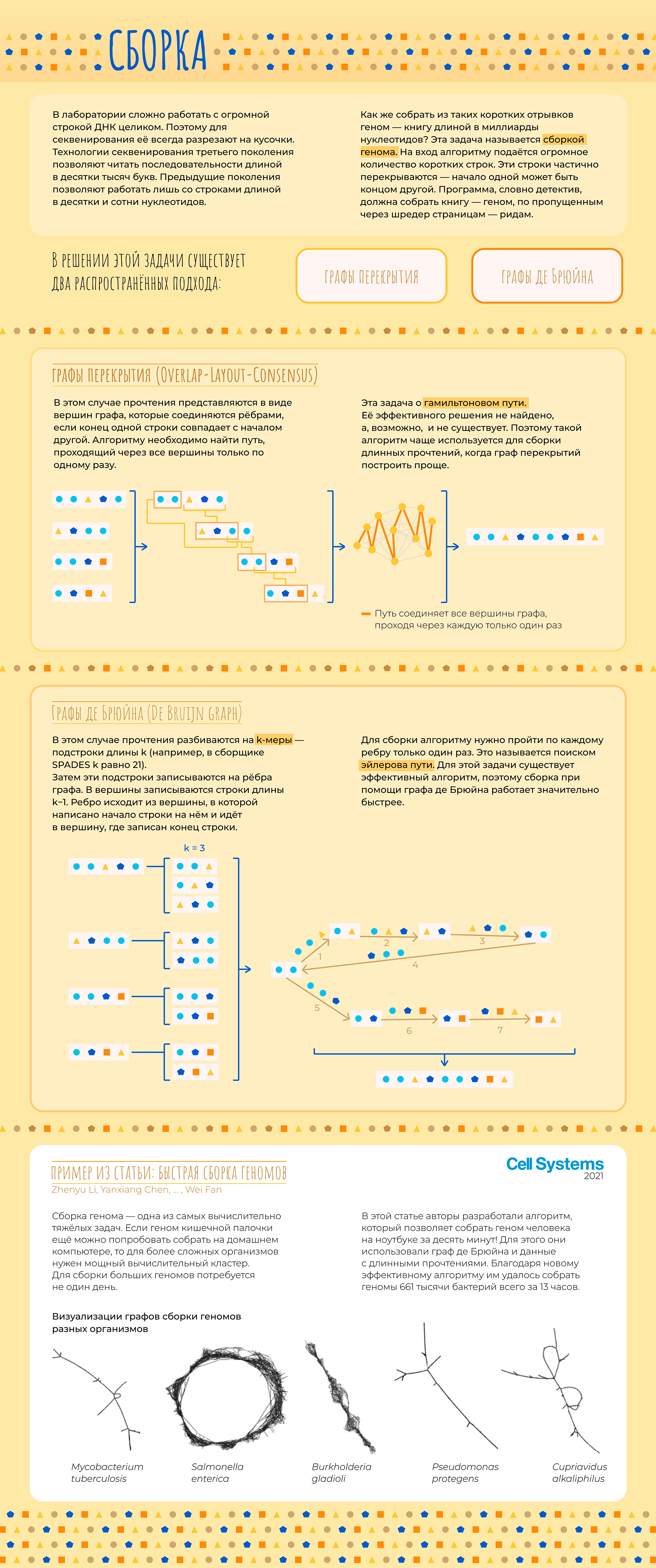
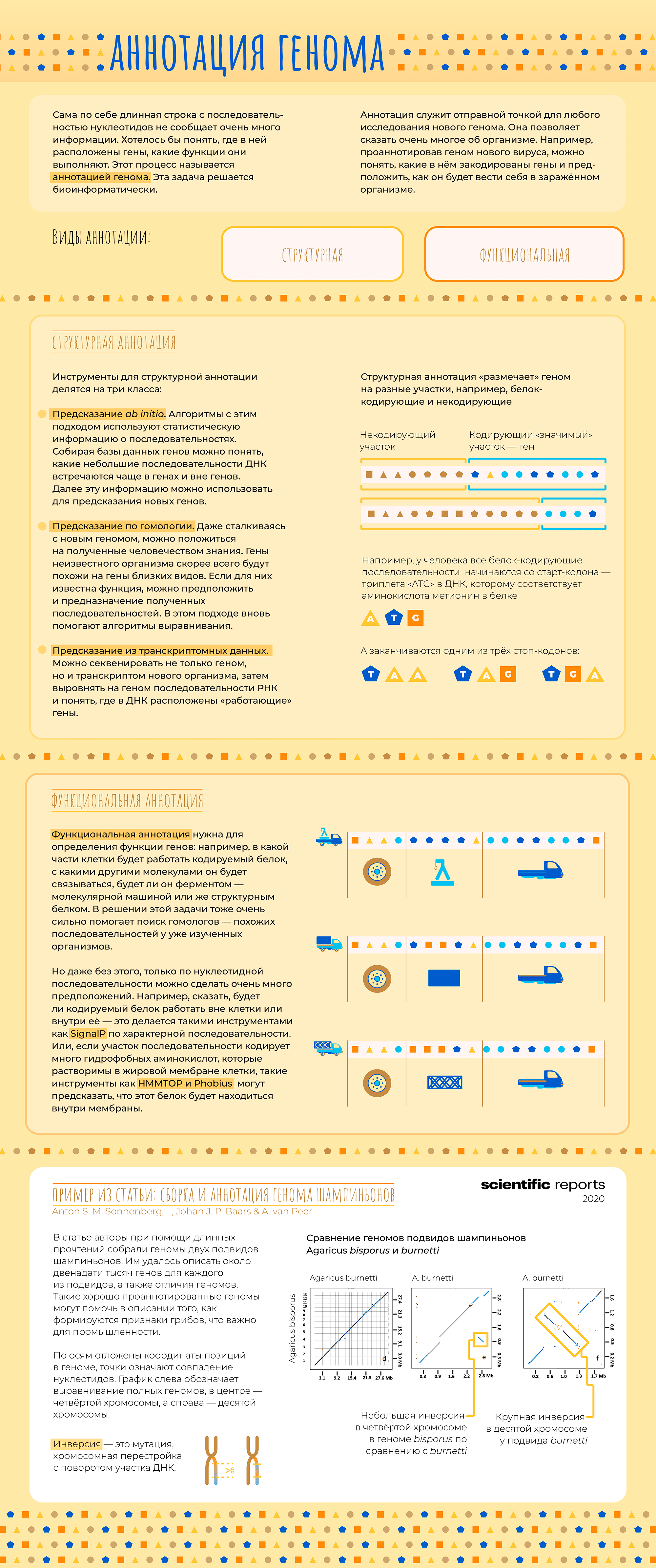
Инфографика на конкурс «Био/Мол/Текст»: Что это за звери — биоинформатики, где они обитают и как выглядит их работа? В этой статье мы расскажем и покажем, какие существуют биоинформатические методы и как их можно применять. Биологам это поможет понять, как устроены инструменты, с которыми они сталкиваются в статьях. А программисты узнают о том, как их навыки применимы к наукам о жизни.
Конкурс «Био/Мол/Текст»-2021/2022
 Эта работа заняла второе место в номинации «Наглядно о ненаглядном» конкурса «Био/Мол/Текст»-2021/2022.
Эта работа заняла второе место в номинации «Наглядно о ненаглядном» конкурса «Био/Мол/Текст»-2021/2022.

Партнер номинации — компания «Биокоммерц».

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Генеральный партнер конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
В последнее время все чаще слышно слово «биоинформатика». По этой теме открываются программы магистратуры и аспирантуры, создаются научные журналы. Но что же это за зверь такой — биоинформатик — что именно он делает и где обитает? Прояснением этого вопроса мы сегодня и займемся.
Начнем с того, что «биоинформатика» — это очень широкий термин. Под ним можно понимать применение математических методов и программирования к наукам о жизни. Этого достаточно для поверхностного понимания, но такое определение почти ничего не говорит о конкретном человеке. Все равно, как если бы человек сказал «Я — биолог». Это может значить что угодно: он может работать в поле или в лаборатории, заниматься изучением моллюсков, человека или растений.
Точно так же и биоинформатики занимаются очень разными вещами. Кто-то старается связать мутации в геноме с признаками организма и заболеваниями. Другие моделируют структуры белков на вычислительном кластере. Третьи пишут математические уравнения на бумаге или программный код на компьютере. Есть даже биоинформатики, которые сами делают эксперименты в «мокрой» лаборатории, а затем обрабатывают данные. Всех возможных вариантов не перечислить. Но мы попытаемся.
В этой статье мы расскажем об основных задачах, которые решают биоинформатики и проиллюстрируем их примерами из научных работ, в которых биоинформатика сыграла важную роль.

Скачать инфографику в формате pdf.
Литература
- Rui Wang, Jiahui Chen, Kaifu Gao, Yuta Hozumi, Changchuan Yin, Guo-Wei Wei. (2021). Analysis of SARS-CoV-2 mutations in the United States suggests presence of four substrains and novel variants. Commun Biol. 4;
- Galchenkova M. and Korzhenkov A. (2021). Modern tools for annotation of small genomes of non-model eukaryotes. arXiv.org;
- Nicolas Scalzitti, Anne Jeannin-Girardon, Pierre Collet, Olivier Poch, Julie D. Thompson. (2020). A benchmark study of ab initio gene prediction methods in diverse eukaryotic organisms. BMC Genomics. 21;
- Anton S. M. Sonnenberg, Narges Sedaghat-Telgerd, Brian Lavrijssen, Robin A. Ohm, Patrick M. Hendrickx, et. al.. (2020). Telomere-to-telomere assembled and centromere annotated genomes of the two main subspecies of the button mushroom Agaricus bisporus reveal especially polymorphic chromosome ends. Sci Rep. 10;
- Z. Li, Y. Chen, D. Mu, J. Yuan, Y. Shi, et. al.. (2012). Comparison of the two major classes of assembly algorithms: overlap-layout-consensus and de-bruijn-graph. Briefings in Functional Genomics. 11, 25-37;
- Barış Ekim, Bonnie Berger, Rayan Chikhi. (2021). Minimizer-space de Bruijn graphs: Whole-genome assembly of long reads in minutes on a personal computer. Cell Systems. 12, 958-968.e6;
- Valentine Svensson, Roser Vento-Tormo, Sarah A Teichmann. (2018). Exponential scaling of single-cell RNA-seq in the past decade. Nat Protoc. 13, 599-604;
- Richard C. V. Tyser, Elmir Mahammadov, Shota Nakanoh, Ludovic Vallier, Antonio Scialdone, Shankar Srinivas. (2021). Single-cell transcriptomic characterization of a gastrulating human embryo. Nature. 600, 285-289;
- Uirá Souto Melo, Robert Schöpflin, Rocio Acuna-Hidalgo, Martin Atta Mensah, Björn Fischer-Zirnsak, et. al.. (2020). Hi-C Identifies Complex Genomic Rearrangements and TAD-Shuffling in Developmental Diseases. The American Journal of Human Genetics. 106, 872-884;
- Stephen Curry. (2015). Structural Biology: A Century-long Journey into an Unseen World. Interdisciplinary Science Reviews. 40, 308-328;
- Rhiju Das, Ingemar André, Yang Shen, Yibing Wu, Alexander Lemak, et. al.. (2009). Simultaneous prediction of protein folding and docking at high resolution. PNAS. 106, 18978-18983;
- John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, et. al.. (2021). Highly accurate protein structure prediction with AlphaFold. Nature. 596, 583-589;
- Richard Evans, Michael O’Neill, Alexander Pritzel, Natasha Antropova, Andrew Senior, et. al. Protein complex prediction with AlphaFold-Multimer — Cold Spring Harbor Laboratory;
- The 1000 Genomes Project Consortium, Adam Auton, Gonçalo R. Abecasis, David M. Altshuler, Richard M. Durbin, et. al.. (2015). A global reference for human genetic variation. Nature. 526, 68-74;
- T.F. Smith, M.S. Waterman. (1981). Identification of common molecular subsequences. Journal of Molecular Biology. 147, 195-197;
- Maureen Rebecca Smith, Maria Trofimova, Ariane Weber, Yannick Duport, Denise Kühnert, Max von Kleist. (2021). Rapid incidence estimation from SARS-CoV-2 genomes reveals decreased case detection in Europe during summer 2020. Nat Commun. 12;
- Stephen M. Smith, Gwenaëlle Douaud, Winfield Chen, Taylor Hanayik, Fidel Alfaro-Almagro, et. al.. (2021). An expanded set of genome-wide association studies of brain imaging phenotypes in UK Biobank. Nat Neurosci. 24, 737-745;
- GenBank and WGS Statistics. (2022). NCBI;
- «Омики» — эпоха большой биологии;
- 12 методов в картинках: «сухая» биология;
- Одноклеточное секвенирование: разделяй, изучай и властвуй;
- Организовать геном: запутанная история гипотез и экспериментов;
- Ядро и эпигеном;
- AlphaFold: нейросеть для предсказания структуры белков от британских ученых;
- Биоинформатика в Сколтехе: как программисты и биологи вместе делают науку.







