
Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала
09 ноября 2016
Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала
- 10120
- 0
- 14
Старая кинопленка времен немого кино, вообще говоря, такой же носитель информации, как ДНК или РНК. Но человечество не предусмотрело для пленки места в будущем — в отличие от природы и естественного отбора, сделавших ставку на взаимопревращения ДНК и РНК — действительно хитроумный проект с неисчерпаемыми возможностями.
«Википедия», рисунок модифицирован
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Что общего у вируса иммунодефицита человека, альтернативного сплайсинга, вариабельности поверхностных белков бактерий и решения проблемы недорепликации линейных хромосом? Казалось бы, странный вопрос: перечислены довольно разнородные объекты и процессы, связанные ровно настолько, насколько все явления, присущие жизни, связаны между собой. Однако есть простой и четкий ответ — обратная транскрипция. Всё это существует и работает за счет фермента, строящего ДНК на матрице РНК — обратной транскриптазы, — а значит, имеет общее происхождение. Как же так получилось? Как всё это работает и какое отношение обратная транскрипция имеет, например, к сплайсингу? Постараемся ответить на эти вопросы, а также убедимся, что обратная транскрипция оказала и оказывает неожиданно большое влияние на эволюцию эукариотического генома.
«Био/мол/текст»-2016
Эта работа опубликована в номинации «Свободная тема» конкурса «био/мол/текст»-2016.
Генеральным спонсором конкурса, согласно нашему краудфандингу, стал предприниматель Константин Синюшин, за что ему огромный человеческий респект!
Спонсором приза зрительских симпатий выступила фирма «Атлас».
Спонсор публикации этой статьи — Анатолий Бучин.
Как известно, наследственная информация в клетке реализуется преимущественно в направлении от ДНК к РНК — за счет транскрипции, — а затем от РНК к белку — посредством трансляции. Однако ряд биологических элементов освоил обратный ток, в частности обратную транскрипцию, и рекрутировал для этого особые ферменты — обратные транскриптазы (РНК-зависимые ДНК-полимеразы, ревертазы).
Все обратные транскриптазы (ОТ), открытые на данный момент, практически со стопроцентной вероятностью имеют общее происхождение [1]. Это следует из очень высокого уровня сходства аминокислотных последовательностей этих белков. ОТ разделяют на несколько групп в зависимости от того, какому ретроэлементу «принадлежит» фермент. Под ретроэлементом здесь мы будем понимать любой молекулярный аппарат, выполняющий свои функции за счет обратной транскрипции, будь то вирус, геномный паразит или просто специфический ген. На рисунке 1 представлена филогения ретроэлементов, основанная на сравнении аминокислотных последовательностей их обратных транскриптаз. Многие найдут здесь знакомые слова вроде ретротранспозонов, а с какими-то названиями читатель, возможно, столкнется впервые.
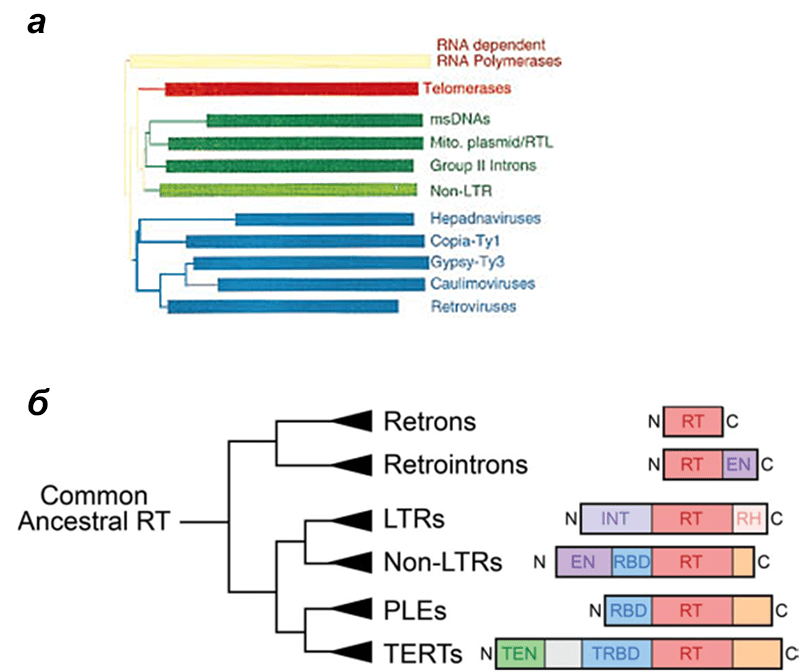
Рисунок 1. Филогенетические деревья ретроэлементов, построенные на основе последовательностей обратных транскриптаз. а — Дендрограмма, на которой видны две основные клады: одна (синяя) включает вирусы и LTR-содержащие ретротранспозоны, вторая (зеленая) включает LTR-несодержащие ретротранспозоны, ретроны (msDNAs), паразитические интроны и теломеразы. Все эти группы будут описаны ниже. Дерево укоренено с помощью метода внешней группы — в данном случае РНК-зависимых РНК-полимераз как ферментов, наиболее родственных ОТ. б — Укороченное дерево, где для укоренения использовали ретроны и интроны прокариот; остальные группы — ретроэлементы эукариот. Справа показана доменная структура, свойственная ОТ этих групп: RT — главный домен, обеспечивающий собственно ревертазную активность; EN — эндонуклеазный домен, позволяющий разрезать ДНК; INT — интегразный домен, позволяющий внедрять генетический материал ретроэлемента в геном; RH — РНКазный домен, позволяющий расщеплять РНК; RBD — РНК-связывающий домен (TRBD — его вариация у теломераз); TEN — ДНК-связывающий домен. По дереву можно судить о разнообразии обратных транскриптаз, и при чтении статьи наверняка возникнет потребность иногда бросить на него взгляд.
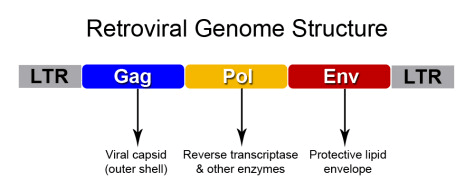
Рисунок 2. Структура генома ретровируса. Рамка считывания gag кодирует полипротеин, содержащий белок капсида; pol — ферменты, ответственные за цикл размножения вируса, а именно: интегразу, обратную транксриптазу и протеазу, разрезающую полипротеины; env — белки внешней оболочки, нужные для проникновения в клетку и выхода из нее. На концах генома есть так называемые LTR-участки — длинные повторы, необходимые для интеграции вируса в ДНК хозяина и экспрессии вирусных генов.
Хотя общий вид эволюционного древа ретроэлементов является более-менее общепринятым, детали эволюции многих их групп остаются неясными. Предлагаем рассмотреть эти группы поочередно, останавливаясь на отличительных чертах и интересных деталях каждой из них, а также на особенностях их эволюции и вкладе в эволюцию жизни в целом.
Очевидное и опасное
Обычно первое, что приходит в голову при словах «обратная транскрипция», — ретровирусы. Вкратце опишем, что они из себя представляют. У ретровирусов геном состоит из одноцепочечной РНК положительной направленности, или полярности («+» цепь). Она устроена примерно одинаково у всей группы и содержит несколько ключевых элементов — открытых рамок считывания, ORF (рис. 2).
Жизненный цикл вируса состоит из нескольких стадий. Сначала вирус связывается с рецепторами на поверхности клетки и проникает внутрь. В цитоплазме он «распаковывается», и начинается обратная транскрипция. Ее задача — сделать из одноцепочечной вирусной РНК двухцепочечную ДНК, которая затем сможет встроиться в геном хозяина.
Как и любой ДНК-полимеразе, ОТ нужен праймер (затравка) для начала полимеризации нуклеотидов. Ферменту ретровируса, например ВИЧ, праймером служит тРНК клетки-хозяина. Но она отжигается (то есть комплементарно «слипается» с матрицей, по которой пойдет синтез) не у сáмого конца, а ближе к середине вирусной РНК, и поэтому возникает проблема, как «переписать» молекулу РНК полностью. Решается эта проблема за счет хитроумного многостадийного процесса, показанного на рисунке 3.
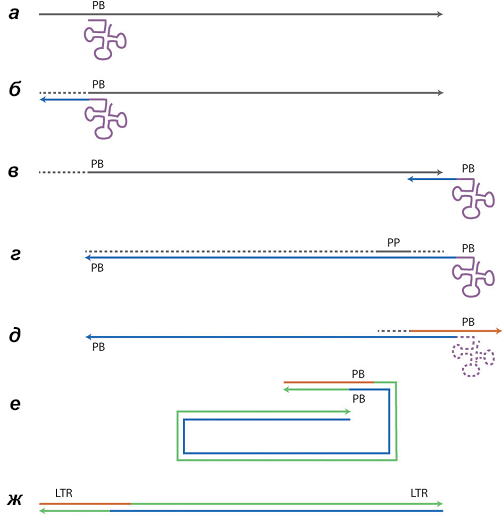
Рисунок 3. Схема обратной транскрипции ВИЧ. Участок 3’-конца тРНК (сиреневый трилистник) комплементарен области PB (от англ. primer binding site — сайт связывания с праймером) вирусного генома (а). Однако эта область находится не на конце генома, поэтому сразу синтезировать полногеномную кДНК (комплементарную ДНК) не получается. Сначала синтезируется один конец кДНК — формируется гетеродуплекс. Затем из гетеродуплекса за счет РНКазной активности обратной транскриптазы удаляется РНК (б). Синтезированная кДНК (синяя линия) отсоединяется от оставшейся РНК и отжигается на другом конце генома за счет повторов на его концах (в). Теперь ОТ может достроить цепь до конца, а РНКаза — расщепить РНК (г). Так строится первая цепь кДНК. Вторая цепь начинает синтезироваться благодаря тому, что часть РНК в середине вирусного генома (полипуриновый участок) не деградирует и может служить праймером (г, д). Построение второй цепи (зеленая линия) сильно не отличается от синтеза первой и протекает в два этапа (е, ж). Кстати говоря, именно за счет такой двухстадийной полимеризации на концах генома формируются LTR.
Геном вируса, переведенный в форму двухцепочечной ДНК, встраивается в геном клетки с помощью специального фермента интегразы, которая вносит двунитевой разрыв в ДНК хозяина и вставляет туда ДНК вируса [2].
Стоит отметить, что ретровирусы — не единственная группа вирусов, практикующая обратную транскрипцию: она встречается, например, и у гепаднавирусов, к которым относится вирус гепатита B. Однако если у ретровирусов нет выбора — их РНК в ДНК хозяина никак не встроить, — то капризные во всех отношениях гепаднавирусы просто склонны к техническим изыскам: их геном и так представлен ДНК, но реплицируется он через промежуточную РНК-стадию [3].
Ретроэлементы эукариот
Паразиту сложность ни к чему
Обратная транскриптаза есть далеко не только у вирусов. Если посмотреть на эукариотический геном, в частности человеческий, видно, что последовательности, кодирующие собственно клеточные белки, составляют ничтожно малую его долю — всего несколько процентов [4]. Что же представляет собой основная часть генома? В немалой степени — ретротранспозоны, мобильные генетические элементы, использующие обратную транскрипцию. Эти элементы занимают около 50% (!) генома человека. Ретротранспозоны делятся на два основных семейства: LTR-содержащие (от англ. long terminal repeat — длинный концевой повтор) и LTR-несодержащие. По сути, ретротранспозоны не заняты ничем, кроме создания новых копий самих себя, поэтому их устройство до крайности примитивно: они кодируют только белки, нужные для транспозиции (создания копии и вставки ее в хозяйский геном), либо не кодируют вообще ничего.
Итак, ретротранспозоны, содержащие LTR, очень похожи на ретровирусы и, несомненно, им родственны. Просто у LTR-ретротранспозонов нет рамки считывания env, и поэтому они не могут перебираться из клетки в клетку. Соответственно, цикл транспозиции представляет собой цикл вируса, запертого внутри одной клетки (рис. 4), и используется здесь та же самая, уже знакомая нам, вирусная обратная транскрипция.
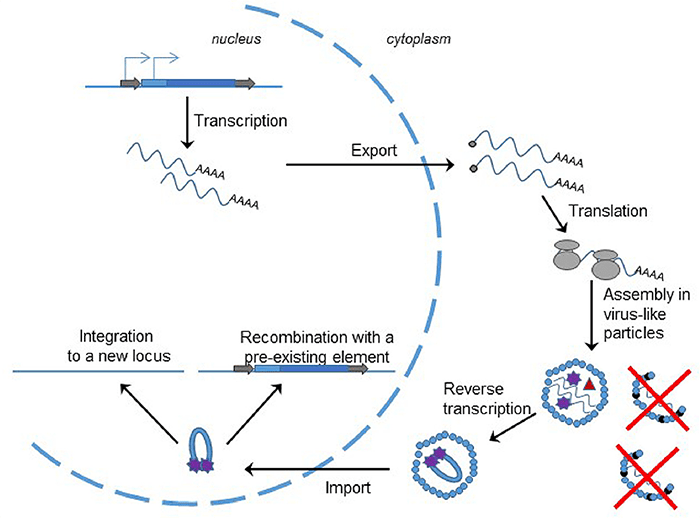
Рисунок 4. Цикл транспозиции LTR-ретротранспозона. Он практически такой же, как у ретровируса. Транскрибированная РНК выходит из ядра и транслируется. Затем формируются вирусоподобные частицы, неспособные из-за отсутствия внешней оболочки выйти из клетки. Внутри них происходит обратная транскрипция, где праймером служит та же захваченная клеточная тРНК. Затем кДНК в комплексе с интегразой проникает в ядро и либо встраивается в новый локус, либо рекомбинирует с гомологичным транспозоном.
Из-за такого сходства LTR-ретротранспозоны называют эндогенными ретровирусами. Ретротранспозоны этого типа широко распространены в эукариотических геномах. Человеческий, например, почти на 20% состоит именно из них.
Чудеса дрессировки
Вторая большая группа ретротранспозонов не содержит LTR и куда меньше похожа на ретровирусы. В этой группе выделяют два класса: LINE (long interspersed nuclear elements — длинные диспергированные ядерные элементы) и SINE (short interspersed nuclear elements — короткие диспергированные ядерные элементы).
LINE устроены так, чтобы их РНК полностью имитировала матричную РНК (мРНК) хозяина и, следовательно, без проблем транслировалась в клетке. На 5’-конце у них есть 5’-нетранслируемый участок (5’-UTR), содержащий промотор РНК-полимеразы II, которая в норме отвечает за синтез мРНК. На 3’-конце расположены 3’-нетранслируемый участок и поли(А)-последовательность, необходимая для транспорта мРНК и трансляции. Обычно поли(А)-последовательность к клеточным мРНК пристраивается после транскрипции, но у LINE она закодирована прямо в его геноме. Между 3’-UTR и 5’-UTR расположены две рамки считывания — ORF1 и ORF2. ORF2 кодирует ОТ, которая обладает и полимеразной, и эндонуклеазной активностями. Функция продукта ORF1 (ORF1р) точно не известна, однако он содержит домен взаимодействия с нуклеиновыми кислотами, «сигнал» ядерной локализации и, видимо, обладает шаперонной активностью.
После транскрипции и трансляции РНК ретротранспозона формирует комплекс с синтезированными по ней же молекулами ОТ, формируя рибонуклеопротеид (RNP). Но обратная транскрипция происходит не сразу — сначала RNP транспортируется в ядро. Там мобильному элементу нужно решить сразу две задачи: обратную транскрипцию и интеграцию (рис. 5). Геном LINE не кодирует отдельную интегразу, потому-то ОТ должна сама уметь разрезать ДНК, и обратная транскрипция у LINE сопрягается с интеграцией. Этот процесс называется target-primed reverse transcription (TPRT). Мишень транспозиции, то есть геномная ДНК, сама служит праймером для обратной транскрипции (рис. 6) — отсюда и название. Получается, что обратная транскрипция происходит чуть ли не прямо внутри генома. Такой механизм явно отличает ретротранспозоны этого типа от LTR-содержащих и ретровирусов. Более того, встраивание LTR-несодержащих элементов приводит к дупликации небольших участков мишени в месте встраивания — генерируются короткие прямые повторы, фланкирующие ретротранспозон. Однако далее мы увидим, что это еще не самый экзотический механизм интеграции ретроэлемента в геном.
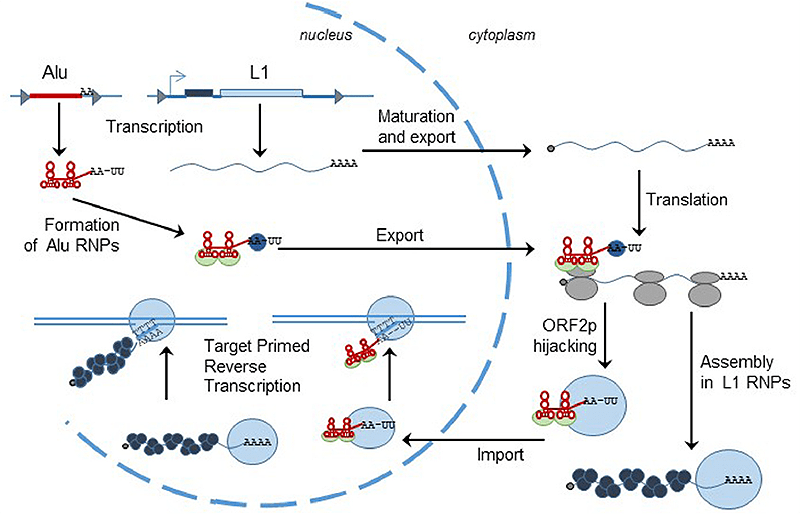
Рисунок 5. Цикл транспозиции LINE и SINE на примере L1 и Alu соответственно. Голубая волнистая линия — РНК L1 с «кэпом» и поли(А) на концах, красная — РНК Alu. Серые эллипсы на РНК L1 — рибосомы. Голубой круг — Orf2p (то есть белок, закодированный во второй рамке считывания) — обратная транскриптаза L1, а Orf2p hijacking — процесс «обобществления» элементом Alu обратной транскриптазы L1: собственной ОТ у воришки нет. Темно-серые кружочки — Orf1p.
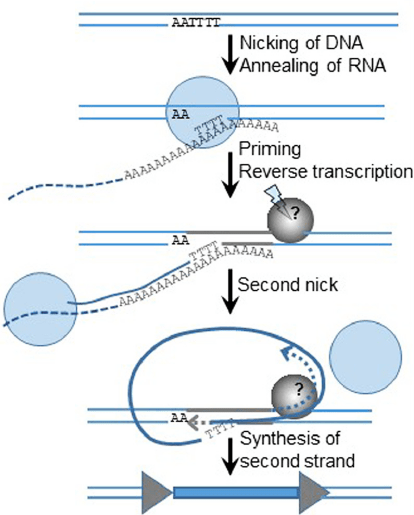
Рисунок 6. Механизм TPRT. Обратная транскриптаза (голубой круг), связанная с РНК транспозона (голубой пунктир), взаимодействует с АТ-богатым сайтом в геноме клетки, локально подплавляя ДНК, и с помощью своей эндонуклеазной активности вносит однонитевой разрыв в цепь, которая комплементарна поли(А)-участку РНК. Образуется ДНК-РНК-гетеродуплекс, и свободный 3’-конец ДНК служит праймером для ОТ, синтезирующей кДНК. Со второй хромосомной нитью пока не всё понятно: не исключено, что ее разрезает какой-то клеточный фермент (серый круг), и хозяйская ДНК-полимераза затем строит цепь по матрице первой кДНК. В результате застраивания системой репарации разрывов между хромосомной ДНК и синтезировавшимися цепями транспозонной формируются характерные короткие повторы ДНК мишени (серые треугольники).
Если все рассмотренные выше ретроэлементы кодируют собственные белки и действуют как автономные паразиты, то ретротранспозоны типа SINE являют собой апогей упрощения. По сути, SINE паразитируют на LINE. Они не кодируют никаких белков и используют для транспозиции молекулярную машинерию LINE. Соответственно, и механизм транспозиции у них такой же. Можно представить, что если один многоклеточный организм паразитирует на другом и у первого есть в геноме LINE и SINE, то выстраивается четырехуровневая система «паразит—хозяин». Такие «пирамиды» — достаточно распространенная вещь в биологии.
LINE и SINE представлены у человека семействами L1 [5], [6] и Alu [7] соответственно. Вместе они занимают около 30% человеческого генома (рис. 7)! Вообще, очень большая доля эукариотических геномов просто «набита» ретротранспозонами. В теории это может доставить клеткам ужасные неудобства, ведь если транспозон встроится в рабочий ген, то, скорее всего, он его «сломает». Таким образом, у клеток есть необходимость подавления, контроля активности ретротранспозонов, ну или хотя бы минимизации ущерба от них. Для этого клетки выработали несколько механизмов, наиболее распространенные из которых — эпигенетические: метилирование ДНК, подавляющее экспрессию прилегающих участков, и РНК-интерференция — синтез малых РНК, которые при взаимодействии с комплементарной РНК ретротранспозона запускают каскад, приводящий к разрушению этого ретроэлемента. Оба механизма очень важны и используются клеткой для регуляции экспрессии не только молекулярных паразитов, но и других генов [8], [9].
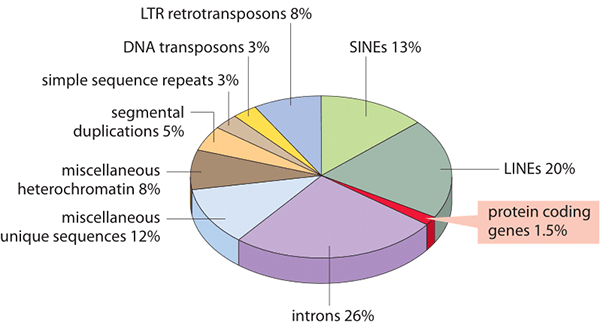
Рисунок 7. Состав человеческого генома.
сайт book.bionumbers.org
Кроме простого подавления, можно заставить транспозоны встраиваться в участки хромосом, где мало генов, — например, в «молчащие» области или концевые участки (теломеры). И так действительно происходит. Более того, похоже, что это явление эволюционировало в четкий механизм удлинения теломер, и таким образом эукариотическая клетка приобрела абсолютно «прирученную» систему обратной транскрипции. Речь идет о ферменте (а вернее, ферментативном комплексе) под названием теломераза.
Теломераза — это фермент, состоящий из нескольких белков и теломеразной РНК. Он активен в клетках простейших и дрожжей, а также в раковых и стволовых клетках многоклеточных эукариот. В большинстве соматических клеток теломераза неактивна.
По сути, теломераза представляет собой рибонуклеопротеиновый комплекс (рис. 8), основными элементами которого являются обратная транскриптаза (telomerase reverse transcriptase, TERT), теломеразная РНК (telomerase RNA, TR или TER) и несколько вспомогательных белков, важных для созревания, регуляции и локализации комплекса. Теломеразная РНК обладает сложной вторичной структурой, необходимой для связывания TERT и других белков, и содержит так называемый template — короткий участок, который служит матрицей для наращивания теломер. Рабочий цикл теломеразы состоит из четырех этапов: узнавания субстрата, элонгации, транслокации, диссоциации (рис. 9). За один такой цикл фермент добавляет один теломерный повтор [10].
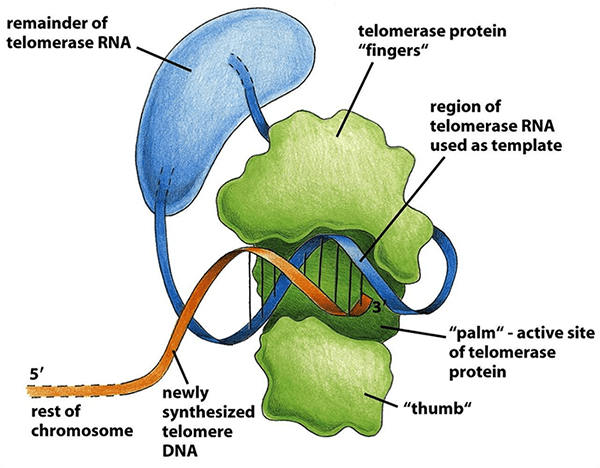
Рисунок 8. Теломераза за работой. TERT обозначена зеленым, TER — синим, геномная теломерная ДНК — оранжевым. Процесс синтеза показан на момент окончания элонгации. TERT имеет трехмерную структуру, свойственную всем полимеразам, — похожую на человеческую руку, с ладонью (palm), большим (thumb) и остальными пальцами (fingers).
Весь этот механизм функционирует для того, чтобы клетка могла продолжать делиться, ведь с каждой репликацией ДНК концы линейных хромосом укорачиваются (из-за особенностей механизма репликации). Поэтому клетки, у которых нет активного теломеразного комплекса, делятся до определенного предела, так называемого предела Хейфлика [10]. Эмбриональные и раковые клетки, синтезирующие все компоненты теломеразы, этого предела лишены, а значит, могут делиться сколько угодно.
Скорее всего, теломераза произошла от ретротранспозонов, на что наиболее красочно указывает один вполне современный факт. Теломераза есть почти у всех эукариот, но, например, у дрозофилы — классического модельного объекта биологии — ее нет. Тем не менее хромосомы у нее, разумеется, линейные, и проблема недорепликации концов остается. Так вот, вместо теломеразы для достройки теломер плодовая мушка использует LTR-несодержащие ретротранспозоны (Het-A, TAHRE и TART) [11]. Такая взаимозаменяемость весьма показательна. Кроме того, аминокислотные последовательности TERT очень похожи на последовательности обратных транскриптаз LINE, да и транскрипционный механизм теломераза применяет по сути тот же — TPRT.
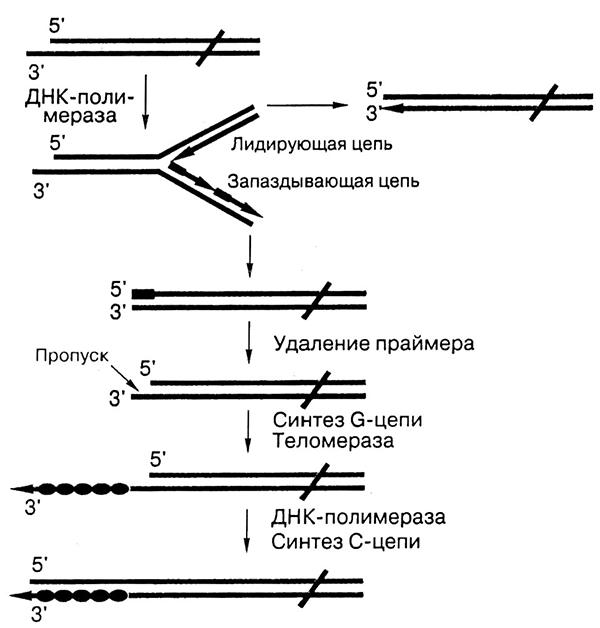
Рисунок 9. Схема решения проблемы концевой недорепликации хромосом за счет работы теломеразы. При репликации используется РНК-праймер, синтезированный специальным ферментом праймазой. После репликации он удаляется, и 5’-конец каждой из синтезированных цепей (они находятся в разных концах хромосомы) становится короче на длину праймера. Теломераза связывается посредством комплементарного участка теломеразной РНК с выступающей (G-богатой) цепью и удлиняет ее на шесть нуклеотидов по той же теломеразной РНК в качестве матрицы (все полимеразы могут начинать синтез только с 3’-конца). Затем фермент перемещается и повторяет процесс. Другая цепь достраивается классическим способом — ДНК-полимеразой, по матрице из синтезированных теломерных повторов.
сайт medznate.ru
Таким образом, происхождение теломеразы от ретротранспозонов кажется наиболее вероятным вариантом. Однако оба элемента встречаются только у эукариот. Следовательно, возникает вопрос (правда, почему-то нечасто): что же появилось раньше — ретротранспозоны или линейные хромосомы? Если сначала появились линейные хромосомы, то как удлинялись их концы? А если ретротранспозоны — то получается, что эукариотические хромосомы некоторое время были нелинейными (ведь некомпенсированная недорепликация — вещь для одноклеточных, коими были первые эукариоты, опасная: могла быть потеряна большая часть генетического материала). А может, вообще ретротранспозоны произошли от теломеразы? Но, пожалуй, лучше будет вернуться к этим вопросам чуть позже...
Несмотря на все защитные меры, принимаемые клеткой, ретротранспозоны оказывают большое влияние на геном эукариот и его эволюцию. Их влияние невозможно расценить как вредное или полезное — обычно это биологическая лотерея. Какие же геномные события связаны с деятельностью ретротранспозонов?
- Транспозиция — распространение ретротранспозонов, за счет которого гены могут тем или иным способом «ломаться».
- Рекомбинация: когда в геноме много похожих последовательностей, легко может произойти эктопическая рекомбинация, ведущая к геномным перестройкам (рис. 10б).
- Одомашнивание — превращение генов ретроэлементов в собственные гены организма (как в случае с теломеразой), хотя это больше характерно для ДНК-транспозонов, которые здесь не рассмотрены, поскольку не применяют обратную транскрипцию для транспозиции [12].
- Образование химерных генов: ОТ — очень неточный фермент, он допускает много ошибок при полимеризации, а кроме того, ОТ транспозонов имеют свойство терять собственную РНК и работать с мРНК клетки, что ведет к образованию химерных генов.
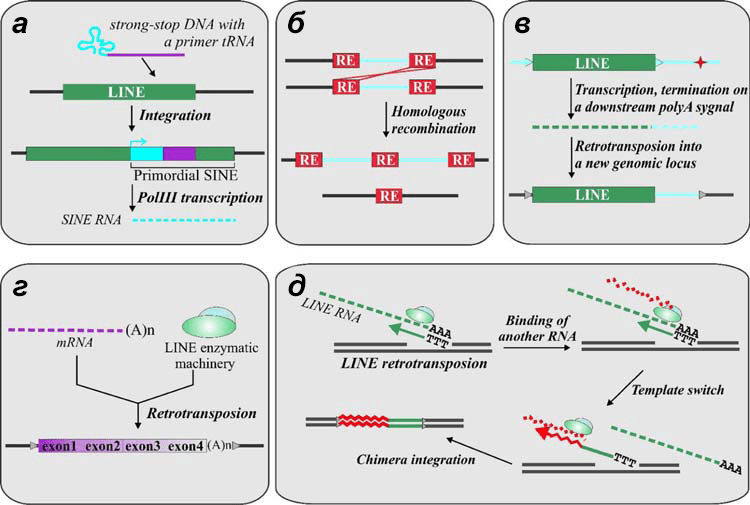
Рисунок 10. Варианты влияния ретротранспозонов на геном: а — формирование гибридного SINE за счет слияния ретровирусной РНК в комплексе с тРНК и LINE [13]; б — эктопическая гомологичная рекомбинация, приводящая к хромосомной перестройке; в — вариант мутации, когда РНК-полимераза транскрибирует помимо LINE еще и часть хромосомной ДНК (поли(А) транспозона оказывается слишком слабым стоп-сигналом для полимеразы), тогда перемещение ретротранспозона происходит вместе с соседним участком генома; г — формирование псевдогена, когда ОТ ретротранспозона «захватывает» клеточную мРНК; д — формирование химерного гена при смене РНК в процессе TPRT.
Эктопическая рекомбинация за счет присутствия ретроэлементов может быть причиной некоторых болезней, таких как гликогеноз и синдром Альпорта.
Возникновение псевдогенов или химерных генов за счет работы ОТ ретроэлементов нельзя оценить однозначно. Псевдоген — это нерабочая копия нормального гена, в которой что-то сломано. Так, например, обратная транскриптаза LINE по ошибке может вместо собственной РНК использовать в качестве матрицы мРНК какого-то хромосомного гена — спутать их несложно из-за одинаковых концевых «аксессуаров». Фермент встраивает в хромосому эту последовательность так же, как транспозонную, образуя псевдоген (рис. 10г). Более того, обратная транскриптаза LINE может сменить матрицу прямо во время TPRT, и тогда образуется химерный ген — частично транспозон, частично родной ген клетки (рис. 10д). Такие гены иногда даже оказываются рабочими и приобретают специализированную функцию.
Кстати, такая смена матрицы снова «намекает» на происхождение теломеразы от LINE-ретроэлементов, ведь теломеразная РНК закодирована в отдельном гене и совсем не похожа на РНК ретротранспозонов. Таким образом, она могла когда-то случайно «захватиться» ревертазой и приобрести новую функцию [13].
Помимо прямого вмешательства в кодирующие области генов, ретротранспозоны могут влиять и на их регуляцию. Считается, что некоторые промоторы эукариот происходят от ретротранспозонов. Известно немало примеров, когда в результате ретротранспозиции у гена возникал альтернативный промотор, который обеспечивал тканеспецифичную транскрипцию гена. За счет способности альтернативного регуляторного элемента связывать другие факторы транскрипции ген начинал экспрессироваться в той ткани, где до этого не работал, и даже приобретал там другую функцию. Ретротранспозоны также могут играть роль энхансеров транскрипции и, наоборот, избирательно «выключать» какие-то энхансеры в зависимости от типа ткани [13].
Итак, мы познакомились с двумя крупными категориями ретроэлементов — вирусными и эукариотическими. Но на этом разнообразие ретроэлементов не заканчивается...
Ретроэлементы прокариот
Существует еще несколько групп ретроэлементов, менее известных, но ничуть не менее интересных, — прокариотических.
Любопытно, что эти группы специфичны для безъядерных организмов — точно так же, как описанные выше элементы специфичны для эукариот. Так что в общеизвестный школьный список отличий эукариот от прокариот можно добавить пункт «разные ретроэлементы».
Саморазрезатели
Начнем с наиболее изученной и, видимо, наиболее эволюционно значимой группы — интронов группы II (да, название так себе, но иначе его не перевести). И да, вопреки распространенному мнению, интроны встречаются не только у эукариот, но и у бактерий и архей [14]. Интроны группы II, как и ретротранспозоны, являются мобильными элементами и имеют похожий «жизненный» цикл. Однако у них есть одна интересная особенность, радикально отличающая их от ретротранспозонов и, по всей видимости, оказавшая большое влияние на эволюцию эукариотического генома.
У ретротранспозонов в процессе всех их преобразований при транспозиции молекула РНК никак не изменяется: она не разрезается, не формирует специфических вторичных структур и т.п. А для интронов группы II как раз эти явления очень важны. Дело в том, что синтезированная с участка бактериального генома РНК интрона — это рибозим, то есть РНК, обладающая каталитической активностью, небелковый фермент. Таким образом, кроме белков в транспозиции участвует и РНК — не только как носитель информации, но и как фермент (рис. 11) [15]. Это еще одно решение механизма транспозиции: от нескольких белков у вирусов через один многофункциональный у LTR-несодержащих ретротранспозонов мы пришли к использованию каталитических свойств РНК у паразитических интронов.
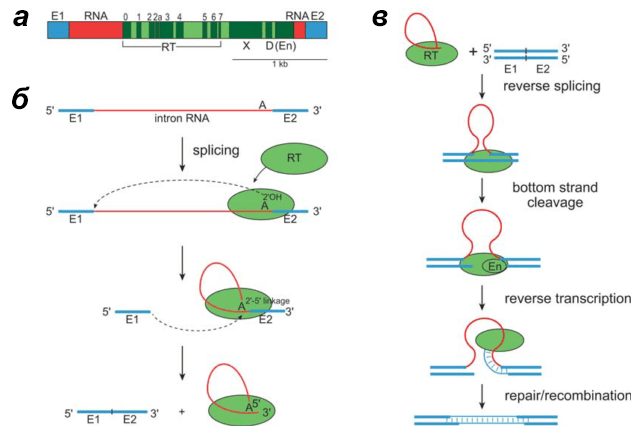
Рисунок 11. Строение и цикл транспозиции интронов группы II. а — Строение гена интрона группы II. б — Схема самосплайсинга интрона. в — Схема обратного сплайсинга интрона.
После того как рибосома синтезировала обратную транскриптазу с интронной РНК, эта РНК формирует (за счет комплементарности азотистых оснований) специфическую трехмерную структуру, в результате чего приобретает каталитическую активность. Она направлена на работу не с каким-то внешним субстратом, например ДНК, как можно было бы предположить, а с самόй РНК интрона — РНК разрезает сама себя. Отрезаются концевые участки РНК, экзоны, которые затем сшиваются между собой, и таким образом получаются два куска РНК — сшитые экзоны и сам интрон, который всё еще имеет определенную трехмерную структуру. Этот на первый взгляд жутковатый процесс саморазрезания называется самосплайсингом, или автосплайсингом, а РНК — самосплайсирующейся. Конечная структура интрона после этого дополнительно стабилизируется связывающейся с ней ОТ, синтезированной до того: это делает процесс самосплайсинга термодинамически более выгодным.
Получившаяся структура теперь способна встроиться в ДНК, причем тоже сама — за счет обратного сплайсинга. Так же, как РНК интрона отрезала себе концы, она вставляет саму себя в определенное место одной из цепей геномной ДНК бактерии. Получается, что в ДНК вставлен ничему не комплементарный кусок РНК — довольно странная ситуация. Чтобы превратить РНК в ДНК используется TPRT-механизм, как и у LTR-несодержащих ретротранспозонов. Для этого ОТ разрезает другую цепь ДНК и достраивает кусок, комплементарный вставленной РНК. Доделывают дело — избавляются от «отработанной» РНК и латают бреши — системы репарации и рекомбинации.
Интроны II группы распространены среди бактериальных геномов и геномов симбиотических органелл (митохондрий и хлоропластов). Их разделяют на несколько групп на основе различий в структуре РНК [15]. В органеллах паразитические интроны обычно присутствуют в неактивной форме. То, что такие интроны очень редко встречаются внутри ключевых генов, — признак именно паразитизма и неучастия в поддержании жизнедеятельности самόй бактерии. Иногда эти интроны могут вставляться внутрь других интронов, тогда формируется так называемый твинтрон. На основе интронов группы II даже созданы векторы для генной инженерии — таргетроны — интроны с измененной спецификой узнавания сайта обратного сплайсинга в геноме. Правда, сейчас при наличии в арсенале ученых системы CRISPR-Cas [16] этот инструмент становится довольно бесполезным.
Существует гипотеза о роли интронов группы II в эволюции, и если она верна, то эту роль сложно переоценить. Как мы уже отмечали, механизмы транспозиции интронов и LTR-несодержащих ретротранспозонов сходны между собой, как и последовательности их обратных транскриптаз, что говорит о родстве этих групп молекулярных паразитов. Но еще более интересным представляется прямое влияние этих ретроэлементов на эукариотический геном. Уже прозвучавшее слово «сплайсинг», возможно, многим знакомо. Сплайсинг очень важен для эукариот: их гены, в отличие от генов бактерий, устроены мозаично — они состоят из чередующихся интронов (участков, которые не войдут в конечную мРНК) и экзонов (формирующих конечную мРНК). Такое устройство позволяет создать дополнительную вариативность за счет комбинирования кусочков гена (альтернативный сплайсинг). У некоторых генов может быть несколько тысяч (!) сплайс-вариантов. РНК, синтезированная с большинства генов, сплайсируется специальным рибонуклеопротеидным комплексом — сплайсосомой. Это происходит по специфическому механизму, весьма схожему с механизмом автосплайсинга интронов II группы, и в сплайсосоме каталитическую функцию тоже выполняет РНК, а не белок (рис. 12). Из-за такого сходства многие считают, что интроны в геноме эукариот происходят от интронов группы II, вернее, от их предковых форм. Таким образом, мы имеем еще один эпизод одомашнивания паразитического ретроэлемента, повлекший за собой большие эволюционные последствия.
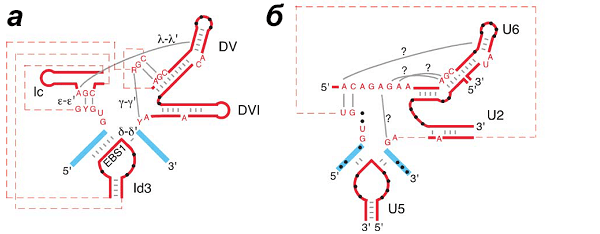
Рисунок 12. Механизмы сплайсинга, используемые интронами группы II (вверху) и эукариотической сплайсосомой (внизу). Голубым обозначены экзоны, красным — интроны.
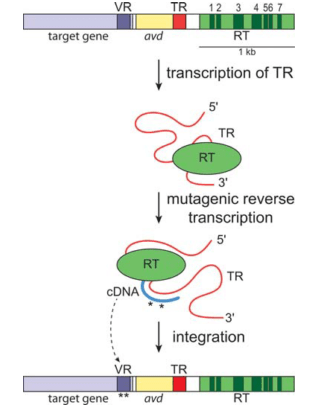
Рисунок 13. Схема кассеты DGR и мутагенного ретрохоминга. RT — ген обратной транскриптазы; VR — вариабельный участок; TR — матричный повтор; Avd (accessory variability determinant) — вспомогательный ген, кодирующий необходимый для ретрохоминга полипептид.
Ошибки — это хорошо
Во всех предыдущих разделах мы рассматривали группы ретроэлементов, по сути паразитических — не занятых ничем, кроме самокопирования (то, во что они могут эволюционировать, — это уже другой вопрос). Может сложиться впечатление, что паразитизм — главное применение обратной транскрипции (что, как мы позже увидим, не лишено смысла), но теперь рассмотрим группу ретроэлементов, которые не мешают своему владельцу, а исключительно помогают, — так называемые DGR, diversity generating retroelements (толкового русского аналога этого названия нет, а подстрочник примерно таков: «ретроэлементы, генерирующие разнообразие»).
DGR, как и интроны группы II, не встречаются у эукариот. Они не мобильны, не самокопируются, по сути это генная кассета со специфической функцией — создавать изменчивость в белках. Кассета DGR состоит из следующих элементов (рис. 13):
- гена, кодирующего обратную транскриптазу (куда ж без нее!);
- целевого гена (часто гена поверхностного белка), в котором есть вариабельный участок — тот самый, который нужно изменять;
- матричного повтора — последовательности, очень похожей на вариабельный участок целевого гена.
Как всё это работает? Система DGR использует одну уже упомянутую особенность ОТ — большое количество ошибок во время реакции полимеризации. РНК, синтезированная с матричного повтора, подвергается обратной транскрипции, при которой все аденины заменяются на случайный нуклеотид, а затем полученная кДНК заменяет вариабельный участок в целевом гене. Этот механизм называют мутагенным ретрохомингом (рис. 13). Он до сих пор до конца не изучен, но, возможно, включает TPRT, о которой говорилось выше. Так возникает новый вариант гена. Белок, им кодируемый, в одном из участков имеет другую аминокислотную последовательность, нежели до замены. В теории так можно получить огромное количество вариантов белка [17].
О существовании DGR узнали сравнительно недавно. На самом деле, они были найдены не только у бактерий, но и у бактериофагов (вирусов, поражающих бактерий). И пример, который приводит большинство авторов, — именно фаговый DGR. Речь идет о поверхностном белке фага BBP-1, поражающего бактерий рода Bordetella. Эти патогенные бактерии чередуют в своем жизненном цикле вирулентную и авирулентную фазы, которые различаются, в частности, набором поверхностных молекул. Фаг, как и любой вирус, связывается с определенной молекулой-рецептором на поверхности бактерии, чтобы проникнуть в клетку. Фаг BBP-1 для взаимодействия с рецептором использует белок Mtd (major tropism determinant). Mtd в данном случае и является целевым геном DGR. Если бы не DGR, при смене фазы жизненного цикла фаг перестал бы заражать бактерию, а так вирус изменяет строение белка Mtd, чтобы он мог связываться с другим рецептором (рис. 14). Этот феномен называется сменой тропизма. Фаг подстраивается под своего хозяина и продолжает заражать новые клетки [17].
Похоже, что и большинство других целевых белков DGR выполняет подобную функцию. В основном это поверхностные белки фагов или паразитических бактерий. И хотя их аминокислотные последовательности редко имеют что-то общее, они обычно обладают очень схожей трехмерной структурой.
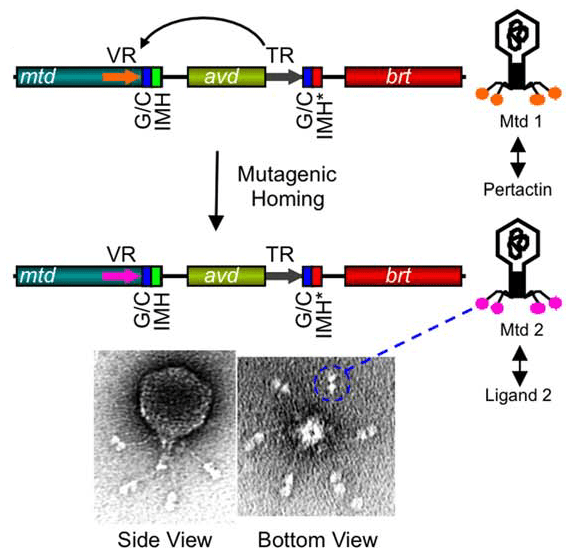
Рисунок 14. Схема изменения тропизма фага BBP-1 за счет работы DGR. С помощью белка Mtd фаг связывается с поверхностным рецептором бактерии (лигандом — по отношению к Mtd). При мутагенном ретрохоминге Mtd1 превращается в Mtd2 и начинает связывать не пертактин (pertactin), поверхностный фактор вирулентности бактерии, а другой лиганд. Brt — ген обратной транскриптазы фага. IMH (initiation mutagenic homing) вместе с GC-богатым участком (G/C) служат сигналом окончания вариабельного участка.
Пустотелые химеры
Обо всех упомянутых выше ретроэлементах известно достаточно много. В той или иной степени известно, как они устроены, как работают и зачем нужны. Но теперь мы подошли к довольно загадочной категории ретроэлементов, о которых вообще мало что можно сказать с уверенностью. Ретроны — очень любопытная группа, встречающаяся как у бактерий, так и у фагов.
Ретрон в геноме представляет собой ген, кодирующий ОТ, перед которым есть небольшая последовательность, транскрибирующаяся вместе с ним. С мРНК, естественно, синтезируется ОТ, а потом начинается удивительное (рис. 15). РНК ретрона складывается во вторичную структуру, образуя несколько шпилек. Обратная транскриптаза начинает синтезировать кДНК с РНК ретрона, праймером при этом служит 2’-OH-группа остатка гуанина. Обратная транскрипция идет не до конца, то есть кДНК синтезируется только с части РНК, причем эта часть затем расщепляется РНКазой. Другая часть РНК остается нетронутой, со всеми своими вторичными структурами. В итоге получается химерная молекула нуклеиновой кислоты (msDNA): частично РНК, частично ДНК [18].
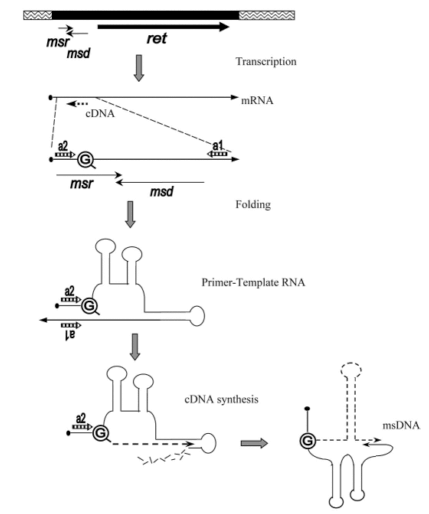
Рисунок 15. Схема формирования msDNA ретрона. Ретрон содержит локус msr—msd—ret, где названия первых двух последовательностей — аббревиатуры от msRNA и msDNA соответственно, а ret — ген ОТ. В результате фолдинга часть msr/msd-транскрипта приобретает особую вторичную структуру, которая сохранится и в составе msDNA. Происходит сближение инвертированных повторов a1 и a2 и прилегающих к ним областей, в одной из которых находится остаток гуанина (G) — праймер для обратной транскрипции. Начинается синтез кДНК (с параллельной деградацией матричной РНК), который, однако, не завершается, и полученная молекула msDNA представляет собой гибрид ДНК и РНК, связанных через «праймерный» гуанин.
Совершенно непонятно, какую функцию выполняет эта молекула. Мобильность ретронов не показана, то есть msDNA не внедряется потом в геном, за ней не замечено никакой каталитической активности или другой полезной функции.
Но наверное, msDNA всё-таки делает что-то полезное для бактерий, а иначе ретроны в ходе эволюции вычистились бы из генома. Ведь это в нашей ДНК полно «лишнего» генетического материала, а бактерии свой геном поддерживают компактным, необремененным нефункциональными вставками. Существуют разные гипотезы относительно биологического смысла ретронов, но ни одна из них даже не приближается к универсальной, а применима лишь к какой-то одной группе бактерий [18].
Заключение
Теперь становится понятно, что именно фермент обратная транскриптаза объединяет совершенно разные генетические элементы, обосновавшиеся в филогенетически далеких друг от друга хозяевах. И в конце хотелось бы еще раз вернуться к влиянию ретроэлементов на геном в глобально эволюционном смысле. Итак, паразитические интроны породили интроны геномные и сплайсинг, а ретротранспозоны породили теломеразы. Получается, что современный вид эукариотических хромосом — результат работы обратных транскриптаз. А значит, вопрос эволюции ретроэлементов тесно связан с вопросом возникновения эукариотической клетки и теорией симбиогенеза.
Вокруг темы происхождения эукариот строилось и строится множество гипотез (рис. 16). Более или менее ясно, откуда взялись митохондрии и хлоропласты: это эндосимбионты протоэукариотической клетки, современными аналогами которых являются альфа-протеобактерии и цианобактерии соответственно. Однако не до конца понятно, как линейные хромосомы возникли из кольцевых и как сформировалось ядро — из каких предшественников и до или после «органоидного» симбиоза.
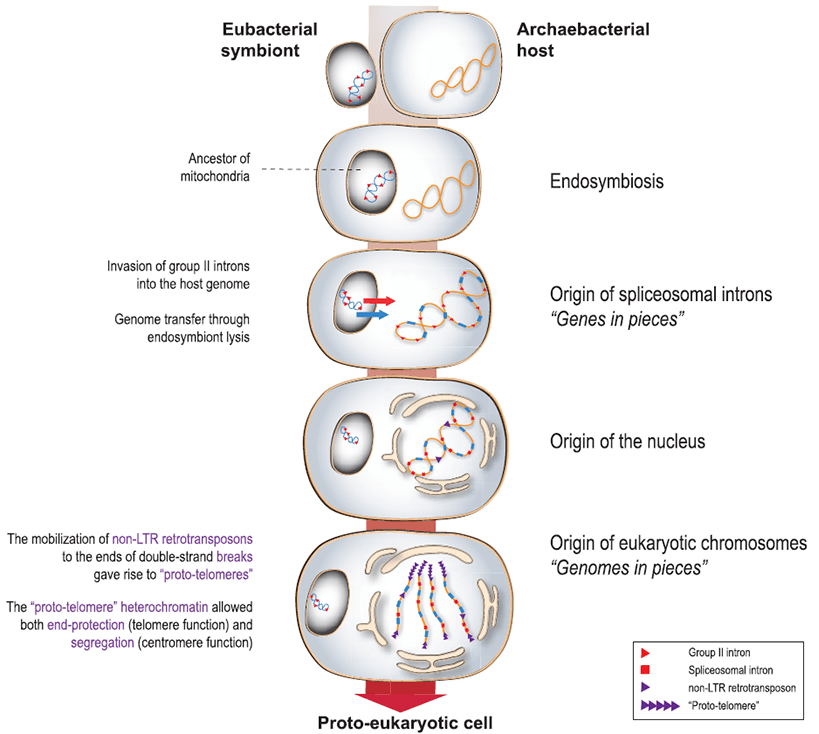
Рисунок 16. Гипотетическая схема возникновения эукариот и роли мобильных ретроэлементов в нём.
Одна из гипотез гласит, что с геномом бактериального симбионта в клетку-хозяина вторглись геномные паразиты, предшественники современных интронов группы II. Они начали активно внедряться в геном хозяина, формируя экзон-интронную структуру. В это же время из них эволюционировали LTR-несодержащие ретротранспозоны, которые приступили к встраиванию в места двухцепочечных разрывов, не давая их репарировать. А возникновение системы мембран, ставшей потом ядром, представляется мерой защиты от мобильных ретроэлементов [19].
В заключение добавим, что методами биоинформатики выявлено большое количество генов обратных транскриптаз даже в геномах бактерий, которые пока еще не охарактеризованы [20]. Исследования в этой области продолжаются. Например, не так давно обнаружили вариацию системы CRISPR-Cas с использованием обратной транскрипции [16]. Ну разве не можем мы теперь сказать, что в природе есть самая настоящая и на редкость живучая мода на ретро? А в каких формах эта мода еще может проявиться — посмотрим...
Литература
- Nakamura T.M. and Cech T.R. (1998). Reversing time: Origin of telomerase. Cell. 92 (5), 587–590;
- Saxena S.K. and Chitti S.V. Molecular biology and pathogenesis of retroviruses. In: Advances in molecular retrovirology / Ed. Saxena S.K. InTech, 2016. — 198 p.;
- Hu J. and Seeger C. (2015). Hepadnavirus genome replication and persistence. Cold Spring Harb. Perspect. Med. 5 (7), a021386;
- Сколько сора в нашей ДНК;
- Тайны «молекулярных паразитов», или Как путешествовать по геному;
- Разнообразия много не бывает: чем занимаются мобильные элементы генома в мозге;
- Alu: история одной последовательности;
- Эпигеном: параллельная реальность внутри клетки;
- Обо всех РНК на свете, больших и малых;
- Autexier C. and Lue N.F. (2006). The Structure and function of telomerase reverse transcriptase. Annu Rev. Biochem. 75, 493–517;
- Servant G. and Deininger P.L. (2016). Insertion of retrotransposons at chromosome ends : adaptive response to chromosome maintenance LTR-retrotransposons. Front. Genet. 6, 358;
- Alzohairy A.M., Gyulai G., Jansen R.K., Bahieldin A. (2013). Plasmid transposable elements domesticated and neofunctionalized by eukaryotic genomes. Plasmid. 69, 1–15;
- Gogvadze E. and Buzdin A. (2009). Retroelements and their impact on genome evolution and functioning. Cell Mol. Life Sci. 66 (23), 3727–3742;
- Belfort M., Reaban M.E., Coetzee T., Dalgaard J.Z. (1995). Prokaryotic introns and inteins: A panoply of form and function. J. Bacteriol. 177 (14), 3897–3903;
- Lambowitz A.M. and Zimmerly S. (2011). Group II Introns : mobile ribozymes that invade DNA. Cold Spring Harb. Perspect. Biol. 3 (8), a003616;
- Найдены системы CRISPR, использующие обратную транскрипцию;
- Guo H., Arambula D., Ghosh P., Miller J.F. (2014). Diversity-generating retroelements in phage and bacterial genomes. Microbiol. Spectr. 2 (6);
- Lampson B., Lampson B.C., Inouye M., Inouye S. (2005). Retrons, msDNA, and the bacterial genome. Cytogenet. Genome Res. 110 (1–4), 491–499;
- От сложного к простому: трудности симбиогенеза;
- Zimmerly S. and Wu L.I. (2015). An Unexplored diversity of reverse transcriptases in bacteria. Microbiol. Spectr. 3 (2);
- Podlevsky J.D. and Chen J.J. (2016). Evolutionary perspectives of telomerase RNA structure and function. RNA Biol. 13 (8), 720–732;
- Garavís M., González C., Villasante A. (2013). On the origin of the eukaryotic chromosome: the role of noncanonical DNA structures in telomere evolution. Genome Biol. Evol. 5 (6), 1142–1150.







