Эволюция как векторное поле: подход evo-velocity в анализе белков
15 января 2026
Эволюция как векторное поле: подход evo-velocity в анализе белков
- 162
- 0
- 3
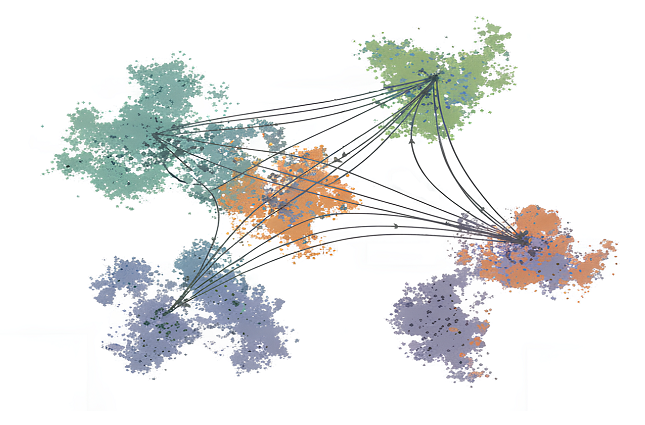
Схема направленных эволюционных траекторий белков в модельном латентном пространстве.
Рисунок в полном размере.
иллюстрация автора
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Современные белковые языковые модели позволяют по-новому взглянуть на эволюцию белков, рассматривая ее как направленное движение в латентном пространстве последовательностей. В статье разбирается подход evo-velocity, который использует вероятностные оценки языковых моделей для реконструкции направлений и относительной динамики эволюционных изменений без построения филогенетических деревьев. Показано, как метод воспроизводит известные эволюционные траектории вирусов и белковых семейств, какие предпосылки лежат в его основе и какие ограничения следует учитывать при интерпретации результатов. Подход evo-velocity иллюстрирует, что статистика белковых последовательностей содержит информацию не только о структуре и функции, но и о динамике эволюции.
Конкурс «Био/Мол/Текст»-2025/2026
Эта работа опубликована в спецноминации «Искусственный интеллект в биологии» конкурса «Био/Мол/Текст»-2025/2026.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Партнер номинации — компания Центральный университет в лице совместной с институтом AIRI Лаборатории ИИ, анализа данных и моделирования.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Эволюция белков традиционно рассматривается как исторический процесс, реконструируемый на основе родственных связей между последовательностями. Классические методы — филогенетические деревья, модели замен аминокислот и анализ консервативных позиций — позволяют восстановить вероятный порядок расхождения линий и оценить действие отбора. Однако эти подходы опираются на дискретные структуры и требуют явного задания эволюционных моделей, что ограничивает их применимость для больших и быстро эволюционирующих наборов данных.
Развитие белковых языковых моделей (protein language models, PLM, или БЯМ: читайте о них в статье «Как языковые модели покорили мир белков» [1]) показало, что статистические закономерности эволюции могут быть представлены иначе — как геометрия в непрерывном латентном пространстве последовательностей. Такие модели обучаются на миллионах естественных аминокислотных последовательностей и неявно усваивают структурные, функциональные и эволюционные ограничения. В этом представлении эволюция предстает не только как история ветвлений, но и как направленное движение в пространстве последовательностей.
БЯМ, как и «обычные» языковые модели, обучают двумя основными способами: авторегрессионное предсказание следующего токена («буквы» / «слова») — так называемый GPT-style (трансформер только с декодером); и маскированное языковое моделирование — это BERT-style (трансформер только с энкодером). — Ред.
Подход evo-velocity, предложенный Бенджамином Хи и соавторами в 2022 году [2], формализует эту идею, представляя эволюцию белков в виде векторного поля в латентном пространстве. В отличие от классической филогенетики, метод не требует множественного выравнивания или явных моделей замен и способен работать с большими массивами последовательностей. Было показано, что evo-velocity корректно воспроизводит направление и относительную динамику эволюции для различных белковых семейств, включая белки вируса гриппа A, а также согласуется с известными адаптивными траекториями ферментов и сенсорных белков (рис. 1).
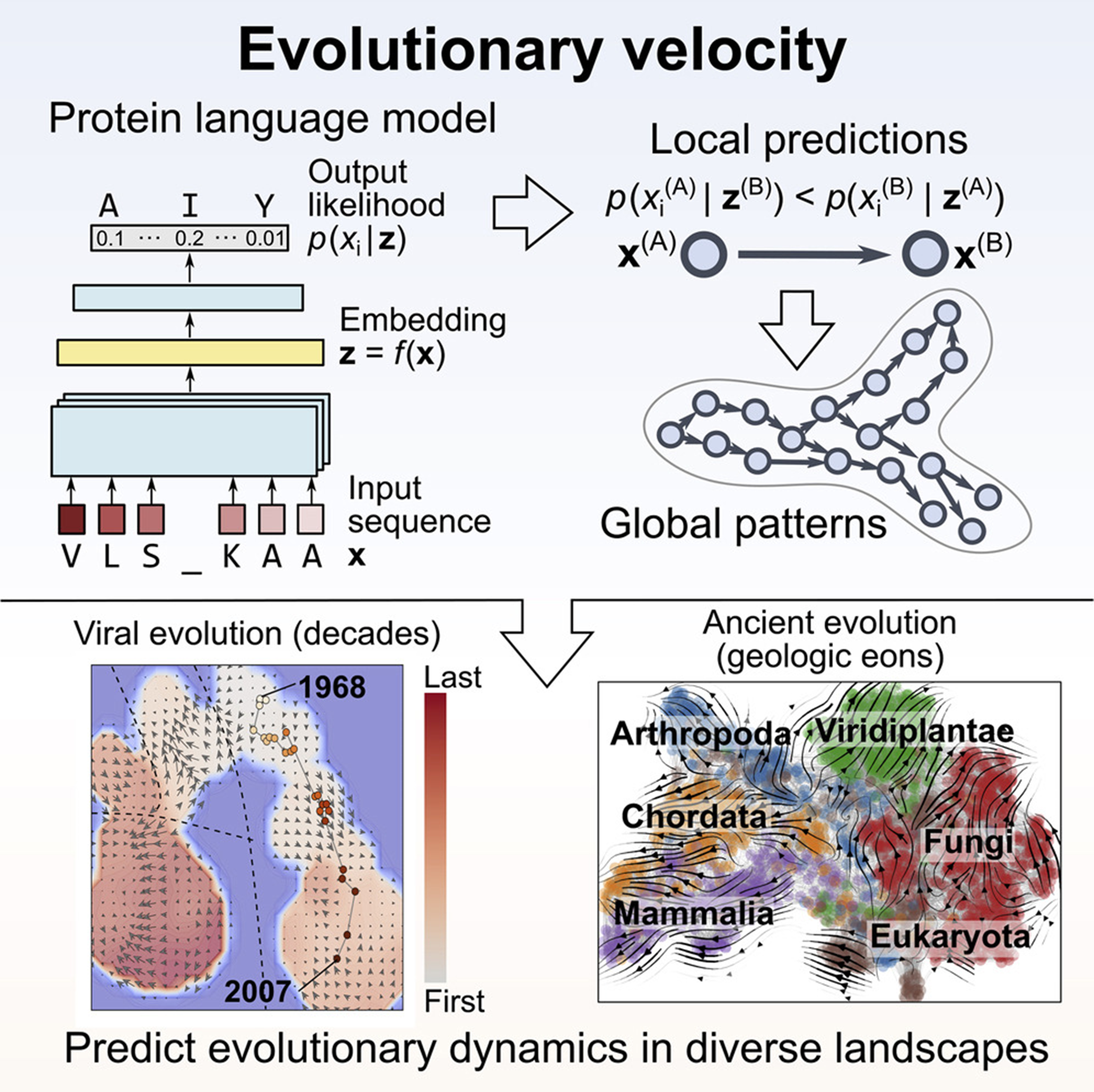
Рисунок 1. Эволюционная скорость как векторное поле в латентном пространстве белков. Вверху показано, как белковая языковая модель превращает аминокислотную последовательность в латентное представление: модель по контексту аминокислот вычисляет распределение вероятностей для каждой позиции и тем самым задает «грамматику» допустимых мутаций. Справа от схемы иллюстрируется идея evo‑velocity: если для пары близких последовательностей языковая модель считает одну более вероятной, чем другую, то между ними проводится направленное ребро, и множество таких локальных переходов образует ориентированный граф на пространстве эмбеддингов. Внизу слева показан пример быстрой эволюции вируса: на карте латентного пространства для белка гриппа A каждая точка соответствует последовательности, стрелки обозначают локальные векторы evo‑velocity, а цвет отражает порядок во времени; траектория проходит от ранних штаммов к поздним, демонстрируя, как векторное поле «подхватывает» направление реальных эволюционных переходов. Внизу справа приведен пример древней эволюции белков: в едином латентном пространстве располагаются последовательности из разных таксонов (Arthropoda, Viridiplantae, Fungi, Eukaryota и др.), а наложенное векторное поле показывает согласованное течение от предковых состояний к современному разнообразию, то есть один и тот же статистический механизм описывает как эволюцию за десятилетия, так и изменения за геологические эры.
Чтобы понять, каким образом из вероятностных оценок языковой модели извлекается направление эволюционных изменений, необходимо кратко рассмотреть принцип работы метода evo-velocity. Он основан на сравнении условных вероятностей близких последовательностей, вычисляемых языковой моделью, и использовании этих локальных сравнений для построения ориентированного графа в латентном пространстве. Агрегация таких локальных направлений позволяет задать псевдовременную шкалу и выявить согласованные эволюционные потоки внутри белкового семейства (рис. 2).
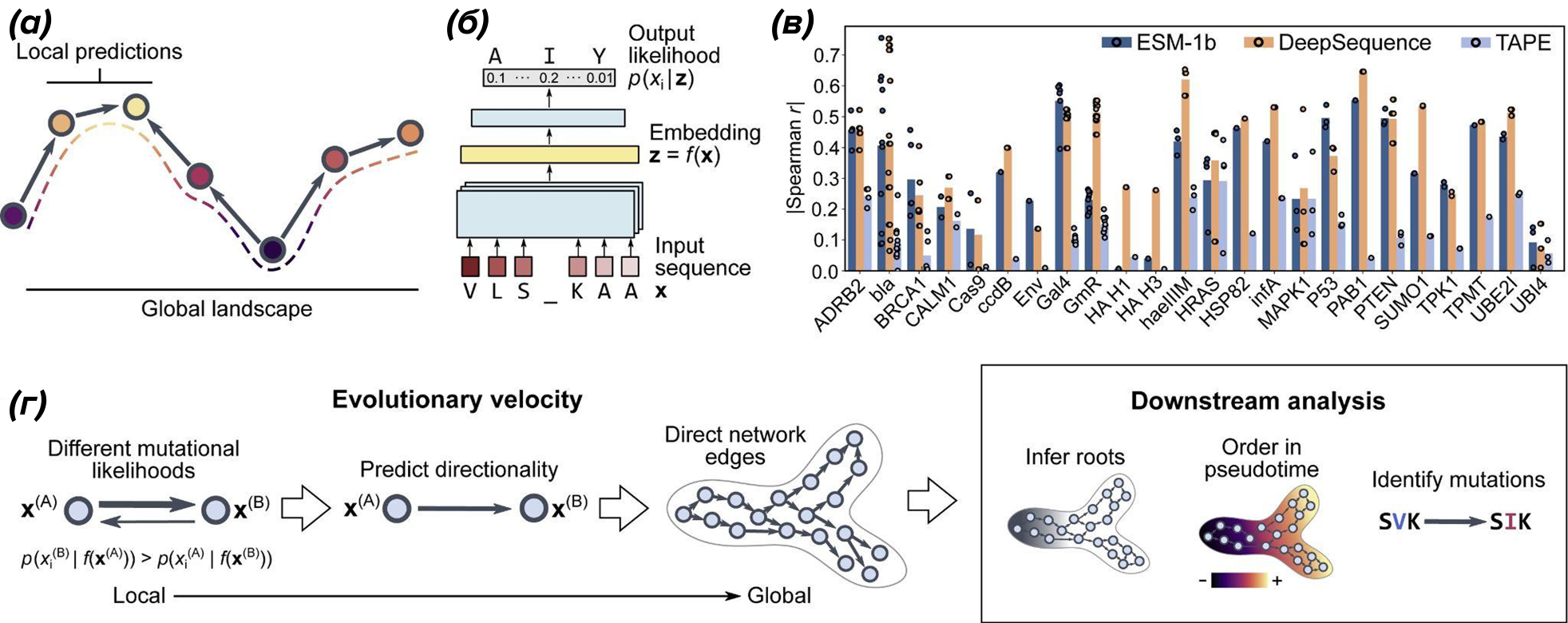
Рисунок 2. Принцип работы evo‑velocity на основе белковой языковой модели. (а) — локальные вероятностные оценки языковой модели связывают соседние последовательности и отражают их положение на глобальном эволюционном ландшафте; (б) — БЯМ кодирует входную последовательность в латентный вектор и по нему вычисляет распределение вероятностей аминокислот; (в) — сравнивая вероятности двух соседних последовательностей, можно определить направление более вероятного эволюционного перехода; (г) — на графе ближайших соседей эти локальные направления формируют ориентированные ребра, задающие глобальное векторное поле и псевдовремя эволюции.
В данной статье мы подробно разбираем предпосылки этого подхода, его методологическую интуицию, результаты применения в реальных биологических системах и ограничения, которые необходимо учитывать при интерпретации получаемых выводов.
Белковые языковые модели как источник эволюционной информации
Перед тем, как перейти к конкретным методам, полезно отметить, что редакция «Биомолекулы» уже подробно разбирала предпосылки применения искусственного интеллекта в биологии [3] и принципы работы белковых языковых моделей [1]. В частности, в обзорных материалах, посвященных истории внедрения ИИ в биологические исследования и тому, как языковые модели научились работать с белковыми последовательностями, обсуждается, почему статистический анализ больших корпусов последовательностей способен выявлять структурные, функциональные и эволюционные закономерности. Подход evo-velocity напрямую опирается именно на эти идеи и может рассматриваться как их развитие в контексте эволюционной биологии.
Белковые языковые модели обучаются на больших корпусах аминокислотных последовательностей без явной разметки. Их задача формально аналогична задаче языковых моделей в обработке естественного языка: предсказывать вероятность аминокислоты в данном контексте последовательности. При этом модель вынуждена учитывать статистические зависимости между позициями, возникающие вследствие структурных, функциональных и эволюционных ограничений.
Работы последних лет показали, что такие модели неявно усваивают информацию о третичной структуре белков, контактах между аминокислотами и функционально значимых мотивах. Важным предшественником evo-velocity стала работа Hie et al. (2021) [4], в которой было показано, что языковые модели способны различать «грамматически допустимые» мутации и мутации, приводящие к функциональным изменениям, в частности к ускользанию вирусов от иммунного ответа.
Ключевая идея здесь состоит в том, что вероятность, приписываемая моделью конкретной аминокислоте в данном контексте, может рассматриваться как прокси для ее эволюционной допустимости. Хотя эта величина не является прямой мерой приспособленности в популяционно-генетическом смысле, она отражает согласованность последовательности с распределением, сформированным естественной эволюцией.
Основная идея evo-velocity: эволюция в латентном пространстве
В основе evo-velocity лежит представление каждой белковой последовательности в виде вектора в латентном пространстве языковой модели. Такие эмбеддинги формируются на промежуточных слоях модели и отражают высокоразмерные статистические свойства последовательности.
— Кого пугает слово «эмбеддинг» — добро пожаловать в статью «Как языковые модели покорили мир белков» [1], особенно в словарик к ней. — Ред.
Далее для набора гомологичных белков строится граф ближайших соседей: каждая последовательность соединяется с несколькими наиболее близкими в латентном пространстве. Для каждой пары соседей можно оценить направление вероятного эволюционного перехода, сравнивая условные вероятности последовательностей друг относительно друга. Интуитивно, если последовательность B имеет более высокую вероятность в контексте A, чем наоборот, то переход от A к B считается более вероятным в эволюционном смысле.
Эти локальные направления агрегируются в векторное поле, определенное на латентном пространстве. Полученное поле отражает согласованное направление эволюционных изменений внутри данного белкового семейства. Важно подчеркнуть, что речь идет не о восстановлении точных временных траекторий, а о реконструкции относительных направлений и потоков эволюции.
Что означает «скорость» в evo-velocity
Термин velocity в названии метода может вводить в заблуждение, если воспринимать его буквально. В контексте evo-velocity скорость не является физической величиной и не измеряется в единицах времени. Это скалярная характеристика, отражающая согласованность направления локальных переходов в латентном пространстве.
Формально она связана с градиентом логарифма вероятности последовательностей, оцененной языковой моделью, вдоль предполагаемого эволюционного направления. На практике это позволяет упорядочить последовательности по «эволюционному времени» — относительной шкале, отражающей положение последовательности вдоль основного направления эволюционного потока.
Такое упорядочивание не заменяет датировку по молекулярным часам, но может служить альтернативным способом анализа, особенно в случаях, когда филогенетическая реконструкция затруднена или неоднозначна.
Проверка метода на реальных биологических системах
Грипп А (H1N1)
Одним из наиболее наглядных примеров применения evo-velocity стал анализ эволюции серотипа вируса гриппа А (H1N1). Используя последовательности спайк-белка, авторы показали, что векторное поле корректно отражает последовательное появление и замещение вариантов, известных как варианты, вызывающие обеспокоенность (рис. 3).
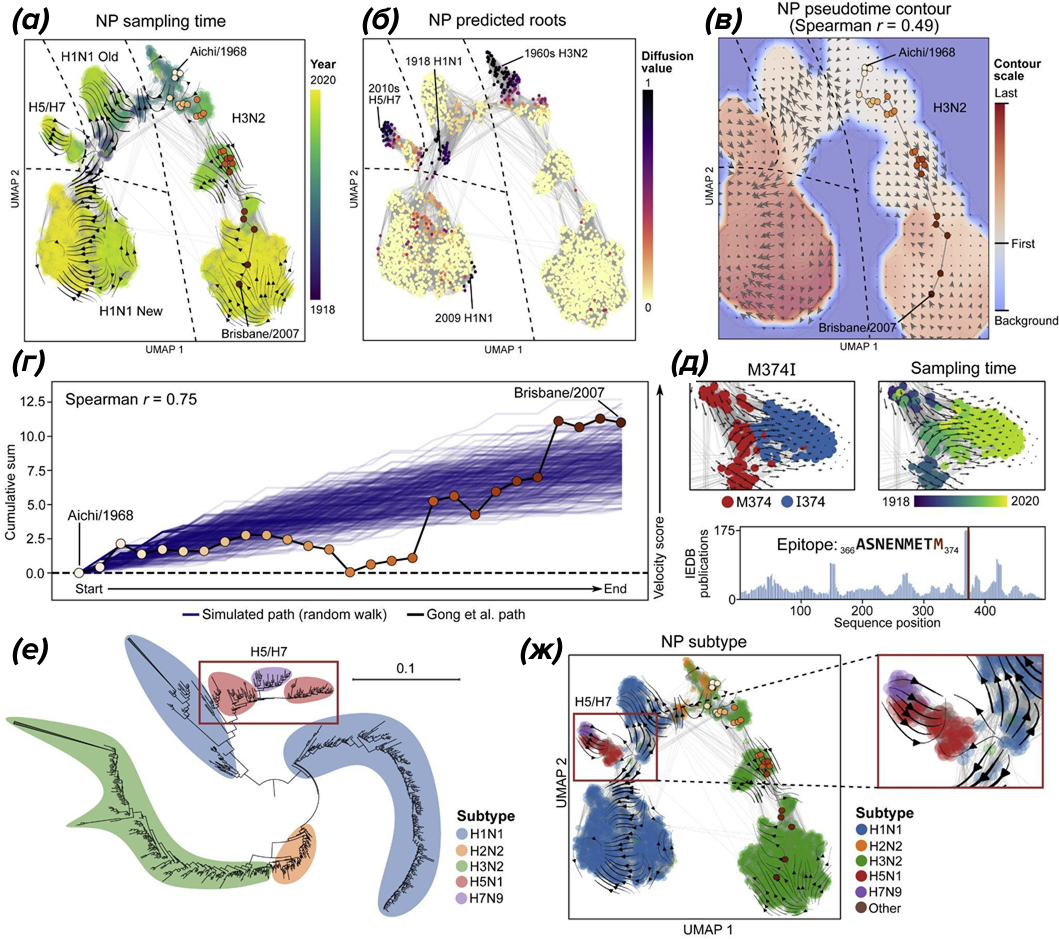
Рисунок 3. Evo‑velocity для нуклеопротеина гриппа A. (а), (б) — карта латентного пространства для нуклеопротеина гриппа A и предсказанные корни эволюции, раскрашенные по времени выделения изолятов; (в) — псевдовремя, полученное из evo‑velocity, согласуется с хронологией выборки; (г) — сравнение реальной эволюционной траектории с моделированными случайными путями показывает, что реальные переходы следуют направлению векторного поля; (е), (ж) — связь эволюционного потока с филогенетическим деревом подтипов и изменениями иммунодоминантных эпитопов.
Примечательно, что метод воспроизводит основное направление эволюции без использования временных меток или эпидемиологических данных. Это демонстрирует, что статистика последовательностей сама по себе содержит информацию о направлении эволюционных изменений.
Другие белковые семейства
Авторы также применили метод к родопсинам, ферментам и белкам, связанным с устойчивостью к антибиотикам. В этих системах evo-velocity показал согласованность с известными функциональными и структурными переходами (рис. 4), хотя степень выраженности векторного поля варьировала в зависимости от плотности выборки и эволюционной динамики семейства.
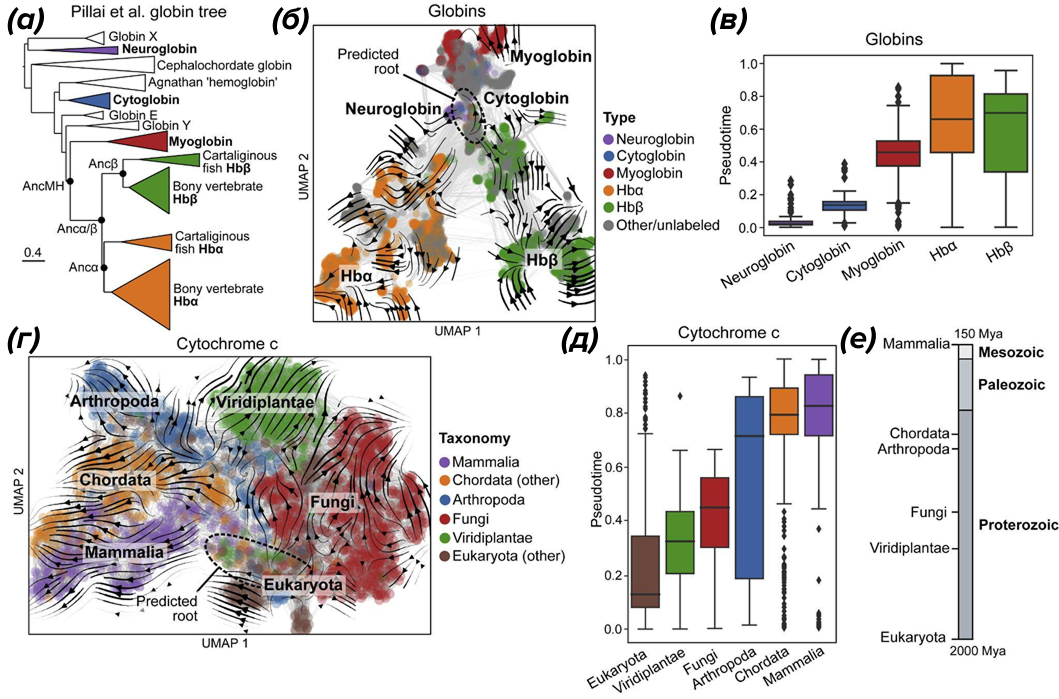
Рисунок 4. Evo‑velocity в эволюции глобинов и цитохрома c. (а) — филогенетическое дерево глобинов; (б) — расположение различных типов глобинов (нейроглобин, цитоглобин, миоглобин, гемоглобины) в латентном пространстве с наложенным векторным полем evo‑velocity и предсказанным корнем; (в) — распределение псевдовремени по типам глобинов, отражающее известные эволюционные отношения; (г), (д) — аналогичный анализ для цитохрома c: карта латентного пространства с таксономическими кластерами и псевдовременной шкалой, согласующейся с геологическим временем дивергенции основных линий.
Связь evo-velocity с другими современными подходами
Структурные языковые модели
Работа ESMFold продемонстрировала, что языковые модели способны предсказывать трехмерную структуру белков без явного использования эволюционных профилей [5]. Это подтверждает, что латентные пространства БЯМ содержат богатую информацию о биофизических ограничениях, что является важной предпосылкой корректности evo-velocity (рис. 5).
Интересно, что процитированная работа Lin et al., 2022 [5] и впрямь по большей степени посвящена алгоритму ESMFold, который, хотя и делает примерно то же самое, что и более известный биологам AlphaFold [6–8], основывается не на явно конструируемых «глубоких» выравниваниях последовательностей (MSA), как это делает AF; а на «чистой» языковой модели ESM-2, которой, между тем, в статье в Science [5] уделено совсем немного внимания, хотя по сути она стала стандартом де-факто в биоинформатике. AlphaFold, между тем, языковой моделью не является, обучаясь вовсе не на массивах последовательностей (как модели ESM), а напрямую на задаче предсказания 3D-структуры, в которой последовательности в виде MSA служат обязательными входными данными, которые и обрабатывает «эволюционный» блок AF. На практике это отличие дает интересный эффект: поскольку для ESMFold не требуется построение MSA и, следовательно, обращение к гигантским базам данных, эта программа работает намного быстрее AF2/3, где этап запроса последовательностей нельзя пропускать; за это ESMFold получил популярность в задачах in silico скрининга, где требуется «на потоке» предсказать структуру тысяч последовательностей, чтобы по их структурным метрикам решить, какая лучше. Однако, как видно из рис. 5е, качественное предсказание структур требует версии модели ESM-2 с 16 млрд параметрами (и, соответственно, внушительных вычислительных мощностей), тогда как для подавляющего множества других задач достаточно 650M-версии. Алгоритм ESMFold доступен на сервере ESM Metagenomic Atlas, где уже содержится база предсказанных 3D-моделей для >700 миллионов (!) белков. — Ред.
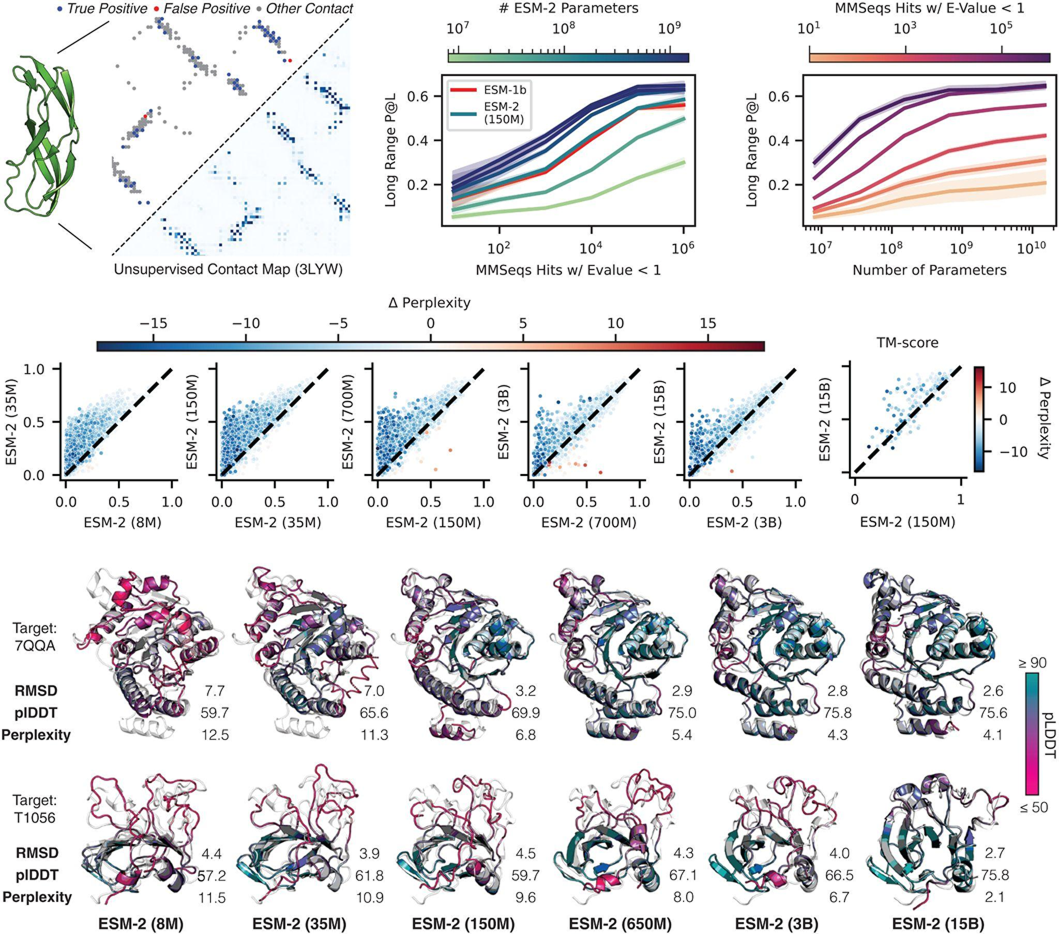
Рисунок 5. Структурная белковая языковая модель ESMFold. Обзор архитектуры модели ESMFold, которая сочетает крупномасштабную языковую модель для аминокислотных последовательностей с модулем прямого предсказания трехмерной структуры, а также примеры совпадения предсказанных атомных структур с экспериментальными. Модель демонстрирует, что латентное пространство БЯМ содержит подробную информацию о свертывании белков.
Биофизически обоснованные модели
Недавние работы, такие как METL [9], [10] (рис. 6), объединяют языковые модели с явными биофизическими параметрами — стабильностью сворачивания, энергией контактов. Эти подходы позволяют снизить риск генерации биологически невозможных последовательностей и подчеркивают, что evo-velocity следует рассматривать как статистический, а не физический метод .
В биоинформатическом сообществе давно прослеживается противостояние: ИИ или «физика». Биоинформатики «старой закалки» привыкли считать молекулярную динамику [11] и с плохо скрываемым пренебрежением относятся к «новомодным ИИ-штучкам». Молодые ML-щики же, напротив, имеют встречное когнитивное искажение: что ИИ решит все проблемы и предскажет абсолютно любые эффекты (дайте только нам побольше данных). Однако в последнее время назревает консенсус, что следующий ароморфоз в биоинформатике произойдет от вдумчивого сочетания обеих отраслей [12], [13], поскольку только так возможно скомбинировать сильные стороны и подстраховаться в слабых местах. — Ред.
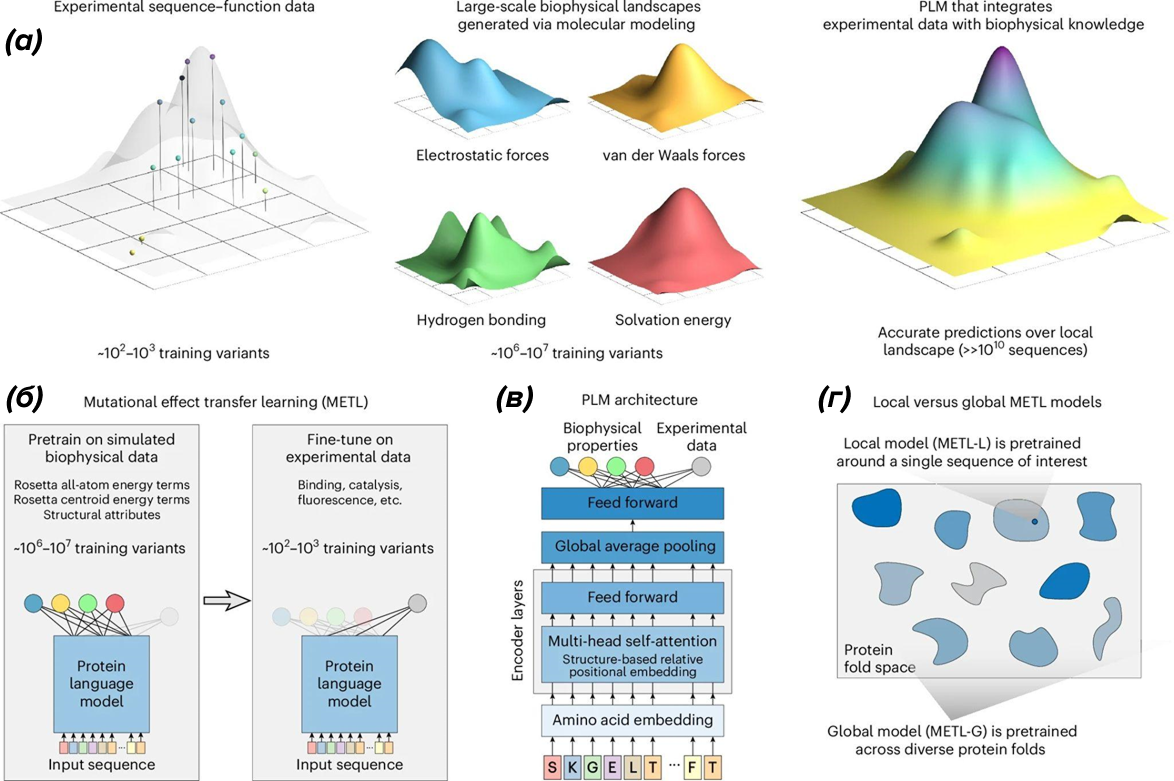
Рисунок 6. «Биофизические БЯМ» и подход METL. (а) — ограниченный по размеру экспериментальный ландшафт «последовательность—функция» объединяется с крупномасштабными биофизическими ландшафтами, полученными из молекулярного моделирования (электростатические взаимодействия, ван‑дер‑ваальсовы силы, водородные связи, энергия сольватации), что задает богатое представление последовательностей; (б) — Mutational Effect Transfer Learning (METL) — белковая языковая модель предварительно обучается на синтетических биофизических данных, а затем дообучается на небольшом экспериментальном наборе; (в) — архитектура БЯМ интегрирует признаки, полученные из биофизических и экспериментальных источников; (г) — сравнение локальных (METL‑L) и глобальных (METL‑G) моделей показывает различие между моделями, сконцентрированными вокруг одного белка, и моделями, охватывающими широкий спектр складок.
Генеративные и прогностические модели
Модели, ориентированные на генерацию или прямую оценку распространенности вариантов, такие как CoVFit [14], решают иные задачи. В отличие от них, evo-velocity не предсказывает будущие варианты, а реконструирует согласованное направление эволюции на основе уже наблюдаемых данных.
Ограничения и область применимости
Ключевое ограничение evo-velocity заключается в зависимости от качества и репрезентативности обучающей выборки. В слабо представленных или глубоко дивергировавших семействах векторное поле может быть неустойчивым. На примере серпинов и метаболических ферментов видно, как форма поля зависит от распределения таксонов (рис. 7).
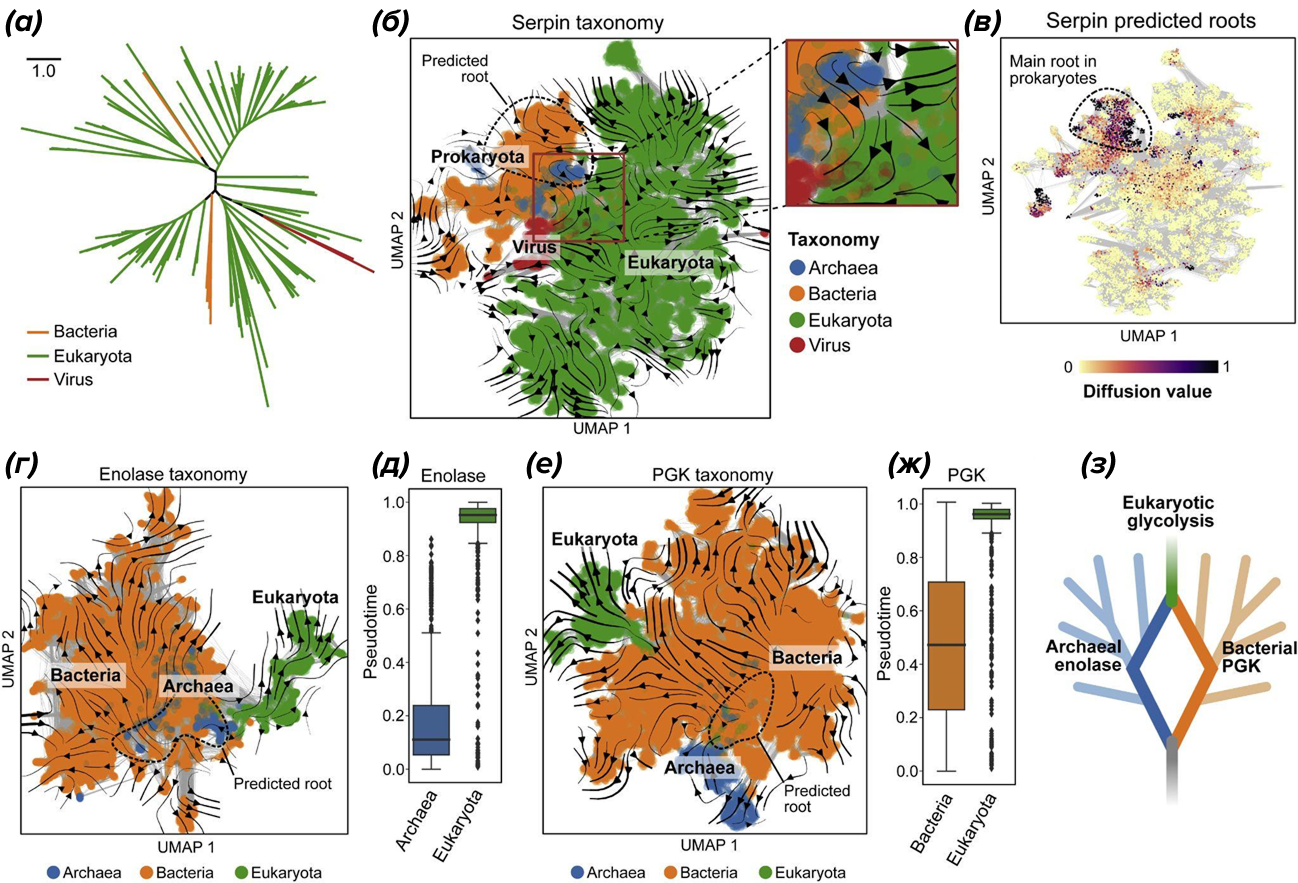
Рисунок 7. Эволюционные потоки в белковых семействах с неоднородным покрытием. (а) — филогенетическое дерево серпинов, разделяющее бактериальные, эукариотические и вирусные последовательности; (б), (в) — карта латентного пространства с векторным полем evo-velocity и предсказанными корнями для серпинов, демонстрирующая смещение источников в сторону прокариот; (г—з) — аналогичный анализ для энолазы и фосфоглицераткиназы (PGK), где различия между археями, бактериями и эукариотами подчеркивают чувствительность формы векторного поля к структуре выборки.
При этом авторы показали, что качество упорядочивания по evo‑velocity превышает простые статистические и филогенетические бейзлайны и остается устойчивым при даунсемплинге данных (рис. 8).
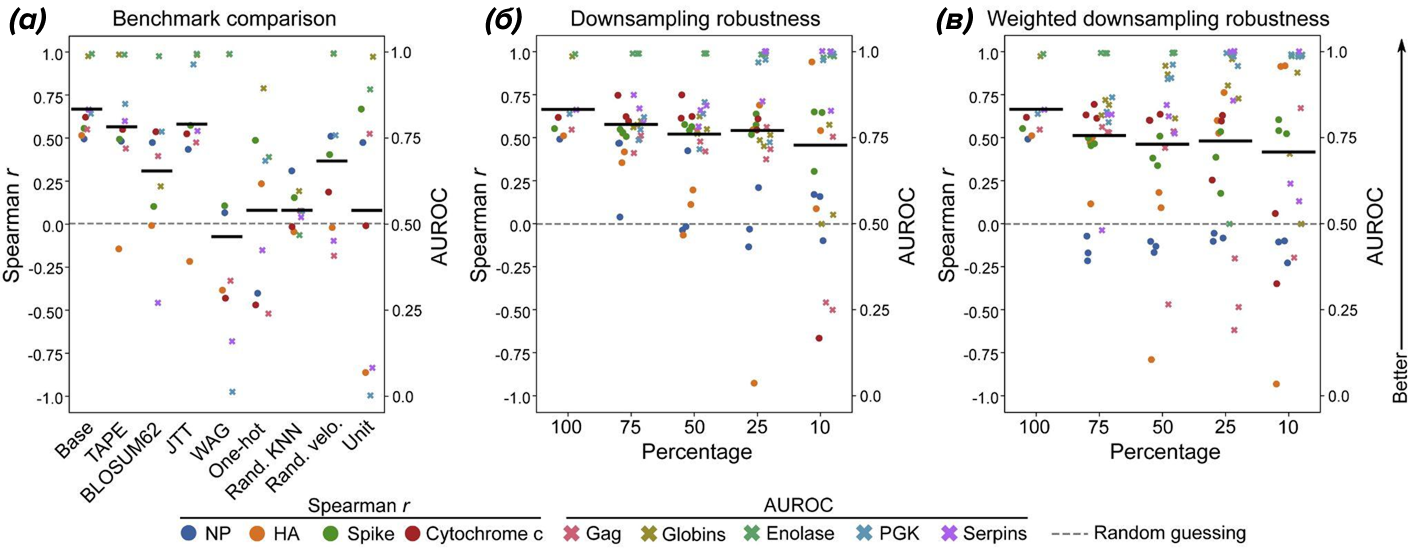
Рисунок 8. Сравнение evo‑velocity с альтернативными моделями и оценка устойчивости (а) — точность воспроизведения известных эволюционных порядков для различных белковых семейств при использовании разных представлений (evo‑velocity, филогенетические и статистические бейзлайны); (б), (в) — влияние уменьшения объема данных и взвешенного даунсемплинга на качество упорядочивания: evo‑velocity сохраняет высокие значения корреляции Спирмена и AUROC даже при сильном сокращении выборки.
Кроме того, метод не учитывает внешние факторы, такие как изменения среды или демографические эффекты, и потому не может заменить популяционно-генетические модели. Его следует рассматривать как инструмент интерпретации, дополняющий существующие подходы.
Заключение
Подход evo-velocity предлагает новый взгляд на эволюцию белков, представляя ее как направленное движение в латентном пространстве последовательностей. Он демонстрирует, что белковые языковые модели содержат не только информацию о структуре и функции, но и о динамике эволюционных изменений.
Хотя метод имеет ограничения и требует осторожной интерпретации, он открывает перспективы для анализа быстро эволюционирующих систем и для интеграции статистических и эволюционных подходов в молекулярной биологии.
Литература
- Как языковые модели покорили мир белков;
- Brian L. Hie, Kevin K. Yang, Peter S. Kim. (2022). Evolutionary velocity with protein language models predicts evolutionary dynamics of diverse proteins. Cell Systems. 13, 274-285.e6;
- История развития искусственного интеллекта и его пришествия в биологию;
- Brian Hie, Ellen D. Zhong, Bonnie Berger, Bryan Bryson. (2021). Learning the language of viral evolution and escape. Science. 371, 284-288;
- Zeming Lin, Halil Akin, Roshan Rao, Brian Hie, Zhongkai Zhu, et. al.. (2023). Evolutionary-scale prediction of atomic-level protein structure with a language model. Science. 379, 1123-1130;
- AlphaFold: нейросеть для предсказания структуры белков от британских ученых;
- Белковые галлюцинации: как справляется AlphaFold?;
- Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024);
- Sam Gelman, Bryce Johnson, Chase Freschlin, Arnav Sharma, Sameer D’Costa, et. al. Biophysics-based protein language models for protein engineering — Cold Spring Harbor Laboratory;
- Roman Bushuiev, Anton Bushuiev, Raman Samusevich, Corinna Brungs, Josef Sivic, Tomáš Pluskal. (2025). Self-supervised learning of molecular representations from millions of tandem mass spectra using DreaMS. Nat Biotechnol;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- Jonathan Martin, Marcos Lequerica Mateos, José N. Onuchic, Ivan Coluzza, Faruck Morcos. (2024). Machine learning in biological physics: From biomolecular prediction to design. Proc. Natl. Acad. Sci. U.S.A.. 121;
- Giorgio Bonollo, Gauthier Trèves, Denis Komarov, Samman Mansoor, Elisabetta Moroni, Giorgio Colombo. (2025). Advancing Molecular Simulations: Merging Physical Models, Experiments, and AI to Tackle Multiscale Complexity. J. Phys. Chem. Lett.. 16, 3606-3615;
- Jumpei Ito, Adam Strange, Wei Liu, Gustav Joas, Spyros Lytras, et. al.. (2025). A protein language model for exploring viral fitness landscapes. Nat Commun. 16.