
Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024)
10 октября 2024
Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024)
- 2295
- 1
- 7
Дэвид Бейкер, Демис Хассабис и Джон Джампер — три лауреата Нобелевской премии по химии 2024 года. Эту троицу уже видели вместе — до того они успели получить «научный Оскар» (Breakthrough Prize) в 2021 году (Бейкер) и в 2023 (Хассабис с Джампером).
Рисунок в полном размере.
коллаж автора на основе иллюстрации Нобелевского комитета
-
Автор
-
Редакторы
-
Рецензенты
Представьте, что вы можете заказать самособирающегося робота, разработанного эксклюзивно под ваши нужды. Такими естественными роботами в наших клетках служат белки. Дэвид Бэйкер, нынешний нобелевский лауреат, может проектировать эти молекулы на заказ — чтобы они служили крошечными машинами, наносенсорами или лекарствами. Два других награжденных — Демис Хассабис и Джон Джампер — создали программу, которая решает противоположную задачу. Их разработка удивительно точно предсказывает строение белков по последовательности, которой они закодированы в ДНК, — а это имеет самое непосредственное отношение к заветной проблеме биофизики: фолдингу белка. В этой статье мы разберем, как работают инструменты, за которые награждены нынешние лауреаты; а затем пофантазируем, какое будущее сулят нам их проекты.
Для начала следует озвучить несколько базовых истин, за открытие которых также были вручены Нобелевские премии — но это было еще в далеком ХХ веке. Белок состоит из цепочки аминокислотных остатков с разными химическими свойствами. И эти «бусы» сворачиваются в трехмерную структуру главным образом из-за взаимодействий между отдельными «бусинами»-звеньями. Почитать об этом подробнее можно в статьях «Роль слабых взаимодействий в биополимерах» [1] и «Проблема фолдинга белка» [2].
Структура белка определяет его функции, поэтому так важно уметь ее прогнозировать. Каждая полипептидная цепь складывается в уникальную для нее структуру — это открыл Нобелевский лауреат 1972 года Кристиан Анфинсен, — причем, как правило, такая четко определенная структура только одна (хотя куда в биологии без исключений: «Одна последовательность — одна структура: был ли Анфинсен неправ?»). Получается, что сама возможность такого предсказания была осознана уже давно, но вот реальный путь оказался долгим. Еще недавно практически единственным вариантом такого предсказания было моделирование по гомологии (за подробностями отсылаем к статье «Торжество компьютерных методов: предсказание строения белков»), но сейчас лидерство, безусловно, принадлежит нейросетевым подходам.
Однако давайте разберемся со всем по порядку.
Дэвид Бэйкер: дизайн белков под новые функции
Группа американского биохимика Дэвида Бэйкера заинтересовалась проблемой фолдинга белка еще в 1990-х, и ее решение они увидели в разработке компьютерной программы, получившей название Rosetta. Опираясь на аналогию с артефактом древности — Розеттским камнем, содержащим один и тот же текст на трех мертвых языках и позволившим, сопоставляя фрагменты, расшифровать два доселе неизвестных языка, — Розетта была задумана как гибрид физического и эволюционного подхода к решению проблемы фолдинга. Делая запрос к базе данных, эта программа выстраивает предположения, какие элементы вторичной структуры будут составлять моделируемый белок; а пространственная укладка этих элементов моделируется путем перебора конформаций в специальном силовом поле . Конечно, это требовало огромных вычислительных ресурсов — некоторые задачи были решены с помощью распределенной сети, включавшей до 70 000 компьютеров: «Новые успехи в предсказании пространственной структуры белков» [5]. Но оказалось, что предсказание структуры — это не самое интересное: неожиданной перспективой стала возможность дизайна новых белковых молекул, никогда не существовавших в природе.
Термин «силовое поле» может звучать непонятно для непосвященных в компьютерную биофизику. Если вам кажется, что это что-то из киберпанка — просим ознакомиться с нашими публикациями: «Молекулярная динамика биомолекул. Часть I. История полувековой давности» [6] и «12 методов в картинках: „сухая“ биология» [7].
В 2003 году команда Дэвида Бэйкера опубликовала дизайн небольшого белка Top7 из 93 аминокислот [8]. Это был совершенно новый белок, не похожий ни на что известное ранее: ни его последовательность, ни структура не содержались ни в одной базе. Мало того: пространственное строение, определенное с помощью рентгеноструктурного анализа, полностью совпало с предсказанным (рис. 1) — такого не делал еще никто.
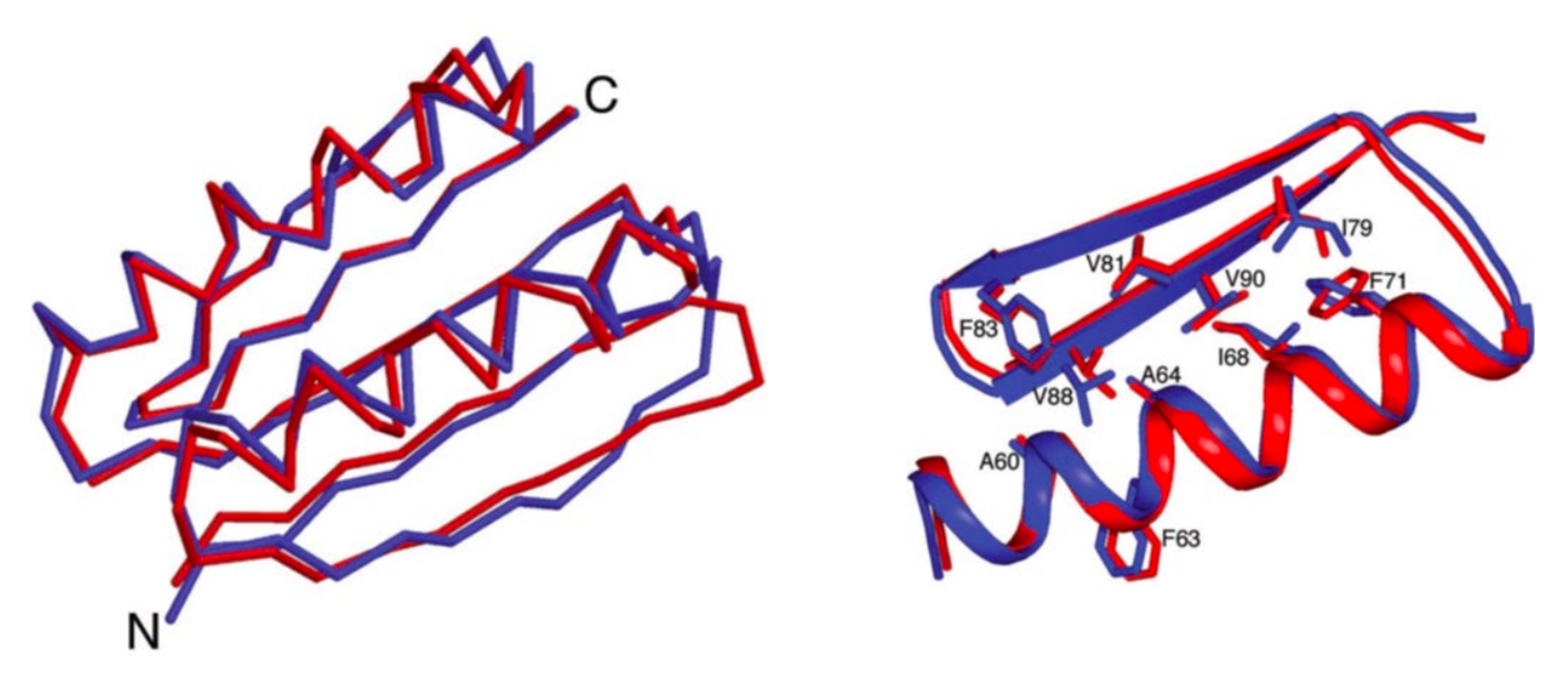
Рисунок 1. «Дизайнерский» полипептид Top7 — один из первых примеров белкового дизайна. Сейчас такими картинками уже никого не удивишь, но, наверное, это был один из первых примеров совмещения структуры, сгенерированной и определенной экспериментально, чтобы они практически полностью совпали.
Слева: сопоставление общей укладки.
Справа: увеличенный фрагмент с деталями устройства.
Интересно, что креативному Бейкеру было мало чисто научных задач — при его участии разработали компьютерную игру Fold It. В ней игрокам предлагается заняться фолдингом белков в формате головоломки, не требуя для этого научной степени и даже окончания вуза. Что интересно, игроки даже стали соавторами научных публикаций! Мы рассказывали об этом замечательном примере гражданской науки в материале «Тетрис XXI века» [10].
За последующие годы команда Бэйкера значительно усовершенствовала программу. Главным трендом стало — чтобы белок не просто был новым, а выполнял какую-то важную функцию: «Дизайнерские ферменты на службе общества» [11]. Розетту научили генерировать ферменты и белки, которые связывают определенные лиганды лучше, чем известные аналоги в природе. Так, в 2017 команда выпустила дизайн белка для быстрого и специфического анализа на фентанил — опасный наркотик. А в 2021 они спроектировали частицы, по форме и свойствам напоминающие вирус гриппа. Они могут применяться для безопасной вакцинации и уже проверены на животных.
Новые метаболические пути фиксации углерода, лекарство от целиакии, «клей» для вирусов гриппа и SARS-CoV2 — вот далеко неполный список работ Института белкового дизайна, который возглавляет Бейкер. За прочими увлекательными подробностями отправим читателя к статье «Конструкторское бюро белков» [12] и к одной научной лекции (видео 1).
Видео 1. Хотите проникнуться идеей белкового дизайна по-настоящему? Смотрите лекцию редактора этой статьи! В лекции, прочитанной для студентов магистратуры «Вычислительная физика конденсированного состояния и живых систем» МФТИ, рассказывается про научную базу и применения белкового дизайна — существующие и перспективные. Кроме Розетты, основанной на физическом моделировании молекул, в лекции много говорится про нейросетевой дизайн белков, который очень роднит лауреатов Нобелевской премии по химии 2024 года.
лекция «Белковый дизайн — технология будущего», прочитанная в МФТИ 17.05.2023
Демис Хассабис и Джон Джампер: AlphaFold — собираем белковую головоломку
Пока Дэвид Бейкер и его команда развивали физические подходы к моделированию и дизайну белков, в компании DeepMind, ставшей недавно научным подразделением ИТ-гиганта Гугл Google DeepMind, решили зайти с другой стороны. Бывший шахматный гений, переквалифицировавшийся в нейробиолога, а затем — специалиста по искусственному интеллекту Демис Хассабис, а также его главный сотрудник Джон Джампер чувствовали, что к предсказанию 3D-структуры белков можно подступиться иначе. Они подключили к делу технологии глубокого обучения, которые незадолго до этого опробовали в нейросети AlphaGo [13], прославившейся тем, что отобрала у человека лидерство в когда-то казавшейся неподвластной компьютерам игре го.
Искусственный интеллект в последние годы обитает буквально в каждом утюге, не обошел стороной он и биологию, о чем мы рассказываем в особом спецпроекте: «История развития искусственного интеллекта и его пришествия в биологию» [14]. Помимо предсказания структуры белков, очень яркие и неожиданные возможности открывают, например, большие белковые модели: «Как языковые модели покорили мир белков» [15]. Вездесущность ИИ можно подтвердить хотя бы тем, что Нобелевскую премию 2024 года по физике (!) тоже выдали за искусственный интеллект — а точнее, его физические основы [16]. В интернетах шутят, что Нобелевка по литературе тоже должна быть выдана за ИИ — за «неизгладимый и важнейший вклад» ЧатаГПТ в развитии литературы и поэзии :-)
Собственно, пасуя перед лицом невероятной сложности решения проблемы фолдинга белка «в лоб», биохимики давно уже заметили, что структуры белковых молекул можно предсказывать, «срисовывая» их с родственных молекул — это называется сопоставительным моделированием или моделированием на основе гомологии [4]. Но серьезных отличий от шаблона такой метод предсказать не может — тут требовалось что-то иное.
Новый алгоритм AlphaFold 2 (прорывной считают вторую версию [17], хотя была и первая [18]) основывался на искусственной нейросети глубокого обучения [19], [20]. Она способна выявлять закономерности в огромных массивах белковых последовательностей, которые указывают на пространственную близость одних фрагментов последовательностей белка к другим.
Причина успеха AlphaFold 2 — большие данные для обучения глубокой нейросети и гигантские требуемые вычислительные мощности. В DeepMind поняли, что по-настоящему большие данные надо искать в метагеномике — «сборнике» всех генетических последовательностей, которые поколения ученых секвенировали [21] в течение десятков лет. Они включали в себя [20] даже геномы организмов, которых никто никогда не видел, но генетический материал которых выловили где-то в океане или выкопали в почве. В них «отпечатались» сотни миллионов лет эволюции, закономерности которой AlphaFold 2 было необходимо просчитать. Идею использовать метагеномику испытывали и раньше [22], но именно DeepMind удалось загрузить в нейросеть все 2,5 миллиарда последовательностей и оптимальным образом натренировать программу предсказывать все существующие на текущий момент 3D-структуры. Схематично работа AlphaFold показана на рисунках 2–4, а кому этого не хватит — приглашаем почитать статью «Как языковые модели покорили мир белков» [15] (там есть раздел про AlphaFold и другие «предсказатели») и посмотреть еще одну лекцию (видео 2). Больше прикладных вопросов (например, о слабых сторонах AlphaFold 2 и 3) обсудили на Guest Club фармацевтической компании BIOCAD в октябре 2024 г. (видео 3).
Мы рассказывали о (мета)геномике в статье «„Омики“ — эпоха большой биологии» [23]. Прочитать о новых трендах в этой области советуем в спецпроекте Мультиомиксные технологии: например, в статье «Факты и легенды о мультиомиксных технологиях» [24] рассказывается о том, как и зачем объединяют геномные данные с большими данными о белках, ДНК и РНК; а как работают экологи и эволюционные биологи — в статьях «Мультиомика и Экологические тайны третьей планеты» [25] и «Как мультиомиксы раздвигают границы современной биологии» [26].
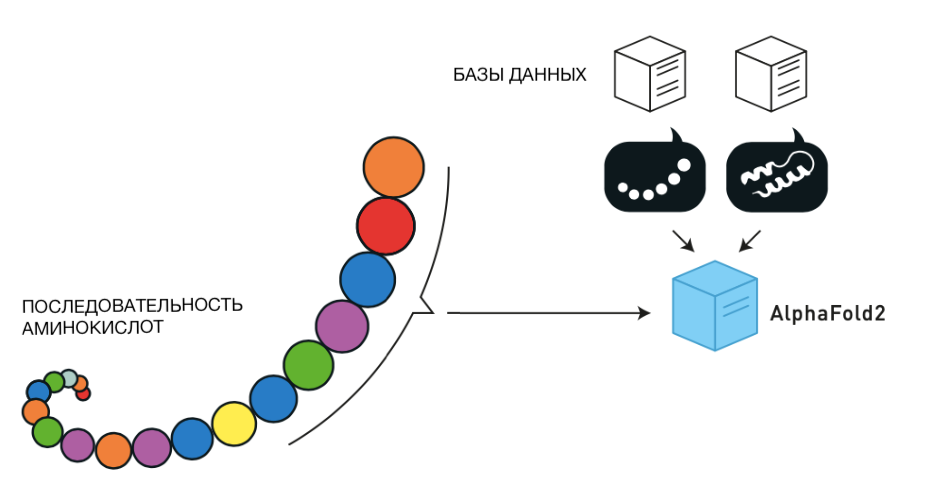
Рисунок 3. Первый этап работы AlphaFold 2: «глубокие» базы данных. В программу загружают информацию о последовательности неизвестного белка, и она ищет похожие последовательности в базах данных — и это ключевой этап: именно многообразие близко- и отдаленно родственных последовательностей в строящемся выравнивании должно указать нейросети на возможный тип укладки. Дополнительно AF2 может «подсосать» и структурную информацию, если она доступна — это повысит точность предсказания, хотя, в отличие от метода моделирования по гомологии, этот этап можно и пропустить, и результаты всё равно будут неплохими.
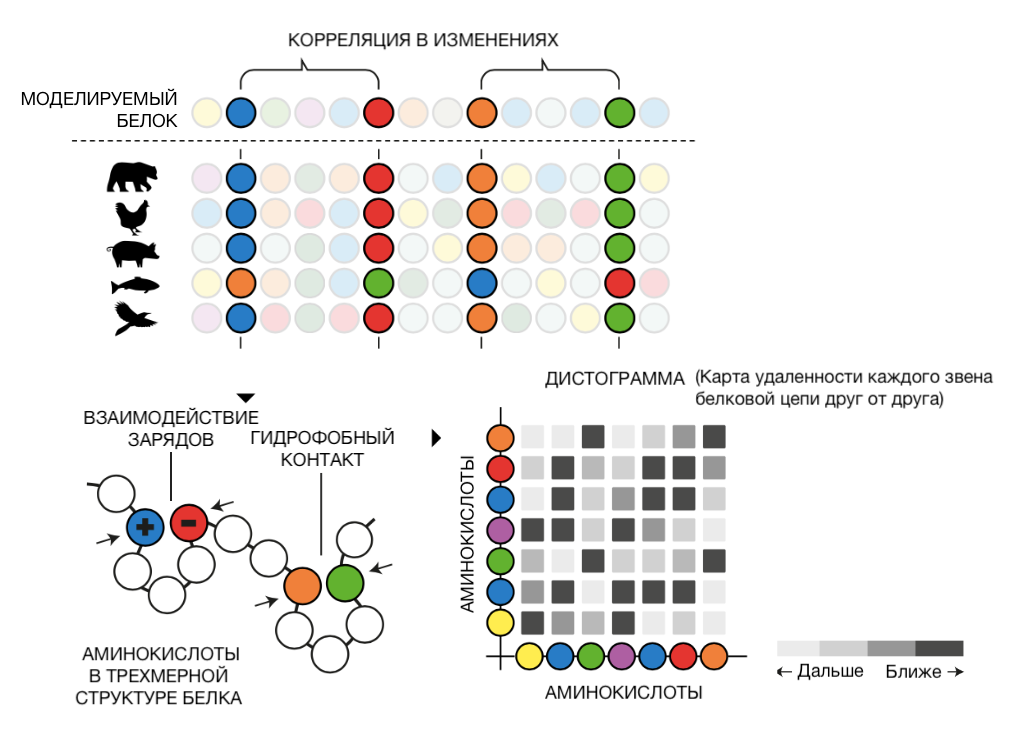
Рисунок 4. Второй этап работы AlphaFold 2: анализ собранных последовательностей. Программа изучает полученное на предыдущем шаге «глубокое» выравнивание, попутно выясняя, какие фрагменты могли бы взаимодействовать друг с другом в трехмерной структуре белка. Это замеряется по скоррелированности аминокислотных замен в разных «эволюционных ветвях» выравнивания. Например, если в белке есть зарядовое взаимодействие, образованное аминокислотными остатками противоположного заряда, то вместе с изменением свойств первого изменятся свойства второго — иначе взаимодействие будет потеряно, а белок станет нефункциональным (и вместе с ним — весь организм). На основе этого анализа AlphaFold 2 собирает оценки удаленности между аминокислотными остатками в карту, которая изображена серыми квадратами (и по-научному называется дистограммой).
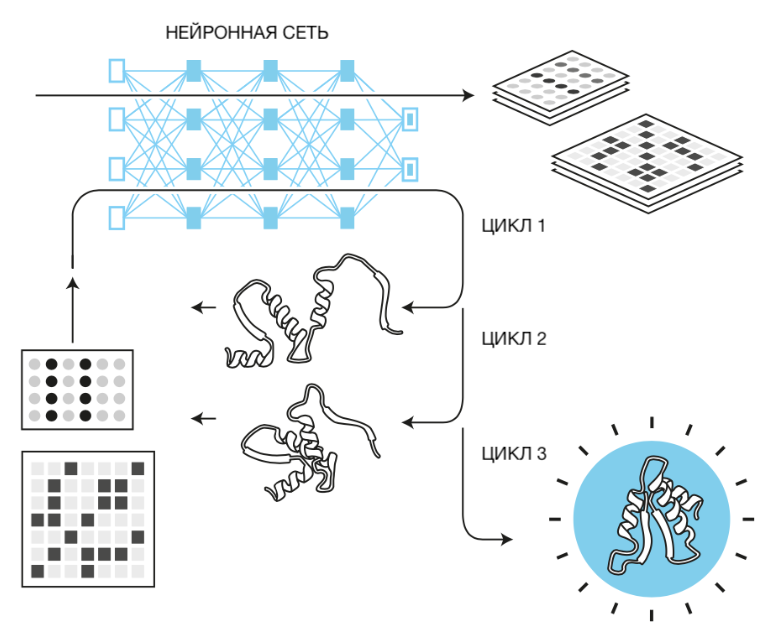
Рисунок 5. Третий и четвертый этапы работы AlphaFold 2: анализ на основе ИИ и подготовка модели структуры белка. На третьем этапе ИИ раз за разом проверяет последовательность и первоначальную карту, постепенно совершенствуя два взаимосвязанных представления белка: «одиночное» (хранит черты аминокислотного выравнивания) и «парное» (постепенно обретает черты 3D-структуры, накапливая информацию о парных расстояниях в ней). Этот процесс реализован в форме набора нейросетей-«трансформеров», приобретших популярность в задачах анализа естественного языка за способность акцентировать «внимание» на особо важных элементах данных, с которыми работает нейросеть. Четвертый этап непосредственно предсказывает структуру белка по «доставшимся в наследство» от третьего одиночному и парному представлениям, декодируя содержащуюся там информацию в форме координат, ориентаций и конформаций отдельных аминокислотных остатков. Интересно, что получающаяся структура может вновь быть отправлена на «доработку» на 3-й этап, и таких циклов может быть несколько, пока модель не «наберет силу» и не покажет достаточное качество по внутреннему алгоритму «самоконтроля», встроенному в AF2.
Проект AlphaFold стал небывало популярным, потому что Google DeepMind сделали доступ к программе открытым для исследователей по всему миру и также поделились ее кодом. А кроме прочего, создана база данных структур AlphaFold, куда выложили 200 миллионов (!) уже предсказанных структур, так что если вам придет в голову какой-то даже самый заковыристый белок, скорее всего, его модель там уже есть.
Видео 2. В этой статье всё для вас слишком просто? Ее редактор вам расскажет еще раз — чтобы вы не думали, что это легко. В лекции, прочитанной для студентов магистратуры «Вычислительная физика конденсированного состояния и живых систем» МФТИ, рассказывается про метагеномные веяния в структурной биологии и как их смог «оседлать» ИИ; про архитектуру AF2 (в т.ч. видео-цитированием одного из наших сегодняшних героев Джона Джампера); а также про других участников ИИ-гонки — RoseTTAFold, ESMFold и прочих.
лекция «AlphaFold и RoseTTAFold: нейросетевое шаманство» из курса «Новости компьютерного моделирования биосистем», прочитанная в МФТИ 27.04.2023
Видео 3. Антон Чугунов (редактор этой статьи, сооснователь «Биомолекулы» и с.н.с. Института биоорганической химии РАН) и Павел Яковлев (вице-президент BIOCAD по ранней разработке и исследованиям) беседуют о дизайне белков и нейросетях в структурной биологии — в рамках BIOCAD Guest Club. Вот о чем говорили тезисно: кому AlphaFold 2 уступает на соревнованиях по прогнозам структур белков; чем отличается задача дизайна новых белков от прогноза структуры белка; а вы знали? подход создателей AlphaFold построен на чужих идеях; чем различается работа с AlphaFold 2 и 3; какие предсказания белковых структур ученые делают лучше нейросетей; как люди улучшают работу нейросетей; есть ли «галлюцинации» у AlphaFold 2? манипулирует ли Дэвид Бейкер результатами? почему разработчики новых препаратов до сих пор опираются на старые технологии.
Что будет дальше
Одна из функций, которой ученым не хватало в AlphaFold 2 — предсказание взаимодействия белков с другими белками и малыми молекулами. Это очень важно для дизайна фармацевтических препаратов, потому что лекарственные вещества нацелены на конкретные мишени [27] (например, рецепторы в мембране болезнетворных бактерий; или иммунных клеток, вызывающих аутоиммунную реакцию). На этом специализируется Isomorphic Labs — стартап, отделившийся от Google DeepMind в 2021 в качестве полигона для применения ИИ для решения задач фармацевтики. Глобальная цель его команды во главе с Демисом Хассабисом — изменить разработку лекарств, а именно сделать ее быстрее, дешевле и снизить риски. Для этого Isomorphic Labs используют ИИ и вычислительные возможности Google DeepMind и привлекают финансирование от фармкомпаний.
В коллаборации с Isomorphic Labs в 2024 году Google Deepmind выпустили еще одну инкарнацию альфа-фолда — AlphaFold 3. Третья версия предсказывает не только структуру, но и взаимодействие белков с другими белками, ДНК, РНК и малыми молекулами иного состава. Alpha Proteo, тоже выпущенный в коллаборации в 2024 году, специализируется на дизайне «байндеров» — небольших белков, связывающихся с выбранной молекулярной мишенью наподобие антител. Доступ к AlphaFold 3 и Alpha Proteo пока ограничен. Одновременно фармкомпании, которые сотрудничают с Isomorphic Labs, получают эксклюзивные возможности новых нейросетей. Что известно об этих проектах и что они обещают для развития науки и индустрии — нам рассказали два исследователя в области вычислительной молекулярной биологии, авторы «Биомолекулы»: Артур Залевский и Павел Буслаев.
На основе пресс-релиза Нобелевского комитета.
Литература
- Роль слабых взаимодействий в биополимерах;
- Проблема фолдинга белка;
- Одна последовательность — одна структура: был ли Анфинсен неправ?;
- Торжество компьютерных методов: предсказание строения белков;
- Новые успехи в предсказании пространственной структуры белков;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- 12 методов в картинках: «сухая» биология;
- Brian Kuhlman, Gautam Dantas, Gregory C. Ireton, Gabriele Varani, Barry L. Stoddard, David Baker. (2003). Design of a Novel Globular Protein Fold with Atomic-Level Accuracy. Science. 302, 1364-1368;
- A.N. Fedorov, D.A. Dolgikh, V.V. Chemeris, B.K. Chernov, A.V. Finkelstein, et. al.. (1992). De novo design, synthesis and study of albebetin, a polypeptide with a predetermined three-dimensional structure. Journal of Molecular Biology. 225, 927-931;
- Тетрис XXI века;
- Дизайнерские ферменты на службе общества;
- Конструкторское бюро белков;
- David Silver, Thomas Hubert, Julian Schrittwieser, Ioannis Antonoglou, Matthew Lai, et. al.. (2018). A general reinforcement learning algorithm that masters chess, shogi, and Go through self-play. Science. 362, 1140-1144;
- История развития искусственного интеллекта и его пришествия в биологию;
- Как языковые модели покорили мир белков;
- N+1: Магнетизм нейросетей. Где Нобелевский комитет видит границы современной физики;
- Белковые галлюцинации: как справляется AlphaFold?;
- AlphaFold: нейросеть для предсказания структуры белков от британских ученых;
- Andrew W. Senior, Richard Evans, John Jumper, James Kirkpatrick, Laurent Sifre, et. al.. (2020). Improved protein structure prediction using potentials from deep learning. Nature. 577, 706-710;
- John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, et. al.. (2021). Highly accurate protein structure prediction with AlphaFold. Nature. 596, 583-589;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Sergey Ovchinnikov, Hahnbeom Park, Neha Varghese, Po-Ssu Huang, Georgios A. Pavlopoulos, et. al.. (2017). Protein structure determination using metagenome sequence data. Science. 355, 294-298;
- «Омики» — эпоха большой биологии;
- Факты и легенды о мультиомиксных технологиях;
- Мультиомика и Экологические тайны третьей планеты;
- Как мультиомиксы раздвигают границы современной биологии;
- Поиск лекарственных мишеней;
- Fangyu Liu, Cheng-Guo Wu, Chia-Ling Tu, Isabella Glenn, Justin Meyerowitz, et. al.. (2024). Large library docking identifies positive allosteric modulators of the calcium-sensing receptor. Science. 385.






