
Сколько у нас генов?
29 октября 2018
Сколько у нас генов?
- 11463
- 2
- 7
Найти ответ на этот вопрос оказалось куда сложнее, чем кто-либо предполагал
-
Автор
-
Редактор
Статья на конкурс «био/мол/текст»: Это интересный вопрос, ответ на который должен был дать проект «Геном человека», завершившийся в 2003 году. После того как ученые получили основную информацию о геноме человека, они попытались определить число генов, но эта задача оказалось не такой простой. Цель настоящей статьи — суммировать и проанализировать научные данные по составлению каталога генов у человека.
Конкурс «био/мол/текст»-2018
Эта работа опубликована в номинации «Свободная тема» конкурса «био/мол/текст»-2018.

Генеральный спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

Спонсором приза зрительских симпатий выступил медико-генетический центр Genotek.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Как же мало известно о генах! Первый раз я остро ощутила это, находясь на практике в лаборатории медицинской генетики Харбинского медицинского университета. Исследовательская группа, где я проходила стажировку, занималась изучением онкогена Sei-1, который индуцирует образование двухминутных хромосом (DM), что способствует развитию онкогенеза. Однако механизм образования онкогена Sei-1 остается неизвестным до сих пор. А ведь различные мутации генов являются причиной возникновения и других опасных заболеваний человека, помимо рака. Итак, в данной статье мы изложим некоторые соображения о том, почему мы все еще многое не знаем о генах, а также сформулируем наше мнение о том, сколько генов у человека.
В 1977 году Фредерик Сэнгер впервые разработал метод секвенирования ДНК [1], основанный на терминации ДНК-полимеразной реакции с помощью дидезоксинуклеотидов, за что в 1980 году был удостоен Нобелевской премии в области химии. В этом же году Нобелевскую премию получили Максам и Гилберт, которые предложили метод секвенирования ДНК путем химической деградации. В 1985 году была выявлена первая полная последовательность ДНК бактерии (Haemophilus influenzae), в 1996 году получен первый геном эукариотической клетки (дрожжи Saccharomyces ceravisiae), а в 1998 году расшифрован геном дождевого червя (Caenorhabditis elegans). Завершение в 2003 году проекта «Геном человека» привело к публикации полной последовательности человеческого генома. Но «полной» ее можно назвать весьма условно, учитывая, что около 8% не секвенировано и по сей день [2].
Проект «Геном человека» и полный список генов
Выявление полного списка генов необходимо для выяснения молекулярных механизмов возникновения и развития рака, шизофрении [3], деменции, а также многих других заболеваний человека. Секвенирование ДНК, выделенной из тканей больных, позволяет выявлять такие мутации, как нуклеотидные замены, делеции и вставки, ответственные за возникновение этих заболеваний.

Рисунок 1. Арт-проект на выставке «Геном — расшифровка кода жизни» в Национальном музее естественной истории в Вашингтоне
Собственно, ради этого и затевался проект «Геном человека» (Human genome project, HGP), который продолжался с 1990 по 2003 год. Его основной задачей было определение нуклеотидной последовательности ДНК человека и локализации 100 000 человеческих генов (как тогда полагали) [4]. Параллельно с этим планировалось изучить ДНК набора модельных организмов, чтобы получить сравнительную информацию, необходимую для понимания функционирования генома человека. Предполагалось, что информация, полученная в результате HGP, станет настольной книгой для биомедицинской науки в XXI веке [5]. Целями данных исследований являлось получение информации о причинах ряда болезней [6] и, в конечном итоге, разработка способов лечения более чем 4000 генетических заболеваний, которые затрагивают человечество, включая многофакторные, в которых генетическая предрасположенность играет важную роль. Считалось, что результаты секвенирования генома позволят определить локализацию каждого гена и их общее количество. Однако последовавшие за этим события доказали обратное: сегодня существует несколько баз данных генов, которые существенно отличаются друг от друга. Причем число белок-кодирующих генов совпадает, а число генов других типов расходится.
Проект «Протеом человека»
В 2010 году по инициативе Организации по изучению протеома человека (Human proteome organization, HUPO) был начат проект «Протеом человека» (HPP), целью которого является создание полного списка белков вида Homo sapiens [7]. Для этого, во-первых, предполагается идентифицировать и охарактеризовать, по крайней мере, по одному белковому продукту белок-кодирующих генов, их однонуклеотидные полиморфизмы и варианты сплайсинга, а также виды посттрансляционной модификации белков [8]. Во-вторых, данные протеомики, полученные в результате реализации HPP, способствуют, в дополнение к геномным данным, решению различных биомедицинских задач и созданию новых аннотированных баз знаний, таких как neXtProt [9].
В настоящее время neXtProt содержит информацию о 17 487 белках, существование которых экспериментально подтверждено, 1728 белках, подтвержденных на уровне транскриптов, 515-и, определенных на основании гомологии, 76-и предсказанных и 571-м неизвестной природы. Особый интерес вызывают белки, существование которых экспериментально не доказано, хотя данные о том, что они кодируются геномом, существуют. Это так называемые «потерянные» белки, которые составляют примерно 18% всех кодируемых белков. Для выявления и характеристики таких белков создан ресурс MissingProteinPedia [7].
«Протеом человека» является продолжением проекта «Геном человека». Предполагается, что благодаря проекту по изучению протеома мы узнаем точное количество белок-кодирующих генов, что впоследствии позволит понять, сколько всего генов у человека.
Немного о РНК
Проект «Геном человека» показал, что молекулы РНК также важны для жизни, как и ДНК. Внутри клеток существует множество РНК (рис. 2). Изначально РНК подразделяются на некодирующие РНК (нкРНК), которые не транслируются в белки, и кодирующие РНК (мРНК), служащие матрицей для синтеза полипептидных цепей белка. Некодирующие РНК имеют более сложную классификацию. Они бывают инфраструктурными и регуляторными. Инфраструктурные РНК представлены рибосомными РНК (рРНК) и транспортными РНК (тРНК). Молекулы рРНК синтезируются в ядрышке и составляют основу рибосомы, а также кодируют белки субъединиц рибосомы. После того, как рРНК полностью собраны, они переходят в цитоплазму, где в качестве ключевых регуляторов трансляции, участвуют в чтении кода мРНК. Последовательность из трех азотистых оснований в мРНК указывает на включение определенной аминокислоты в последовательность белка. Молекулы тРНК, приносят указанные аминокислоты на рибосомы, где синтезируется белок.
Дополнительно о РНК читайте в статьях «Биомолекулы»: «Обо всех РНК на свете, больших и малых», «Кодирующие некодирующие РНК» и «Власть колец: всемогущие кольцевые РНК» [10–12].
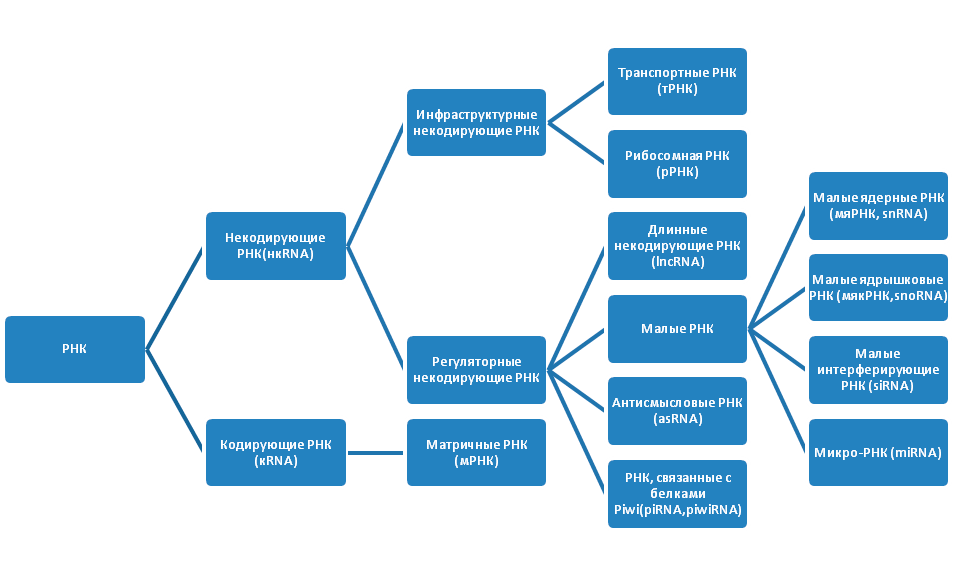
Рисунок 2. Виды РНК
рисунок автора статьи
Регуляторные нкРНК очень широко представлены в организме, классифицируются в зависимости от размера и выполняют ряд важных функций (табл. 1).
| Название | Обозначение | Длина | Функции | |
|---|---|---|---|---|
| Длинные некодирующие РНК | днкРНК, lncRNA | 200 нуклеотидов | 1. Регулируют избирательное метилирование ДНК, направляя ДНК-метилтрансферазу 2. Руководят избирательной посадкой репрессорных комплексов polycomb |
|
| Малые РНК | Малые ядерные РНК | мяРНК, snRNA | 150 нуклеотидов | 1. Участвуют в сплайсинге 2. Регулируют активность факторов транскрипции 3. Поддерживают целостность теломер [13] |
| Малые ядрышковые РНК | мякРНК, snoRNA | 60–300 нуклеотидов | 1. Участвуют в химической модификации рРНК, тРНК и мяРНК 2. Возможно, участвуют в стабилизации структуры рРНК и защите от действия гидролаз |
|
| Малые интерферирующие РНК | миРНК, siRNA | 21–22 нуклеотидов | 1. Осуществляют антивирусную иммунную защиту 2. Подавляют активность собственных генов |
|
| Микро-РНК | мкРНК, miRNA | 18–25 нуклеотидов | Подавляют трансляцию путем РНК-интерференции | |
| Антисмысловые РНК [14] | asRNA | 1. Короткие: менее 200 нуклеотидов 2. Длинные: более 200 нуклеотидов |
Блокируют трансляцию, образуя гибриды с мРНК | |
| РНК, связанные с белками Piwi | piRNA, piwiRNA | 26–32 нуклеотидов | Их также называют «стражами генома», они подавляют активность мобильных генетических элементов во время эмбриогенеза | |
Проблема терминологии
Прежде чем ответить на вопрос: «Сколько у нас генов?», нужно понять, что же такое ген?
Основное внимание HGP было направлено на белок-кодирующие гены [15]. Однако, как было указано в первоначальном докладе HGP в 2001 году, «тысячи генов человека продуцируют некодирующие РНК (нкРНК), являющиеся их конечным продуктом», хотя на тот момент было известно около 706 генов нкРНК [2]. В своей недавней статье, опубликованной в журнале BMC Biology Стивен Зальцберг (Steven L. Salzberg) дает следующее определение гена [16]:
- Ген
- любой участок хромосомной ДНК, который транскрибируется в функциональную молекулу РНК или сначала транскрибируется в РНК, а затем транслируется в функциональный белок.
Это определение включает как гены некодирующих РНК, так и белок-кодирующие гены, и позволяет определять все варианты альтернативного сплайсинга в одном локусе как варианты одного и того же гена. Это позволяет исключить псевдогены – нефункциональные остатки структурных генов, утратившие способность кодировать белок.
Результаты двух первых исследований свидетельствовали о наличии у человека 31 000 [2] и 26 588 белок-кодирующих генов [17], а в 2004 появилась полная последовательность генома человека [4], и авторы подсчитали, что полный каталог насчитывает 24 000 белок-кодирующих генов. Каталог человеческих генов Ensembl включает 22 287 белок-кодирующих генов и 34 214 транскриптов [18].
Секвенирование нового поколения (NGS)
Появление высокопроизводительных методов параллельного секвенирования (в ходе такого секвенирования миллионы фрагментов ДНК из одного образца секвенируются одновременно) или секвенирования нового (следующего) поколения (next-generation sequencing, NGS) [1] позволило значительно ускорить поиск функциональных участков генома [4]. Биотехнологические компании разработали и коммерциализировали различные платформы для NG-секвенирования, позволяющие секвенировать от 1 млн до десятков млрд коротких последовательностей (ридов, reads) длиной 50–600 нуклеотидов каждая. К наиболее популярным платформам относятся такие, как Illumina и IonTorrent, использующие амплификацию ДНК с помощью ПЦР [19], а также платформы одномолекулярного секвенирования, такие как Helicos Biosciences HeliScope, Pacific Biosciences SMRT (single molecule real-time sequencing), и нанопорового секвенирования Oxford Nanopore, осуществляющие секвенирование в реальном времени и позволяющие прочитывать значительно более длинные риды — до 10–60 тыс. нуклеотидов. Кроме того, изобретение секвенирования РНК (RNA-seq) в 2008 году, которое создавалось для количественного определения экспрессии генов, также способствовало обнаружению транскрибируемых последовательностей, как кодирующих, так и некодирующих РНК [20].
Благодаря NGS, базы данных днкРНК и других генов РНК (таких как микро-РНК) резко выросли за десятилетие, и текущие каталоги генов человека теперь содержат больше генов, кодирующих РНК, чем белки (табл. 2).
| Типы генов | Gencode | Ensembl | RefSeq | CHESS |
|---|---|---|---|---|
| Белок-кодирующие гены | 19 901 | 20 376 | 20 345 | 21 306 |
| Гены длинных некодирующих РНК | 15 779 | 14 720 | 17 712 | 18 484 |
| Антисмысловые РНК | 5501 | — | 28 | 2694 |
| Другие некодирующие РНК | 2213 | 2222 | 13 899 | 4347 |
| Псевдогены | 14 723 | 1740 | 15 952 | — |
| Общее число транскриптов | 203 835 | 203 903 | 154 484 | 323 827 |

Рисунок 3. Последовательность ДНК, получаемая после секвенирования человеческого генома
В ходе секвенирования РНК обнаружилось, что альтернативный сплайсинг, альтернативное инициирование транскрипции и альтернативное прерывание транскрипции проиcходят гораздо чаще, чем полагали, затрагивая до 95% человеческих генов. Следовательно, даже если известно местоположение всех генов, сначала нужно выявить все изоформы этих генов, а также определить, выполняют ли эти изоформы какие-либо функции или они просто представляют собой ошибки сплайсинга.
Базы данных генов человека
Задача по составлению каталога всех генов по-прежнему не решена. Проблема заключается в том, что за последние 15 лет только две исследовательские группы составили список доминантных генов: RefSeq, которая поддерживается Национальным центром биотехнологической информации (NCBI) при Национальных институтах здоровья (NIH), и Ensembl/Gencode, которая поддерживается Европейской молекулярно-биологической лабораторией (EMBL). Однако, несмотря на большой прогресс, сейчас в каталогах различается количество белок-колирующих генов, генов длинных некодирующих РНК, псевдогенов, а также варьирует количество антисмысловых РНК и других некодирующих РНК (табл. 2). Каталоги еще дорабатываются: например, в прошлом году сотни генов, кодирующих белок, были добавлены или удалены из списка Gencode. Эти разногласия объясняют проблему создания полного каталога человеческих генов.
В 2017 году была создана новая база данных генов человека — CHESS. Примечательно, что она включает все белок-кодирующие гены как Gencode, так и RefSeq, так что пользователям CHESS не нужно решать, какую базу данных они предпочитают. Бóльшее количество генов может вызывать больше ошибок, но создатели считают, что бóльший набор окажется полезным при исследовании болезней человека, которые еще не отнесены к генетическим. Набор генов CHESS в настоящее время в версии 2.0 еще не окончательный, и, безусловно, создатели работают над его усовершенствованием.
Таким образом, все еще неизвестно, сколько всего генов у человека. Существует ряд проблем, затрудняющих эту задачу. Например, многие гены (особенно, гены днкРНК), видимо, имеют высокую тканеспецифичность. Из этого следует, что пока ученые подробно не исследуют все типы клеток человека, они не могут быть уверены, что обнаружили все человеческие гены и транскрипты. Безусловно, сегодня знания о человеческих генах стали значительно обширнее, чем в начале проекта «Геном человека», а технологии совершеннее. Это дает надежду на то, что в скором времени мы узнаем точный ответ на поставленный вопрос.
Литература
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Eric S. Lander, Lauren M. Linton, Bruce Birren, Chad Nusbaum, Michael C. Zody, et. al.. (2001). Initial sequencing and analysis of the human genome. Nature. 409, 860-921;
- Власть колец: всемогущие кольцевые РНК;
- International Human Genome Sequencing Consortium. (2004). Finishing the euchromatic sequence of the human genome. Nature. 431, 931-945;
- Mohammad Ilyas. (2017). Next-Generation Sequencing in Diagnostic Pathology. Pathobiology. 84, 292-305;
- Геном человека: полезная книга, или глянцевый журнал?;
- Mark S. Baker, Seong Beom Ahn, Abidali Mohamedali, Mohammad T. Islam, David Cantor, et. al.. (2017). Accelerating the search for the missing proteins in the human proteome. Nat Comms. 8, 14271;
- SUMO: японская борьба или уникальная посттрансляционная модификация?;
- Pascale Gaudet, Pierre-André Michel, Monique Zahn-Zabal, Aurore Britan, Isabelle Cusin, et. al.. (2017). The neXtProt knowledgebase on human proteins: 2017 update. Nucleic Acids Res. 45, D177-D182;
- Обо всех РНК на свете, больших и малых;
- Кодирующие некодирующие РНК;
- Власть колец: всемогущие кольцевые РНК;
- Цинковые пальцы смерти;
- Есть ли смысл в антисенсах?;
- Геном человека: как это было и как это будет;
- Steven L. Salzberg. (2018). Open questions: How many genes do we have?. BMC Biol. 16;
- J. C. Venter, H. O. Smith, M. D. Adams. (2015). The Sequence of the Human Genome. Clinical Chemistry. 61, 1207-1208;
- Eric T. Wang, Rickard Sandberg, Shujun Luo, Irina Khrebtukova, Lu Zhang, et. al.. (2008). Alternative isoform regulation in human tissue transcriptomes. Nature. 456, 470-476;
- 12 методов в картинках: полимеразная цепная реакция;
- Hassaan Mehboob Awan, Abdullah Shah, Farooq Rashid, Ge Shan. (2017). Primate-specific Long Non-coding RNAs and MicroRNAs. Genomics, Proteomics & Bioinformatics. 15, 187-195.






