Транслезионный синтез ДНК: выжить любой ценой?
16 февраля 2022
Транслезионный синтез ДНК: выжить любой ценой?
- 2284
- 0
- 5
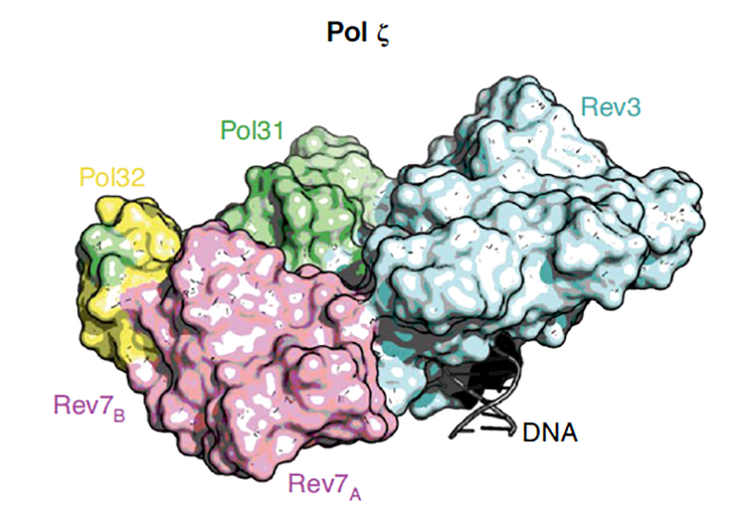
Доменная структура транслезионной ДНК-полимеразы Pol ζ.
Рисунок в полном размере.
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Феномен транслезионного синтеза ДНК (далее — ТЛС; от англ. translesion DNA synthesis — синтез ДНК через повреждение), к сожалению, мало где освещается. В учебниках по молекулярной биологии обычно об этом не пишут. Да и не сказать, что научных статей по этой тематике превеликое множество. Несмотря на вышеперечисленные факты, ТЛС является весьма значимым процессом, необходимым, в первую очередь, для поддержания целостности и стабильности генома клетки. В связи со сложившийся ситуацией автору хотелось бы пролить свет на данную, как ему кажется, неоднозначную, но столь важную тему. В этом обзоре я постарался изложить как фундаментальные аспекты данного процесса, так и затронуть прикладную сторону его изучения. А также попробовал ответить на вопрос (в какой-то степени философский), вынесенный в заголовок статьи.
Конкурс «Био/Мол/Текст»-2021/2022
Эта работа опубликована в номинации «Свободная тема» конкурса «Био/Мол/Текст»-2021/2022.

Партнер номинации — компания SkyGen: передовой дистрибьютор продукции для life science на российском рынке.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Генеральный партнер конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Повреждение повреждению рознь
Казалось бы, генетический материал наших клеток, представленный в виде ДНК, должен быть суперстабильной структурой, чтобы обеспечивать как можно более точную передачу наследственной информации дочерним клеткам. Звучит максимально логично. Однако на деле есть пара нюансов. Сложно поверить, что клеточная ДНК только за день подвергается десяткам тысяч различных повреждений [1]! «Ну и что тут такого? — может возразить читатель. — У нас же есть системы репарации, они подлатают нашу ДНК!». В целом, конечно, он прав. Однако, к сожалению, не все так просто, как кажется на первый взгляд.
Конечно, системы репарации есть, и их несколько, и все они работают по различным механизмам [2]. Но и разнообразие повреждений, а также факторов, которые их вызывают, тяжело себе вообразить. В целом, повреждения ДНК могут являться результатом как внешних (экзогенных), так и внутренних (эндогенных) факторов.
К наиболее важным экзогенным факторам можно отнести ультрафиолетовое (УФ) излучение, ионизирующее излучение (ИИ) и различные (коих бесчисленное множество) химические агенты, модифицирующие ДНК. А такие процессы, протекающие внутри клеток практически каждого организма, как клеточное дыхание, перекисное окисление липидов, ошибочная репликация, спонтанное дезаминирование оснований, составляют группу эндогенных факторов [3].
NB! Важно разграничить понятия «повреждение ДНК» и «мутация». Под повреждением ДНК обычно подразумевают такое изменение первичной структуры ДНК, которое еще возможно вернуть к исходному состоянию. Мутация же представляет собой стойкое и необратимое изменение первичной структуры ДНК, которое будет передаваться дочерним клеткам. Таким образом, повреждение ДНК является потенциальной мутацией. В рамках данной статьи мы будем говорить преимущественно о мутациях, происходящих на генном уровне (не путать с геномными мутациями), а также затронем хромосомные перестройки.
Про виды повреждений, а также факторы, которые способствуют их появлению, можно было бы написать, как минимум, отдельный обзор, но мы ограничимся лишь их краткой характеристикой.
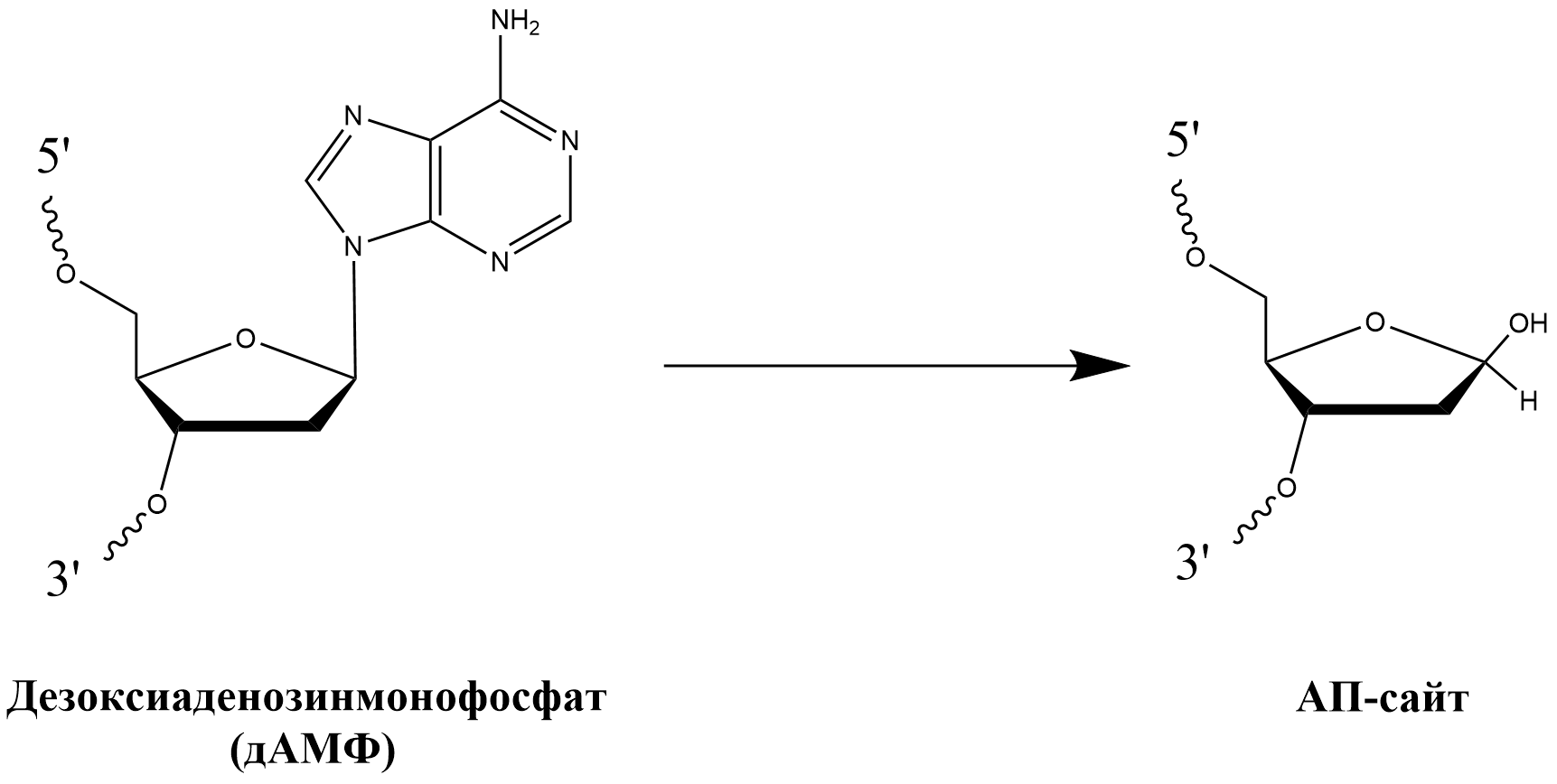
Начнем наш краткий разбор с наиболее «популярного» повреждения — апуриновый/апиримидиновый сайт (АП-сайт) (рис. 1А). Структура АП-сайта в буквальном смысле представляет собой голый сахарофосфатный «скелет», т.е. дезоксирибозу с фосфатной группой, лишенную азотистого основания. А лишиться его несложно — достаточно разорвать N-гликозидную связь между ним и сахаром. Большинство подобных повреждений возникают именно в результате самых обычных физиологических процессов, протекающих в клетке [3].
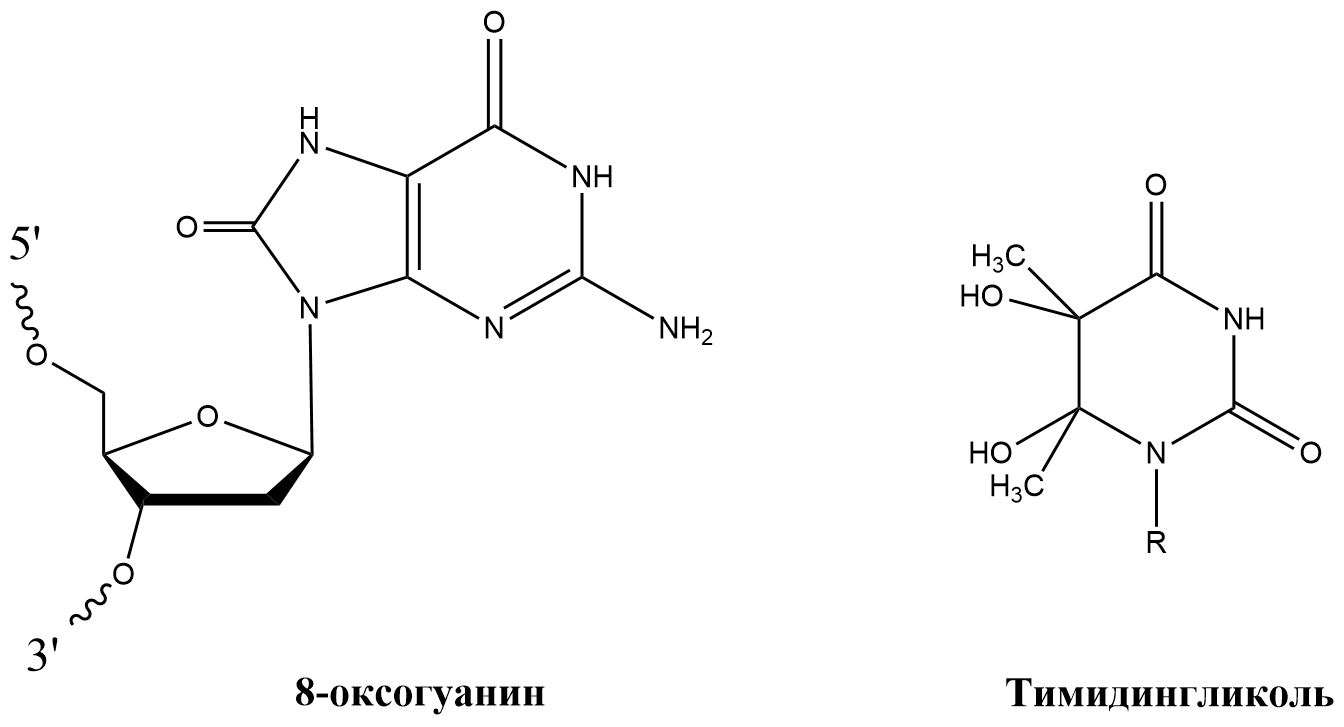
Окислительные повреждения также занимают одно из главных мест в структуре повреждений ДНК (рис. 1Б). Главные виновники — побочные продукты, образующиеся в ходе переноса электронов по дыхательной цепи, благодаря активности которой мы и многие другие организмы получаем энергию. Другими словами, такой физиологический процесс, как дыхание, является причиной образования активных форм кислорода (тех самых побочных продуктов), повреждающих ДНК. Окислению чаще всего подвергается гуанин с образованием 8-оксогуанина и его производных. Не менее опасными повреждениями являются тимидингликоль и различные окисленные «варианты» 5-метилцитозина [3].
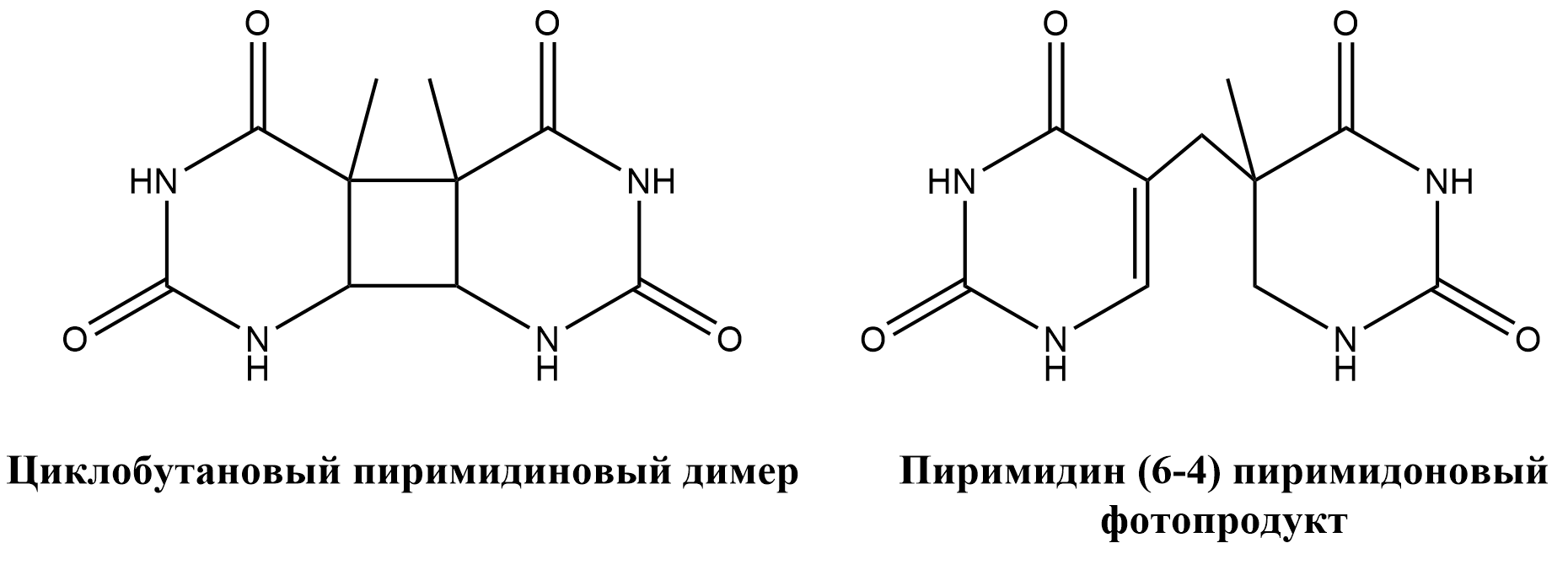
Крайне мутагенные повреждения — фотопродукты, которые появляются, в первую очередь, из-за поглощения нуклеиновыми кислотами УФ излучения (рис. 1В). К таким относятся циклобутановые пиримидиновые димеры и (6–4) фотопродукты. Свойство УФ излучения — димеризация соседних тиминов — обусловлено его способностью образовывать ковалентную сшивку между ними [4].
Количество видов повреждений, вызванных действием алкилирующих агентов, тяжело поддается исчислению, поскольку модифицировать азотистые основания (а сайтов, по которым их можно алкилировать, тоже немало) потенциально может огромный спектр химических веществ, преимущественно экзогенного характера. Вышеперечисленные повреждения; повреждения, индуцированные химиотерапией при онкозаболеваниях; 1,N6-этеноаденин, дезаминированные основания, одно- и двухцепочечные разрывы ДНК... Список можно продолжать практически бесконечно. Повреждений очень много, и каждое, можно сказать, уникально в своем роде. Однако эволюция — штука хитрая, и почти на каждое повреждение найдется та или иная ферментативная система, которая поможет восстановить исходную структуру ДНК.
В зависимости от этиологии и типа повреждения, а также фазы клеточного цикла, задействуется тот или иной путь репарации. Стоит отметить, что большинство механизмов репарации являются высококонсервативными процессами, которые используются как у прокариот, так и эукариот. Конечно, различия между ними есть, но в целом механизмы очень похожи.
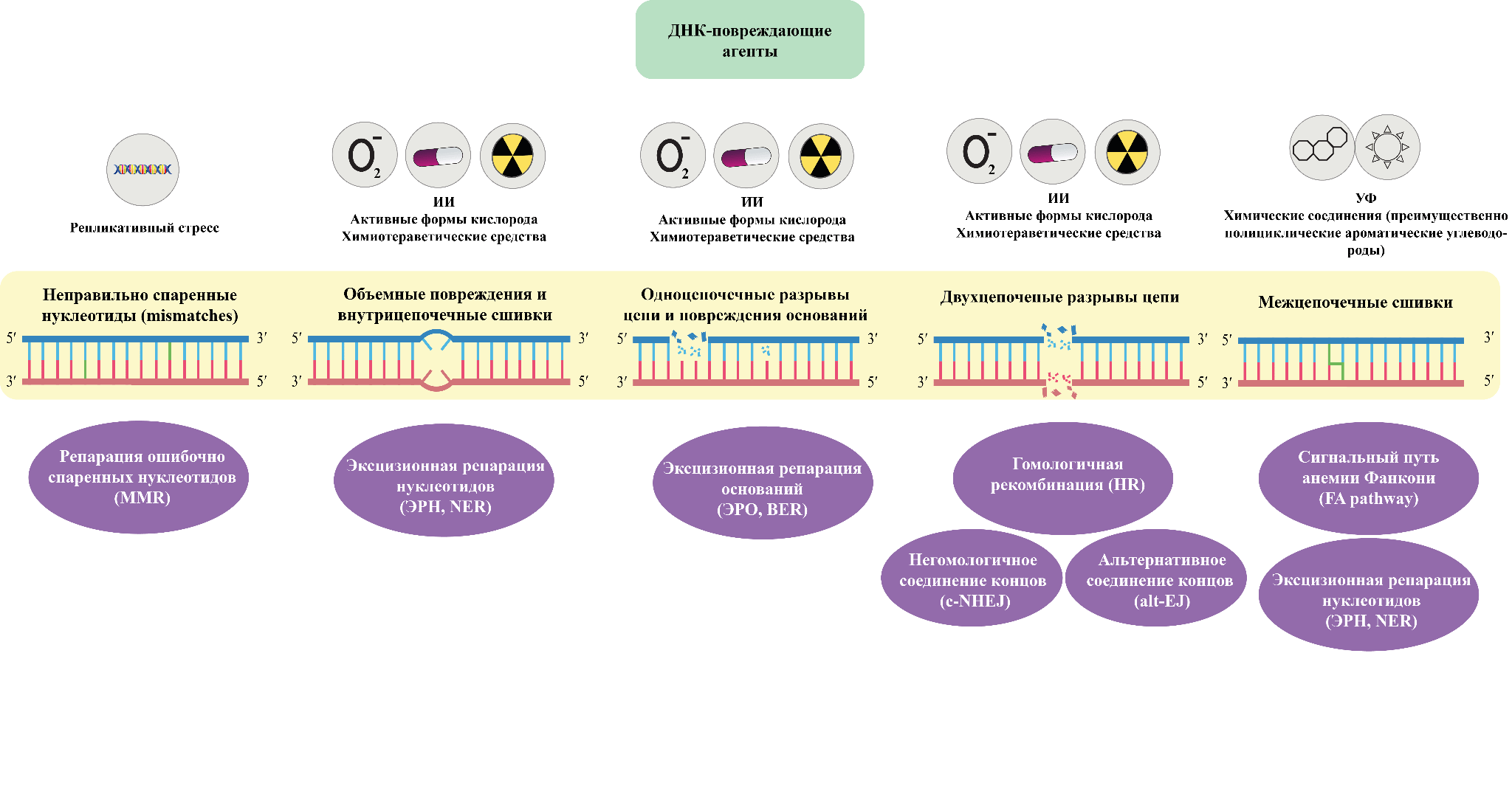
Рисунок 2. Основные репаративные механизмы, активирующиеся в ответ на различные виды повреждений ДНК.
частично адаптировано из BioRender
Клеточный цикл: а часики-то тикают
Неспроста отмечен тот факт, что активация той или иной системы репарации зависит от фазы клеточного цикла. Большинство репаративных систем активируются вне синтетической (S) фазы клеточного цикла — либо на ее ранних стадиях, что на самом деле вполне логично. Поскольку удвоение генетического материала клетки происходит в S фазу, то клетке попросту нужно успеть «отремонтировать» повреждения ДНК до момента его наступления, чтобы они не стали мутациями и не закрепились в геноме [5].
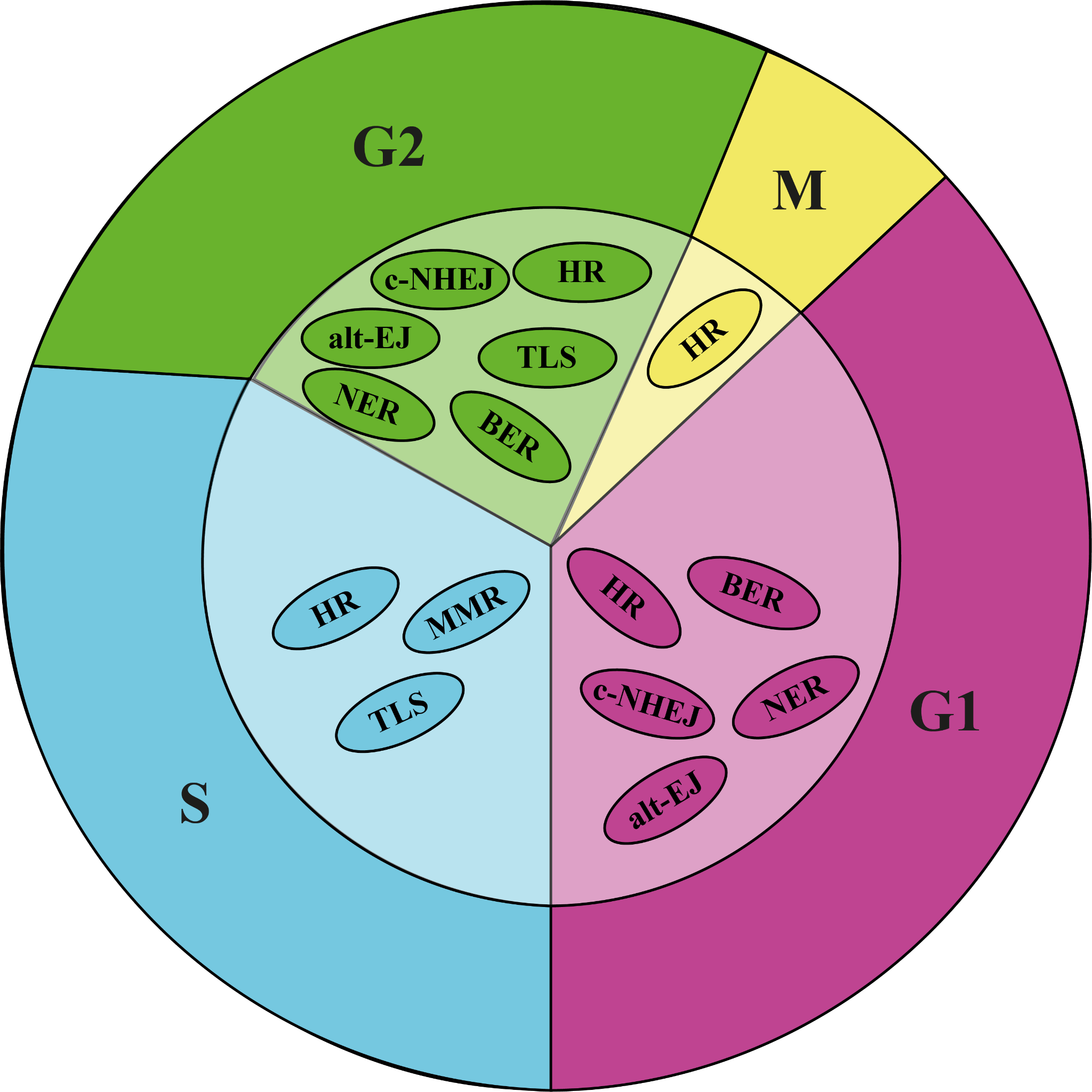
Рисунок 3. Клеточный цикл, контролирующий время активации систем репарации и ТЛС. Тем не менее, некоторые стадии одного и того же процесса (например, гомологичной рекомбинации), могут происходит в разные фазы клеточного цикла, поэтому данное разделение весьма условно, а его границы размыты.
Однако по некоторым причинам повреждения ДНК могут ускользать от систем репарации, что гипотетически может приводить к неприятным последствиям (так, например, для эксцизионной репарации нуклеотидов в приоритете репарация (6–4) фотопродуктов, а вот некоторое количество циклобутановых пиримидиновых димеров «перекочует» в S фазу) [6]. Просто напомню, что человеческий геном все-таки немаленьких размеров, и порой даже нескольких репаративных систем недостаточно, чтобы устранить все последствия ДНК-повреждающих агентов.
Так что же делать, если клеточной ДНК не удалось успеть «залечить свои раны» перед репликацией? Отвечу, что не системами репарации едины, поскольку они являются лишь частью (хоть и значительной) более общего, более «объемного» метаболического пути под названием «ответ на повреждение ДНК» (DNA damage response, DDR) [7]. Помимо различных путей репарации, ответ на повреждение ДНК также может задействовать путь устойчивости к повреждениям ДНК (DDT, от англ. DNA damage tolerance). Конечно, на этапе S фазы, например, работает гомологичная рекомбинация, однако ее «субстратом» служат преимущественно двухцепочечные разрывы. Как быть с остальными типами повреждениями? Вот тут-то и приходит на помощь DDT, активирующий ТЛС. Чтобы понять, как он функционирует, нужно немного погрузиться в уже несколько раз упомянутый процесс репликации. Давайте попробуем более детально рассмотреть данный процесс.

Рисунок 4. Ответ на повреждение ДНК. У клетки есть много способов борьбы с возникновением повреждений ее генетического материала.
рисунок автора статьи
«Столовые приборы» репликации
Мы не будем подробно останавливаться на том, как инициируется репликация, как осуществляется ее регуляция и на других тонкостях этого многогранного процесса. Тем не менее, позволю себе напомнить ключевые аспекты репликации ДНК. Кроме того, предлагаю заострить внимание на таких ключевых компонентах реплисомы, как ДНК-полимеразы. Еще в 1957 году ДНК-полимераза (которая, как позднее оказалось, далеко не одна) была впервые выделена из E. coli американским ученым Артуром Корнбергом, что положило начало изучению ферментов данного класса и репликации в целом [8]. А уже в следующем году блестящие опыты Мезельсона и Сталя позволили раскрыть основные принципы репликации, главный из которых — ее полуконсервативность [9]. Тем не менее, лишь спустя более чем 30 лет была предложена та модель репликации и репликативной вилки, которая известна нам на данный момент (хотя, стоит сказать, небольшие модификации она все же претерпела) [10]. Были совершены открытия и других репликативных ДНК-полимераз. Так, установлено, что в репликации ядерной ДНК эукариот задействовано три ДНК-полимеразы — Pol α, Pol δ и Pol ε. А для репликации митохондриальной ДНК животных предназначен отдельный фермент — Pol γ.
Конечно, ДНК-полимеразы — далеко не единственные участники репликации ДНК. Перед тем, как они «войдут в игру», саму репликацию нужно инициировать, что обычно происходит в специальных областях ДНК под названием точки ori. В таких сайтах в первую очередь происходит образование репликативных вилок. И чтобы их создать, требуется:
- (а) денатурировать участок (разорвать водородные связи между основаниями обеих цепей) в этой области;
- (б) загрузить в данный участок хеликазы, которые будут раскручивать ДНК по мере своего продвижения в противоположные стороны в направлении 5’→3’ (соблюдая, конечно же, принцип антипараллельности цепей), в результате чего образуются две репликативные вилки;
- (в) стабилизировать одноцепочечную ДНК за счет ее связывания со специальными белками и снять топологическое напряжение, которое возникает впереди вилки из-за раскручивания ДНК.
И только после вышеперечисленных событий на «подготовленную почву» приходят ДНК-полимеразы, а по итогу образуется целый белковый ансамбль — реплисома.
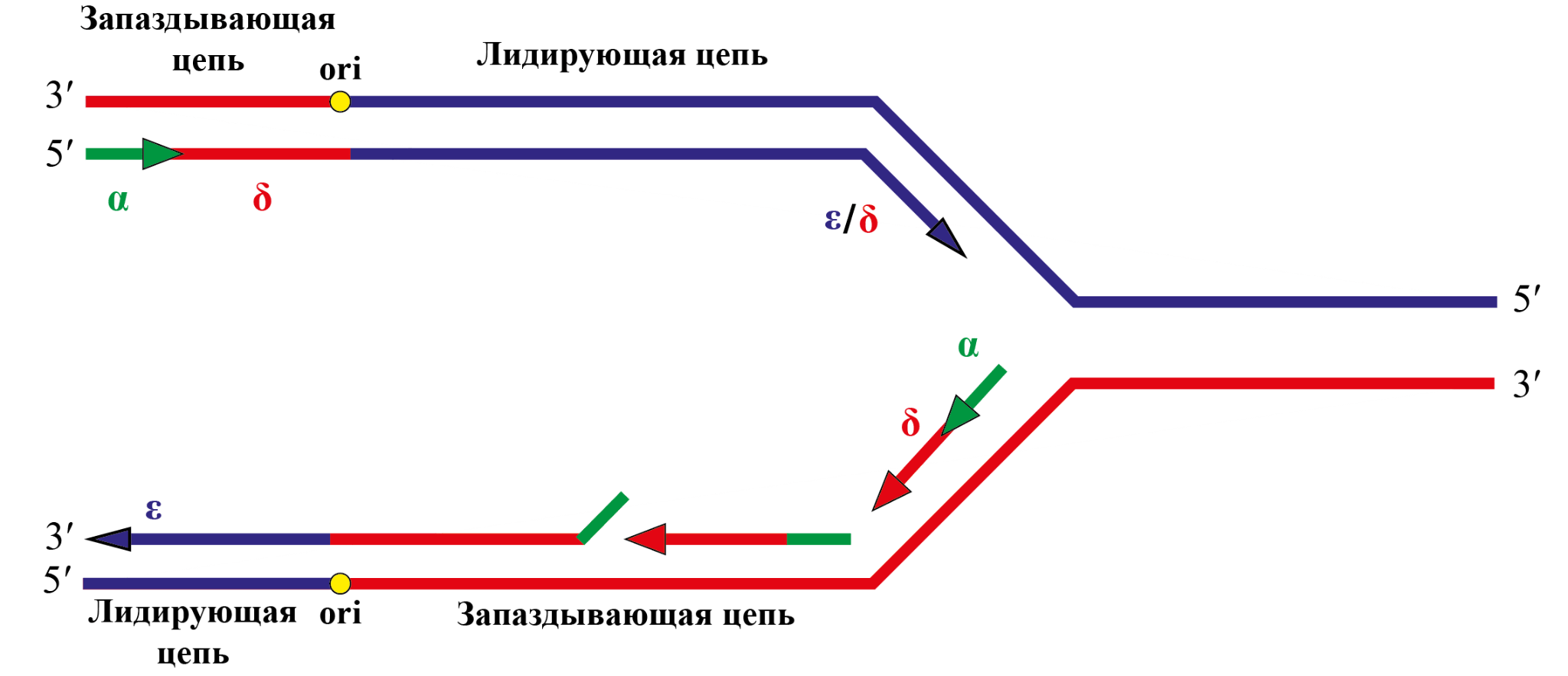
Рисунок 5. Современный взгляд на репликативную вилку и ее участников. Проиллюстрирован двунаправленный синтез, идущий от 5’-конца к 3’-концу из точек ori. Pol α, сочетающая в себе функции праймазы и полимеразы, синтезирует короткие затравки — праймеры, состоящие из рибонуклеотидов (которые впоследствии заменяются на дезоксинуклеотиды). Причем делает она это лишь на запаздывающей цепи, а первый новосинтезированный фрагмент Оказаки, в свою очередь, сам становится затравкой для Pol ε, работающей на лидирующей цепи (и только лишь на ней). Pol ε обеспечивает синтез запаздывающей цепи, хотя иногда помогает Pol ε строить лидирующую.
адаптировано из [10]
Теперь подбираемся к самому интересному. Представим, что наконец репликация инициировалась, громоздкая «машина» под названием реплисома собралась, и репликативная вилка от точки ori начинает свое путешествие. И вдруг прямо по курсу она замечает повреждение! То самое повреждение, которое каким-то чудом ускользнуло от систем репарации. Что же происходит при встрече с повреждением? Наиболее частый исход — застревание (stalling) репликативной вилки [11]. Иными словами, вилка просто останавливается на сайте повреждения и не имеет возможности продвинуться дальше. Такое состояние приводит к репликативному стрессу. На текущий момент науке известно несколько исходов такого явления. А ведь незаконченная репликация может грозить запуском апоптоза... Неужели клетка погибнет при таком раскладе? И вообще, почему вилка останавливается?
Вот где собака зарыта!
Причины, прежде всего, кроются в репликативной ДНК-полимеразе, а точнее — в ее строении. Вообще, чтобы описать строение практически любой ДНК-полимеразы, исследователи часто прибегают к ее сравнению с... правой рукой человека. Весьма необычная аналогия, как кажется автору (по правде говоря, сколько бы он ни смотрел на структуры ДНК-полимераз, лично ему очень тяжело разглядеть здесь руку). Любой фермент содержит область под названием активный центр, и полимеразы не исключение. И вне зависимости от вида ДНК-полимеразы, все они работают по примерно одному и тому же принципу [8].
Активный центр обеспечивает как связывание субстрата, так и его катализ. Каталитический кор ДНК-полимераз, ответственный, собственно, за полимеразную активность фермента, обычно представляет собой совокупность трех доменов, между которыми распределены различные функции. Непосредственно катализ реакции обеспечивает домен Palm (ладонь), в то время как другие домены — Fingers (пальцы) и Thumb (большой палец) осуществляют связывание субстрата (в данном случае субстрат у нас — входящий дезоксирибонуклеозидтрифосфат, дНТФ), удержание матричной и связывание растущей цепи ДНК. И примерно это выглядит так, что полураскрытая кисть правой руки как бы обхватывает вышеперечисленные «компоненты», необходимые для осуществления полимеразной реакции [15].
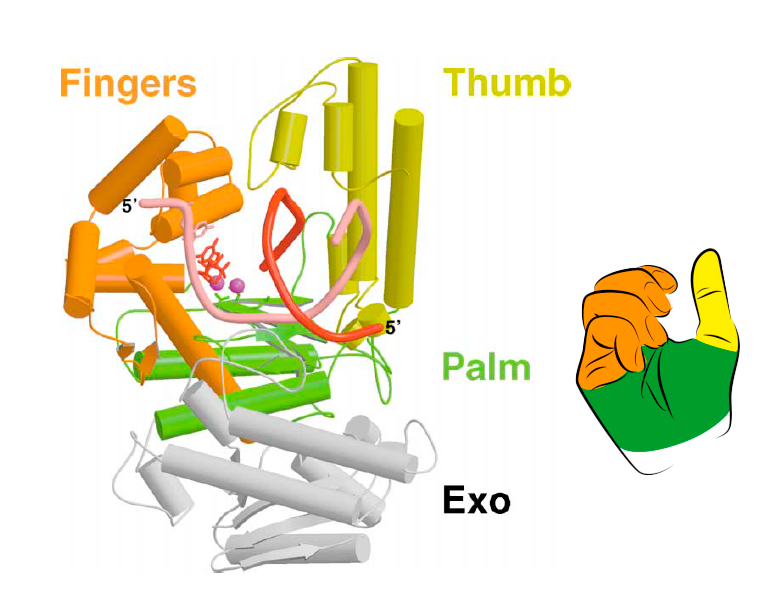
Рисунок 6. Доменная организация репликативной ДНК-полимеразы бактериофага T7. Несмотря на то, что это не эукариотический фермент, его структура хотя бы отдаленно напоминает кисть правой руки.
Но «обхват» у репликативных полимераз, стоит сказать, весьма специфический. Пространство, которое задается этими доменами, имеет ограниченную конфигурацию [16]. Такую, что фермент может лишь «запихнуть» в себя только комплементарные нуклеотиды (за редким исключением). А если все-таки происходит то самое редкое событие, то в их арсенале имеется еще один специальный домен, экзонуклеазный, распознающий и «откусывающий» неправильно вставленный нуклеотид. За этим механизмом самокоррекции (proofreading) стоит его 3’→5’ экзонуклеазная активность.
«Строгий» активный центр, обеспечивающий высокую селективность при включении дНТФ напротив матричного нуклеотида, 3’→5’-корректирующая активность, а также крайне высокая консервативность, обеспечивающая неизменность структуры в процессе эволюции, — «три кита», на которых держится высокоточная репликация генома практически любого организма. Однако, есть и другая сторона медали. Вместе с тем, эти же свойства, в первую очередь конформация активного сайта, не позволяют данным ферментам копировать поврежденные участки, вызывающие искажение двойной спирали ДНК. Они попросту не вписываются в ту геометрию, которая задается активным центром фермента. Кажется, настало время поговорить о тех самых загадочных «героях» нашей темы, «кузенах» репликативных полимераз — высокоспециализированных ферментах, транслезионных ДНК-полимеразах.
Виновники торжества
Итак, ДНК-полимеразы транслезионного синтеза, или просто транслезионные ДНК-полимеразы. На самом деле, их название весьма «говорящее», от англ. trans — через, lesion — повреждение. Что их делает такими уникальными? Да все тот же активный сайт [17]. Именно его особенности и позволяют транслезионной ДНК-полимеразе вставлять нуклеотид напротив повреждения. В процессе эволюции их активный сайт, как и структура в целом, претерпели некоторые изменения, благодаря чему ферменты и участвуют в ТЛС.
| Семейство | Представители | Особенности |
|---|---|---|
| A | Pol γ Pol θ Pol 𝜈 |
Pol γ — репликация митохондриальной ДНК. Pol θ и Pol 𝜈 - участие в репарации двухцепочечных разрывов ДНК, ТЛС. |
| B | Pol ɑ Pol δ Pol ε Pol ζ |
Высокоточные репликативные ДНК-полимеразы, копирующие ядерную ДНК. Pol ζ — транслезионная ДНК-полимераза. |
| X | Pol ꞵ Pol λ Pol ν TdT |
Ферменты репарации, участвующие в эксцизионной репарации оснований и репарации двухцепочечных разрывов; имеют важную роль в обеспечении многообразия антител в организме; ТЛС. |
| Y | Pol η Pol ɩ Pol κ Rev1 |
Высокоспециализированные транслезионные ДНК-полимеразы. |
| AEP | PrimPol | Праймаза-полимераза, участвующая в ТЛС и механизмах, связанных с преодолением блока репликативной вилки. |
Каноническими ДНК-полимеразами ТЛС считаются ферменты семейства Y, а также Pol ζ из семейства B, в которое, как вы могли заметить, входит большинство высокоточных репликативных ДНК-полимераз. Ферменты из семейства A (кроме Pol γ) и семейства X специализируются немного на других вещах, но также способны участвовать в ТЛС. По крайней мере, in vitro так уж точно. Кроме того, есть и другие семейства ДНК-полимераз. Например, семейство C, членами которого являются репликативные ДНК-полимеразы бактерий, а также семейство D, куда входят ферменты архей. Еще выделяют семейство RT, где «обитают» обратные транскриптазы. Однако мы сфокусируемся на эукариотических представителях семейств, представленных в таблице.
С классификацией вроде бы разобрались, теперь можно перейти к особенностям их активного центра, строение которого как раз-таки и заточено под прохождение полимеразой различных повреждений ДНК [18]. Поскольку вариантов повреждений очень много, то существует как минимум несколько ДНК-полимераз ТЛС, специализирующихся на их видах. Часто говорят, что их активный сайт обладает высокой «толерантностью» к повреждениям на матричной цепи ДНК.
Активный центр транслезионных ДНК-полимераз состоит из уже знакомых нам доменов Palm, Fingers и Thumb, выполняющих точно такие же функции, как и у репликативных «собратьев». Соответственно, общие черты «правой руки», ее архитектура, сохраняются. Тем не менее, существуют важные отличия. Так, количество контактов, которые образуют большой палец и пальцы транслезионных ДНК-полимераз с праймером и матричной цепью, соответственно, намного меньше, по сравнению с репликативными сородичами. За счет этого увеличивается «вместимость» активного сайта, что позволяет расположить в нем матричное повреждение [19]. Второе, не менее важное отличие, — наличие домена Little Finger (мизинец) у представителей семейства Y. Именно он и является тем самым важным фактором, определяющим узкую специализацию данных ферментов. Этот домен, который также называют polymerase-associated domain (PAD) важен тем, что образует дополнительные контакты с матрицей [20]. Таким образом, он помогает домену Fingers позиционировать матричный поврежденный нуклеотид в активном сайте фермента. Именно за счет этих свойств видоизмененный активный сайт и способен катализировать реакцию с поврежденными участками ДНК. Так или иначе, а как выглядит сам процесс ТЛС?
«Альтернативная» репликация
Ключевым событием, инициирующим непосредственно ТЛС, считается посттрансляционная модификация (а именно — убиквитинирование) белка PCNA (от англ. proliferating cell nuclear antigen, ядерный антиген пролиферирующих клеток) по лизину в положении 164, которое возникает в ответ на повреждение ДНК. PCNA, в первую очередь, представляет собой гомотример (т.е. состоит из трех идентичных между собой молекул). Вообще, это такой скользящий зажим, который, словно кольцо, обхватывает двухцепочечную спираль ДНК, благодаря которому репликативные полимеразы надежным образом связываются с матрицей (мы еще поговорим, почему это важно). Помимо этого, он является своеобразной «площадкой» для других молекул, работая в качестве молекулярного координатора [21]. На самом деле, его роль выходит далеко за пределы репликации и ТЛС, и об этом можно очень долго рассказывать, однако мы сфокусируемся на его функциях в ТЛС.
Само по себе убиквитинирование PCNA представляет собой очень сложно регулируемый механизм, зависящий от многих факторов: прохождения чекпоинтов клеточного цикла, и, соответственно, его стадии, типа повреждения ДНК, активации других путей, связанных с DTT, а также некоторых других факторов, требующих дальнейшего исследования. «Навешивание» убиквитина катализируется комплексом, состоящим из RAD18 и RAD6. Конечно, событиям привлечения RAD18/RAD6 и модификации PCNA предшествует целый каскад реакций, в котором участвует как минимум несколько белков, что уже немного выходит за рамки ТЛС [22].
Что же делает убиквитинированная форма PCNA? В первую очередь, она привлекает к застрявшей репликативной вилке наши таинственные ферменты. И на данном этапе их задача сводится к тому, чтобы поменяться местами с репликативными полимеразами. И вот тут возникают вопросы, как и при каких обстоятельствах это происходит. И меняются ли они вообще. В связи с этим было предложено несколько моделей, описывающих возможные сценарии, по которым дальше будет протекать ТЛС [22], [23].
Первая и, пожалуй, наиболее изученная называется моделью переключения полимераз (polymerase switching model). Нередко ее обозначают как двухполимеразную модель. Мы уже выяснили, что модифицированный PCNA вербует транслезионные полимеразы к репликативной вилке. Следующий шаг — смена полимераз. Считается, что первой в работу включается Rev1, которая сменяет репликативную полимеразу и связывается с PCNA. Rev1 снабжена множеством специальных регуляторных областей и сайтов связывания, опосредующих белок—белковые взаимодействия. Так, она служит неким «посредником», «хабом», если угодно, между PCNA и другими ДНК-полимеразами ТЛС, которые связываются уже с ней самой. Иными словами, «платформа» под названием Rev1, прежде всего, очень ценится за свои некаталитические функции. В конце концов, формируется комплекс, состоящий из регуляторных белков и нескольких транслезионных ДНК-полимераз — транслесома. И вот теперь модель начинает оправдывать оба своих названия.
Итак, задача первой полимеразы — вставить нуклеотид напротив повреждения. После того, как эта цель будет выполнена, происходит переключение на другую транслезионную полимеразу, которая удлиняет цепь уже после повреждения, вставляя некоторое количество (которое очень сильно варьирует) нуклеотидов напротив неповрежденных оснований матричной цепи. В соответствии с задачей, которую берет на себя та или иная ДНК-полимераза ТЛС, выделяют полимеразу-инсертер (первый этап, англ. insert — вставка, вставить) и полимеразу-экстендер (второй этап, англ. extend — удлинение, удлинять). По итогу все возвращается на круги своя, и репликативная ДНК-полимераза в составе реплисомы продолжает свое увлекательное путешествие, полное различных опасностей и препятствий.
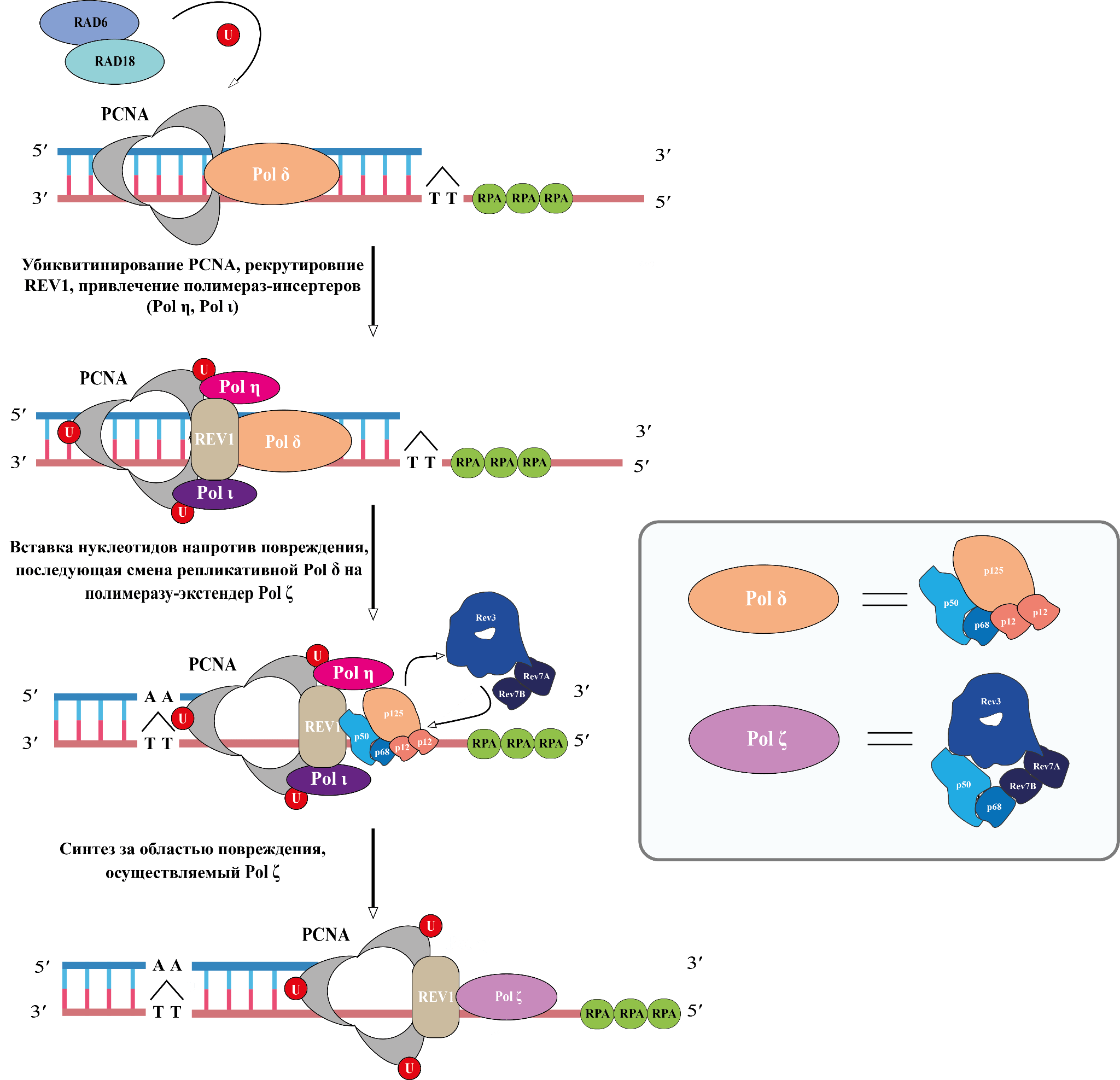
Рисунок 7. Модель переключения полимераз. Пояснение на иллюстрации и в тексте выше.
RPA — белок репликации A, стабилизирующий одноцепочечную ДНК. Интересная особенность: репликативная Pol δ и транслезионная Pol ζ как бы делят между собой несколько субъединиц; в момент удлинения праймера после повреждения каталитическая субъединица p125 и субъединицы p58 и p12 диссоциируют, а на их место приходят субъединицы Pol ζ.
адаптировано из [23]
Второй сценарий — смены между полимеразами не происходит. Точнее, она формально происходит, но если в первом случае репликативные полимеразы как бы «уступали» свои места, временно выходя из состава реплисомы, то в данной модели предполагается, что репликативные и высокоспециализированные полимеразы «сосуществуют» друг с другом внутри реплисомы. И как только они там все вместе уживаются? Выдвигается версия, что это все благодаря PCNA и его структуре, способной «приютить» все эти белки. Видимо, остальные этапы происходят аналогичным образом в соответствии с двухполимеразной моделью.
И, наконец, третья модель, построенная совершенно на иных принципах. Если вышеописанные модели сопряжены непосредственно с процессом репликации, происходящей в S фазу, то здесь развитие событий начинается непосредственно либо под самый ее конец, либо уже в фазу G2. Как только реплисома сталкивается с повреждением, она временно прекращает свою деятельность. Соответственно, напротив повреждения остается «дырка», а уже за ней, т.е. за областью повреждения, фермент PrimPol заново синтезирует праймер (помните, что это не только полимераза, но и праймаза? А в качестве «строительных кирпичиков» она использует не рибонуклеотиды, а дезоксинуклеотиды!), который репликативные полимеразы будут достраивать в составе возобновившей свою работу реплисомы. Такое явление получило название репрайминг [24]. А по окончании работы реплисомы (или когда она будет находиться далеко-далеко от сайта с повреждением) в эту область привлекаются транслезионные полимеразы, которые, образно говоря, «латают брешь» (отсюда и еt название — gap-filling model; русскоязычного аналога не встречал, можно перевести как «заполнение брешей»). Получается эдакая версия пострепликативной репарации. Тем не менее, и такой сюжет имеет место быть.
Конечно, остается очень много пробелов. Как происходит процесс in vivo? На обоих ли цепях могут функционировать ДНК-полимеразы ТЛС? Какая все-таки модель наиболее точно описывает данный процесс? И еще очень-очень много других, не менее важных вопросов, на которые хотелось бы получить ответы.
Выжить, но какой ценой?
Думаю, дорогой читатель задается вопросом: есть ли вообще какая-нибудь практическая значимость изучения данных ферментов? Допустим, ученые идентифицировали эти белки, проанализировали их структурные и биохимические свойства, а что дальше? Как это может помочь, например, в лечении болезней?
Изучением ТЛС и ассоциированных с ним молекул занимаются «представители» фундаментальной науки. Тем не менее, вопросы фундаментальной науки с течением времени могут плавно перетекать уже в задачи прикладной. Поэтому ответ — да, анализ ТЛС является важной задачей (на самом деле, такое утверждение будет справедливым для практически любой молекулы / метаболического пути / явления в целом)! С уверенностью могу сказать, что уже существуют определенные успехи в решении некоторых задач, поставленных перед исследователями. Однако, чтобы более подробно и точно ответить на вопрос, как все-таки изучение такого фундаментального процесса, как ТЛС, способно помочь нам, мы вернемся к транслезионным ДНК-полимеразам.
Как мы уже поняли, ТЛС — вовсе не альтернатива репаративным системам, а скорее крайне важное приложение к ним. Это совершенно иной метаболический путь, помогающий репликативной вилке преодолевать области ДНК с повреждениями. Однако порой бывает так, что активация ТЛС потом может дорого обойтись. Так какая же именно цена должна быть уплачена, чтобы клетка могла продолжить репликацию и выжить?
За измененный активный сайт, способный вмещать в себя различные повреждения, транслезионные ДНК-полимеразы «поплатились», прежде всего, двумя вещами [17–19]. И первая из них — точность. Под этим термином имеется в виду отношение частоты включения правильного нуклеотида к частоте вставки неправильного. Вторая — процессивность — означает такое количество нуклеотидов, которое полимераза способна добавить к растущей цепи праймера всякий раз, когда связывается с матричной цепью. Можно сказать, что этот показатель характеризует свойство сохранять сродство фермента с матрицей на протяжении всей репликации.
Репликативные ДНК-полимеразы обладают высокими значениями как точности, так и процессивности. Про особенности репликативных ДНК-полимераз, обеспечивающих их высокую точность, мы уже поговорили ранее. Что касается другого свойства, то за один раунд связывания с матрицей они способны добавить аж несколько сотен тысяч нуклеотидов! Такие высокие значения достигаются за счет следующих факторов:
- некаталитических субъединиц, обеспечивающих прочное связывание фермента с матрицей;
- клэмпа, плотно обхватывающего молекулы ДНК, словно зажим;
- уникальных доменов, осуществляющих дополнительное «сцепление» с матричной цепью (напр., домен P у дрожжевой Pol ε);
- дополнительных белков (RPA, RFC и др.).
Транслезионные полимеразы будут характеризоваться исключительно низкими показателями как точности, так и процессивности. Исключение составляет разве что Pol ζ, обладающая повышенной процессивностью по сравнению с ферментами семейства Y (но, так или иначе, в этом отношении ей все равно далеко до «родных братьев») [25]. Такое свойство делает из нее отличного кандидата на роль полимеразы-экстендера. Полимеразы ТЛС с низкой процессивностью, в свою очередь, хорошо подходят на роль инсертеров. Вот вроде бы и нашли «актеров» для театра под названием ТЛС. А что они попросят за свое участие?
Просить, конечно же, ничего не будут, однако от их «игры» есть крайне неприятные «побочные эффекты». Вместо того, чтобы препятствовать мутациям и поддерживать стабильность генома, они с немалой долей вероятности сами же могут им способствовать, нарушая и без того хрупкое геномное «равновесие». Причина, как вы, наверное, уже догадались, кроется в их крайне низкой точности. Таким образом, «герои» данной статьи превращаются в антигероев. Ферменты, призванные бороться с неприятностями, нередко сами их и создают. С другой стороны, а что будет бóльшим злом для клетки? Парочка мутаций на генном уровне (которые чаще всего представляют собой однонуклеотидные замены) или крупные хромосомные перестройки (а может даже и гибель клетки в результате незавершения репликации)? Очевидно, что последнее. Вообще, было бы неплохо иметь опцию «из двух зол не выбирать вовсе», но имеем то, что имеем.
За гранью ТЛС
На самом деле, функции транслезионных выходят сильно за пределы ТЛС. Они активно принимают участие в жизни клетки, занимая важные места в самых различных процессах, связанных с ДНК. И даже иногда такие их слабые стороны, как крайне низкая точность, способствующая более интенсивному накоплению мутаций, клетка способна использовать себе во благо.
Так, например, установлено, что практически все члены семейства Y ДНК-полимераз играют важную роль в процессе соматического гипермутагенеза генов иммуноглобулинов, лежащего в основе обеспечения разнообразия антител в организме. Вот здесь-то и пригодились их «умения», поскольку от них только и требуется, что «наделать» мутаций в вариабельных областях тяжелой и легкой цепей иммуноглобулинов в B-клетках иммунной системы [27–31].
Но список этим не ограничивается. Кроме того, многие из них принимают участие в копировании таких сложно реплицируемых областей (для репликативных полимераз), как ломкие сайты хромосом [32], [33]. Тут им приходится быть максимально осторожными, поскольку в этих областях, богатых на повторы, очень часто локализуются онкогены и онкосупрессорные гены.
Транслезионные полимеразы призваны помогать и на других последовательностях, таких как микросателлиты (которые, кстати, также характеризуются наличием повторов) [34–36]. Стоит отметить, что нестабильность этих областей может послужить причиной образования целого ряда неканонических форм ДНК: Z-ДНК, H-ДНК, G-квадруплексы и другие [37]. В репликации последних, кстати, ДНК-полимеразы ТЛС участвуют почти всем семейством Y [38–41].
Вышеперечисленные процессы требуют преимущественно лишь ДНК-полимеразную активность. Наличие дополнительных активностей (например, 5’-дезоксирибофосфатлиазной) и структурных особенностей предположительно позволяет участвовать им в таких репаративных процессах, как BER, c-NHEJ и alt-EJ [42–44]. Так или иначе, многие нерепликативные ДНК-полимеразы имеют в своем арсенале удивительные «способности», значительно расширяющие наше представление о них. Как видите, не такими уж и узкими специалистами, ограниченными лишь ТЛС, они оказались.
Дефекты «дефектных» полимераз
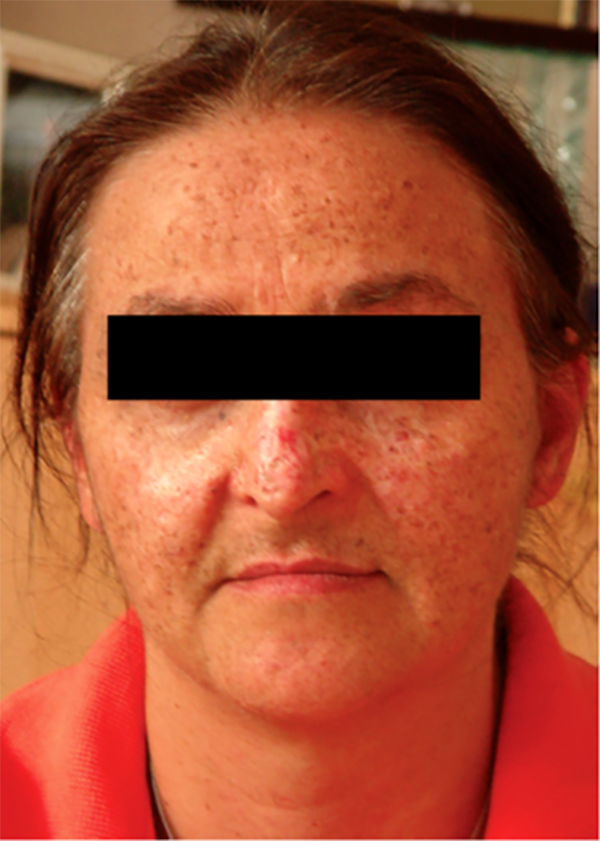
Рисунок 8. Пациент, пораженный пигментной ксеродермой. Пигментная ксеродерма (лат. Xeroderma pigmentosum) — аутосомно-рецессивное заболевание, характеризующееся повышенной чувствительностью к УФ, различными нарушениями со стороны кожи, а также высоким риском рака кожи. Примерно в 15% случаев наблюдается неврологическая симптоматика. Большинство фенотипов (XPA-XPG) связано с различными нарушениями в системе эксцизионной репарации нуклеотидов (ЭРО, NER). Тем не менее, существует другой фенотип — XPV (от англ. Xeroderma pigmentosum variant), развивающийся в результате нарушений в гене POLN. И хотя XPV впервые был описан еще 50 лет назад, молекулярные основы данного заболевания удалось понять только в 1999 году. В процессе ТЛС вместо неактивной или вовсе отсутствующей Pol η начинают задействоваться другие ДНК-полимеразы (предположительно это может быть Pol ι), точность которых напротив фотопродуктов намного ниже, что, в свою очередь, приводит к весьма быстрой аккумуляции мутаций.
Последствия мутаций и полиморфизмов в генах, кодирующих нерепликативные ДНК-полимеразы, в большинстве случаев нелетальны для млекопитающих. Так или иначе, здесь прослеживается зависимость от того, в каком именно гене появится нарушение и какую структурную область ДНК-полимеразы ТЛС оно затронет. Наиболее показательный пример — мутации гена POLN (кодирующего, соответственно, Pol η), приводящие к потере активности фермента. Поскольку основная функция данной полимеразы — прохождение фотопродуктов, возникающих в результате пагубного воздействия УФ на геном клеток кожи, защита организма от такого вида излучения резко падает [45–47]. Такое предраковое состояние называется пигментной ксеродермой [48].
Еще одна интересная взаимосвязь была недавно найдена между нарушениями в гене REV3L (кодирует каталитическую субъединицу Rev3L ДНК-полимеразы Pol ζ) и развитием синдрома Мёбиуса и болезни, характеризующейся задержкой развития с гипотрофией и рядом других проявлений [49], [50].
В теории, помимо рака и вышеперечисленных заболеваний, мутации транслезионных полимераз могут приводить к развитию некоторых нейродегенеративных заболеваний, в патогенезе которых одно из ключевых положений занимает явление под названием «экспансия тринуклеотидных повторов».
Почему так важно соблюдать баланс
Да, с одной стороны, плата за использование ТЛС высока, с другой — клетка выживает. Вроде бы можно и порадоваться за нее. Но точно не в случае ее трансформации в злокачественную в результате накопления критических мутаций в т.н. драйверных генах. Конечно, далеко не обязательно, что транслезионная ДНК-полимераза вставит некорректный нуклеотид напротив матричного, копируя часть именно такого гена. Тем не менее, от этого никто не застрахован, особенно в условиях изменения их активности и экспрессии.
Различные нарушения в регуляции экспрессии транслезионных ДНК-полимераз могут сильно осложнить нам жизнь. И заодно облегчить жизнь опухолевым клеткам. Когда клетка утрачивает контроль над экспрессией и активностью данных ферментов, это выливается в печальные последствия для организма. Ведь если они отвечают за прохождение повреждений в наших клетках (и некоторые другие функции, которые мы разобрали выше), то логично предположить, что как минимум то же самое они делают и в раковых. Рассмотрим несколько ситуаций:
- Недостаточная экспрессия в нормальных клетках. Вроде бы все понятно. Путь ТЛС будет меньше задействоваться, соответственно, клетка становится более чувствительной к ДНК-повреждающим агентам. В лучшем случае, в процессе репликации при столкновении с повреждением клетка отделается «легким испугом» в виде однонуклеотидной замены (другие ДНК-полимеразы ТЛС могут «подменить» слабо экспрессирующийся фермент, однако их специализация на других повреждениях не добавляет им точности), в худшем — получит крупные хромосомные перестройки или вовсе апоптирует.
- Чрезмерная экспрессия/активность в нормальных клетках. Логично, что это будет только способствовать мутагенезу в результате более частого использования транслезионных полимераз (которые, кстати, могут задействоваться, как мы выяснили ранее, и в процессе репликации неповрежденных участков ДНК), а значит, для нормальной клетки такую ситуацию точно не назвать хорошей.
- Чрезмерная экспрессия/активность в раковых клетках. В большинстве случаев это является благоприятной ситуацией для них. «Постойте, но почему это хорошо для опухолевой клетки? — поинтересуется читатель. — Ведь вы же сами написали, что использование данного пути сопряжено с возникновением мутаций». Именно! В таком случае раковая клетка убивает двух зайцев одним выстрелом: с одной стороны, это ведет к еще большей частоте возникновения мутаций (а такая изменчивость, как правило, ей только на руку), а с другой — обеспечивается резистентность, прежде всего, к таким широко использующимся химиотерапевтическим лекарствам, как препараты платинового ряда (цисплатин, оксалиплатин и др.). Пожалуй, вот и место, где можно «размахнуться» исследователям.
- Недостаточная экспрессия/активность в раковых клетках. На самом деле, данная ситуация весьма неоднозначна, поскольку далеко не всегда прослеживается взаимосвязь между этим явлением и изменением частоты мутагенеза и резистентности к химиопрепаратам именно в таких клетках. Нередко это зависит от того, какая именно ДНК-полимераза недостаточно экспрессируется.
Добиваемся гармонии
Очевидно, что если подавить гиперэкспрессию ДНК-полимераз ТЛС в раковых клетках, то можно добиться снижения их устойчивости к химиотерапевтическим препаратам. Вполне неплохая поддержка для базисной терапии, не так ли? Очень часто на роль таких ингибиторов рассматривают класс малых РНК, а точнее их подтип — микроРНК (miRNA), представляющие собой короткие некодирующие одноцепочечные РНК длиной 18–25 п.н. . Связываясь с последовательностью в 3’-нетранслируемой области целевой мРНК, она блокирует дальнейшую трансляцию продукта, в нашем случае — определенной ДНК-полимеразы [51].
Об огромном разнообразии РНК вы можете прочитать в работе «Обо всех РНК на свете, больших и малых» [52].
Конечно, это не единственный способ повлиять на экспрессию/активность «виновников» данной статьи. В качестве иной стратегии можно попробовать связать уже готовый продукт, т.е. сам фермент, тем самым нарушить его взаимодействие с транслесомой. В результате скрининга, методов in silico и экспериментов на клеточных культурах было выявлено несколько потенциальных низкомолекулярных соединений [53]. Еще одним перспективным направлением является синтез РНК- и ДНК-аптамеров , избирательно связывающих белки (и не только) [54].
Подробнее об аптамерах смотрите в комиксе «Аптамеры: графический гайд» [55].
На текущий момент соединения, влияющие на данные свойства полимераз, изучаются преимущественно in vitro, иными словами, проходят доклинические испытания . Тем не менее, по данным на конец 2020 года, первые клинические испытания для ингибиторов Pol θ уже должны были стартовать в 2021 году [56]. Более того, различные свойства ДНК-полимераз (например, уровень их экспрессии, наличие полиморфизмов) и вовсе могут иметь некоторую прогностическую ценность, важную для лечащих врачей [57], [58].
Подробнее о том, как разрабатываются терапевтические препараты, вы можете узнать из цикла статей, включенных в спецпроект «Доклиника».
Надеюсь, у меня получилось объяснить, почему изучение фундаментальных процессов и механизмов, согласно которым они протекают, является важным моментом. Конечно, на данном этапе пока очень рано говорить о клиническом применении вышеперечисленных соединений, поскольку эффективность многих из них была показана лишь in vitro. Более того, мы все прекрасно понимаем, что помимо ДНК-полимераз, участвующих в ТЛС, есть огромное количество других белков, нарушение регуляции которых может повлечь за собой еще более печальные последствия. Тем не менее, поиск, идентификация, разработка и модификация таргетных терапевтических средств, влияющих на активность и экспрессию ДНК-полимераз ТЛС представляет собой, может быть, и не самую важную цель на данный момент, но является весьма перспективным направлением, развитие которого способно спасти не одну жизнь.
Открытый финал
Так кто же все-таки транслезионные полимеразы? Те самые герои из комиксов, пожертвовавшие почти всем ради поддержания стабильности общества генома клетки? Или же коварные притаившиеся враги, только и ждущие, как бы нарушить ее? Конечно, так однобоко судить их мы не можем в силу различных обстоятельств. Впрочем, как и сам этот мир, так и эту жизнь, где нет четкого разделения на черное и белое, но где все имеет свою цену. Клетка, всякий раз активирующая путь транслезионного синтеза, нередко ценой мутаций (скольких?) получает возможность выжить. Равноценный ли это обмен? Лично я затрудняюсь ответить на данный вопрос. Наверное, для самой клетки это выгодная «сделка» здесь и сейчас. Для организма же в целом — однозначно сказать нельзя, покажет только время.
Литература
- А. V. Ignatov, K. A. Bondarenko, A. V. Makarova. (2017). Non-bulky Lesions in Human DNA: The Ways of Formation, Repair, and Replication. Acta Naturae. 9, 12-26;
- Биологическая машина репарации ДНК;
- Nimrat Chatterjee, Graham C. Walker. (2017). Mechanisms of DNA damage, repair, and mutagenesis. Environ. Mol. Mutagen.. 58, 235-263;
- Jean Cadet, Thierry Douki. (2018). Formation of UV-induced DNA damage contributing to skin cancer development. Photochem. Photobiol. Sci.. 17, 1816-1841;
- Nicole Hustedt, Daniel Durocher. (2017). The control of DNA repair by the cell cycle. Nat Cell Biol. 19, 1-9;
- D.L. Mitchell, C.A. Haipek, J.M. Clarkson. (1985). (6–4)Photoproducts are removed from the DNA of UV-irradiated mammalian cells more efficiently than cyclobutane pyrimidine dimers. Mutation Research Letters. 143, 109-112;
- Ghosal G., Chen J. (2013). DNA damage tolerance: a double-edged sword guarding the genome. Transl Cancer Res. 2, 107–129;
- 12 методов в картинках: генная инженерия. Часть II: инструменты и техники;
- M. Meselson, F. W. Stahl. (1958). The replication of DNA in Escherichia coli. Proceedings of the National Academy of Sciences. 44, 671-682;
- Youri I. Pavlov, Anna S. Zhuk, Elena I. Stepchenkova. (2020). DNA Polymerases at the Eukaryotic Replication Fork Thirty Years after: Connection to Cancer. Cancers. 12, 3489;
- Jessica L. Alexander, Terry L. Orr-Weaver. (2016). Replication fork instability and the consequences of fork collisions from rereplication. Genes Dev.. 30, 2241-2252;
- Ralph Scully, Rajula Elango, Arvind Panday, Nicholas A Willis. (2021). Recombination and restart at blocked replication forks. Current Opinion in Genetics & Development. 71, 154-162;
- Kenneth J. Marians. (2018). Lesion Bypass and the Reactivation of Stalled Replication Forks. Annu. Rev. Biochem.. 87, 217-238;
- Апоптоз, или Путь самурая;
- William A Beard, Samuel H Wilson. (2003). Structural Insights into the Origins of DNA Polymerase Fidelity. Structure. 11, 489-496;
- Wei Yang, Yang Gao. (2018). Translesion and Repair DNA Polymerases: Diverse Structure and Mechanism. Annu. Rev. Biochem.. 87, 239-261;
- Alexandra Vaisman, Roger Woodgate. (2017). Translesion DNA polymerases in eukaryotes: what makes them tick?. Critical Reviews in Biochemistry and Molecular Biology. 52, 274-303;
- M. F. Goodman, R. Woodgate. (2013). Translesion DNA Polymerases. Cold Spring Harbor Perspectives in Biology. 5, a010363-a010363;
- Julian E. Sale, Alan R. Lehmann, Roger Woodgate. (2012). Y-family DNA polymerases and their role in tolerance of cellular DNA damage. Nat Rev Mol Cell Biol. 13, 141-152;
- A. V. Makarova, A. V. Kulbachinskiy. (2012). Structure of human DNA polymerase iota and the mechanism of DNA synthesis. Biochemistry Moscow. 77, 547-561;
- E.M. Boehm, M.S. Gildenberg, M.T. Washington. (2016). The Many Roles of PCNA in Eukaryotic DNA Replication. DNA Replication Across Taxa. 231-254;
- Domenico Maiorano, Jana El Etri, Camille Franchet, Jean-Sébastien Hoffmann. (2021). Translesion Synthesis or Repair by Specialized DNA Polymerases Limits Excessive Genomic Instability upon Replication Stress. IJMS. 22, 3924;
- Xiaolu Ma, Tie‐Shan Tang, Caixia Guo. (2020). Regulation of translesion DNA synthesis in mammalian cells. Environ Mol Mutagen. 61, 680-692;
- Elizaveta Boldinova, Paulina Wanrooij, Evgeniy Shilkin, Sjoerd Wanrooij, Alena Makarova. (2017). DNA Damage Tolerance by Eukaryotic DNA Polymerase and Primase PrimPol. IJMS. 18, 1584;
- Alena V. Makarova, Peter M. Burgers. (2015). Eukaryotic DNA polymerase ζ. DNA Repair. 29, 47-55;
- Agnès Tissier, John P. McDonald, Ekaterina G. Frank, Roger Woodgate. (2000). polι, a remarkably error-prone human DNA polymerase. Genes Dev.. 14, 1642-1650;
- Andrew Franklin, Peter J Milburn, Robert V Blanden, Edward J Steele. (2004). Human DNA polymerase‐η, an A‐T mutator in somatic hypermutation of rearranged immunoglobulin genes, is a reverse transcriptase. Immunol Cell Biol. 82, 219-225;
- Leticia K. Lerner, Thuy V. Nguyen, Ligia P. Castro, Juliana B. Vilar, Veridiana Munford, et. al.. (2020). Large deletions in immunoglobulin genes are associated with a sustained absence of DNA Polymerase η. Sci Rep. 10;
- Ahmad Faili, Anne Stary, Frédéric Delbos, Sandra Weller, Said Aoufouchi, et. al.. (2009). A Backup Role of DNA Polymerase κ in Ig Gene Hypermutation Only Takes Place in the Complete Absence of DNA Polymerase η. J Immunol. 182, 6353-6359;
- Anna-Laura Ross, Julian E. Sale. (2006). The catalytic activity of REV1 is employed during immunoglobulin gene diversification in DT40. Molecular Immunology. 43, 1587-1594;
- Jacob G. Jansen, Petra Langerak, Anastasia Tsaalbi-Shtylik, Paul van den Berk, Heinz Jacobs, Niels de Wind. (2006). Strand-biased defect in C/G transversions in hypermutating immunoglobulin genes in Rev1-deficient mice. Journal of Experimental Medicine. 203, 319-323;
- Ryan P. Barnes, Suzanne E. Hile, Marietta Y. Lee, Kristin A. Eckert. (2017). DNA polymerases eta and kappa exchange with the polymerase delta holoenzyme to complete common fragile site synthesis. DNA Repair. 57, 1-11;
- Valérie Bergoglio, Anne-Sophie Boyer, Erin Walsh, Valeria Naim, Gaëlle Legube, et. al.. (2013). DNA synthesis by Pol η promotes fragile site stability by preventing under-replicated DNA in mitosis. Journal of Cell Biology. 201, 395-408;
- Suzanne E. Hile, Xiaoxiao Wang, Marietta Y. W. T. Lee, Kristin A. Eckert. (2012). Beyond translesion synthesis: polymerase κ fidelity as a potential determinant of microsatellite stability. Nucleic Acids Research. 40, 1636-1647;
- Suzanne E. Hile, Kristin A. Eckert. (2008). DNA polymerase kappa produces interrupted mutations and displays polar pausing within mononucleotide microsatellite sequences. Nucleic Acids Research. 36, 688-696;
- M Dixon. (2002). Examining the potential role of DNA polymerases η and ζ in triplet repeat instability in yeast. DNA Repair. 1, 763-770;
- Левозакрученная: загадочная Z-ДНК;
- Sarah Eddy, Magdalena Tillman, Leena Maddukuri, Amit Ketkar, Maroof K. Zafar, Robert L. Eoff. (2016). Human Translesion Polymerase κ Exhibits Enhanced Activity and Reduced Fidelity Two Nucleotides from G-Quadruplex DNA. Biochemistry. 55, 5218-5229;
- Davide Schiavone, Guillaume Guilbaud, Pierre Murat, Charikleia Papadopoulou, Peter Sarkies, et. al.. (2014). Determinants of G quadruplex‐induced epigenetic instability in REV 1‐deficient cells. EMBO J. 33, 2507-2520;
- Sarah Eddy, Amit Ketkar, Maroof K. Zafar, Leena Maddukuri, Jeong-Yun Choi, Robert L. Eoff. (2014). Human Rev1 polymerase disrupts G-quadruplex DNA. Nucleic Acids Research. 42, 3272-3285;
- Rujuta Yashodhan Gadgil, Eric J. Romer, Caitlin C. Goodman, S.Dean Rider, French J. Damewood, et. al.. (2020). Replication stress at microsatellites causes DNA double-strand breaks and break-induced replication. Journal of Biological Chemistry. 295, 15378-15397;
- Katarzyna Bebenek, Agnès Tissier, Ekaterina G. Frank, John P. McDonald, Rajendra Prasad, et. al.. (2001). 5'-Deoxyribose Phosphate Lyase Activity of Human DNA Polymerase ɩ in Vitro. Science. 291, 2156-2159;
- Dheva Setiaputra, Daniel Durocher. (2019). Shieldin – the protector of DNA ends. EMBO Rep. 20;
- Alessandra Brambati, Raymond Mario Barry, Agnel Sfeir. (2020). DNA polymerase theta (Polθ) – an error-prone polymerase necessary for genome stability. Current Opinion in Genetics & Development. 60, 119-126;
- C. Masutani. (2000). Mechanisms of accurate translesion synthesis by human DNA polymerase eta. The EMBO Journal. 19, 3100-3109;
- A HENDEL, O ZIV, Q GUERANGER, N GEACINTOV, Z LIVNEH. (2008). Reduced efficiency and increased mutagenicity of translesion DNA synthesis across a TT cyclobutane pyrimidine dimer, but not a TT 6-4 photoproduct, in human cells lacking DNA polymerase η. DNA Repair. 7, 1636-1646;
- Robert E. Johnson, Lajos Haracska, Satya Prakash, Louise Prakash. (2001). Role of DNA Polymerase η in the Bypass of a (6-4) TT Photoproduct. Mol Cell Biol. 21, 3558-3563;
- Alexei Gratchev, Pamela Strein, Jochen Utikal, Sergij Goerdt. (2003). Molecular genetics of Xeroderma pigmentosum variant. Experimental Dermatology. 12, 529-536;
- Laura Tomas-Roca, Anastasia Tsaalbi-Shtylik, Jacob G. Jansen, Manvendra K. Singh, Jonathan A. Epstein, et. al.. (2015). De novo mutations in PLXND1 and REV3L cause Möbius syndrome. Nat Commun. 6;
- Agnieszka Halas, Jolanta Fijak-Moskal, Renata Kuberska, Victor Murcia Pienkowski, Aneta Kaniak-Golik, et. al.. (2021). Developmental delay with hypotrophy associated with homozygous functionally relevant REV3L variant. J Mol Med. 99, 415-423;
- Priyanka Saha, Tanima Mandal, Anupam D. Talukdar, Deepak Kumar, Sanjay Kumar, et. al.. (2021). DNA polymerase eta: A potential pharmacological target for cancer therapy. J Cell Physiol. 236, 4106-4120;
- Обо всех РНК на свете, больших и малых;
- Anna Schrempf, Jana Slyskova, Joanna I. Loizou. (2021). Targeting the DNA Repair Enzyme Polymerase θ in Cancer Therapy. Trends in Cancer. 7, 98-111;
- Andrei V. Lakhin, Andrei A. Kazakov, Alena V. Makarova, Yuri I. Pavlov, Anna S. Efremova, et. al.. (2012). Isolation and Characterization of High Affinity Aptamers Against DNA Polymerase Iota. Nucleic Acid Therapeutics. 22, 49-57;
- Аптамеры: графический гайд;
- Anna Schrempf, Jana Slyskova, Joanna I. Loizou. (2021). Targeting the DNA Repair Enzyme Polymerase θ in Cancer Therapy. Trends in Cancer. 7, 98-111;
- Srdana Grgurevic, Patricia Montilla-Perez, Alice Bradbury, Julia Gilhodes, Sophie Queille, et. al.. (2018). DNA polymerase ν gene expression influences fludarabine resistance in chronic lymphocytic leukemia independently of p53 status. Haematologica. 103, 1038-1046;
- Roberta Taiane G. de Oliveira, Ivo Gabriel. F. França, Howard L.R. Junior, Giovanna B.C. Riello, Daniela de Paula Borges, et. al.. (2021). c.9253-6T > c REV3L: A novel marker of poor prognosis in Myelodysplastic syndrome. Hematology, Transfusion and Cell Therapy. 43, 377-381.