
Синтетическая биология: от программирования компьютеров к программированию клеток
09 октября 2018
Синтетическая биология: от программирования компьютеров к программированию клеток
- 7126
- 1
- 13
Синтетическая биология — новая научная парадигма, главная цель которой — совершить прорыв в создании новых и модифицировании существующих биологических машин. Автор фото: Ivan Morozov (Virginia Bioinformatics Institute)
-
Автор
-
Редактор
Темы
Статья на конкурс «био/мол/текст»: Биологические исследования всё больше привлекают специалистов из области компьютерных наук. И это не удивительно: ДНК — носитель информации о живых организмах, — для программиста как программный код. Абстракция, используемая в программировании, позволяет работать с объектами, не принимая во внимание особенности их реализации. Такой подход к биологическим объектам оказался весьма перспективным: он значительно упрощает реальное положение дел и при этом позволяет элементарным составляющим функционировать с высокой точностью. Применение главных инженерных принципов — абстракции, стандартизации и автоматизации, делает из биоинженерии настоящую инженерную дисциплину и открывает широкие возможности для создания новых биологических машин. Так появилась синтетическая биология — новое междисциплинарное научное направление, объединяющее генных инженеров, молекулярных биологов, программистов и физиков, одна из главных задач которой — создавать новые биологические машины для решения практических задач.
Конкурс «био/мол/текст»-2018
Эта работа опубликована в номинации «Свободная тема» конкурса «био/мол/текст»-2018.

Генеральный спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

Спонсором приза зрительских симпатий выступил медико-генетический центр Genotek.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
ДНК глазами программиста
Сегодня биологические исследования всё больше привлекают людей из computer science. Для них ДНК — это программный код, а живые организмы — исполнители программ, и вызывают ту или иную функцию в зависимости от полученного сигнала. По их мнению, ДНК в живых клетках может быть описана, понята и изучена, используя термины программирования. Действительно, давайте взглянем на ДНК глазами программиста [1]:
- язык ДНК — цифровой, использует четыре значения: A, T, C и G; «ДНК-байт» состоит их трех знаков и кодирует одну аминокислоту;
- у эукариот часть «кода» — последовательности интронов, — на самом деле ничего не кодирует и в последствии вырезается. Тем не менее они выполняют важнейшую функцию: эта ДНК помогает правильно исполнять код, подобно комментариям в программах;
- универсальность генетического кода делает генетические программы совместимыми с подавляющим большинством живых организмов;
- многие регуляторные сети в живых организмах могут быть описаны с помощью логических выражений. Для примера, вспомним всем известную регуляторную систему E. coli — лактозный оперон. В присутствии глюкозы экспрессия генов оперона подавлена, в отсутствии глюкозы — включена. Используя логические операторы, то же самое можно было бы записать иначе: НЕ(А) = Б, где А — глюкоза, а Б — экспрессия генов лактозного оперона.
Возможно ли применение инженерных принципов к биологическим объектам?
Описывая лактозный оперон с помощью логических выражений, мы абстрагировались от его реализации в клетке, так как нас интересовал только входной и выходной сигналы. Именно абстракция позволяет программистам строить и контролировать системы высокого уровня сложности (рис. 1). Абстрагируясь от некоторых конкретных деталей объекта, можно создать модель, достаточно точно описывающую поведение системы на определенном уровне.
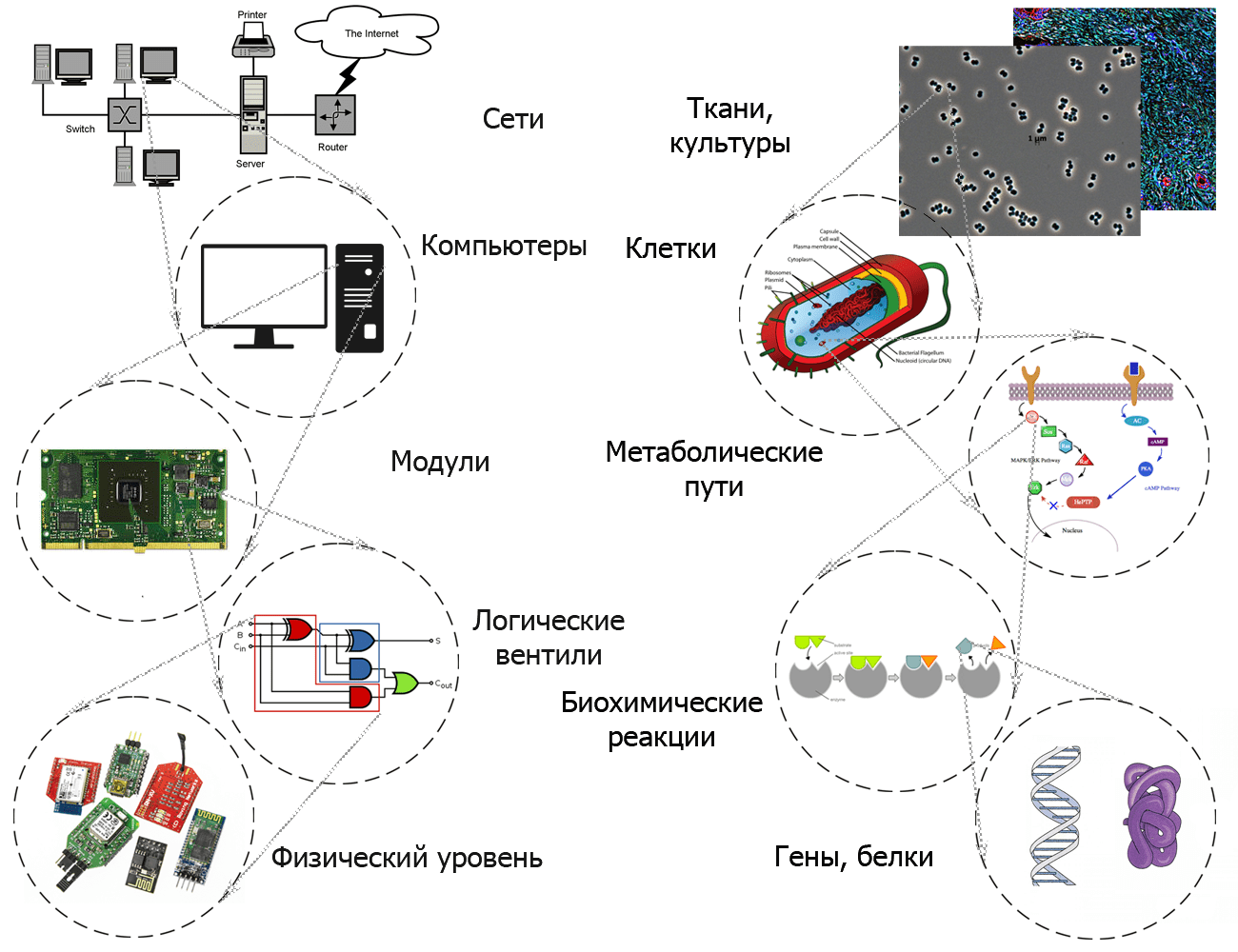
Рисунок 1. Аналогия между иерархической абстракцией в электроинженерии и синтетической биологии
«Википедия», рисунок адаптирован
Еще один важный подход, оказавшийся весьма полезным применительно к биологическим системам — дискретизация сигналов. Живые системы отвечают на изменения окружающей среды сложным комплексом реакций. Сигналы, поступающие из окружения, могут быть классифицированы на цифровые (поведение «включить—выключить») и аналоговые (постепенное повышение концентрации, температуры и т.д.). Синтетические биологи создают специальные устройства, преобразующие аналоговые сигналы в цифровые (например, генетические переключатели с пороговым значением), потому что обработку цифровых сигналов запрограммировать проще. В электронной инженерии дискретизация сигнала позволила сделать технику более защищенной от шума и избавиться от путаницы, возникающей при передаче непрерывных сигналов, что послужило мощным толчком в развитии техники — сегодня цифровые устройства широко применяют во многих сферах жизни. На подобный прорыв в синтетической биологии рассчитывают дизайнеры генетических сетей. Упрощение компонентной базы до простых, но робастных (устойчивых к помехам) элементов, позволяет реализовать сложные системы, способные работать при высоком уровне шума, характерном для биологических объектов.
Проблемы на пути синтетических биологов
Разумеется, живые организмы, даже такие относительно простые как прокариоты, далеки от функционирования на уровне цифровых устройств. Дрю Энди, профессор биоинженерии Стэнфордского университета, видит две главные проблемы, преграждающие путь биоинженерам к созданию систем высокого уровня сложности [2]. Во-первых, это высокая сложность биологических объектов, во-вторых — их недостаточная изученность. Свойства компонентов биологических систем сильно зависят от контекста. Например, сила сайта связывания рибосомы зависит как от вышележащих, так и от нижележащих последовательностей, причем они могут как усиливать (за счет взаимодействия с регуляторными белками и повышения аффинности к сайту связывания), так и ослаблять (при образовании вторичных структур, препятствующие связыванию мРНК с рибосомой) силу связывания рибосомы с мРНК. Компоненты биологических систем связаны многочисленными взаимодействиями (вспомним, к примеру, про плейотропное действие генов), что делает сложным применение редукционистского подхода к биологическим системам. Уникальные свойства многокомпонентных биологических систем не определяются свойствами отдельно взятых ее составляющих. Работая в едином ансамбле, они способны выполнять функции, существование которых, рассматривая только составляющие системы и связи между ними, предсказать практически невозможно из-за сложного переплетения связей. Недостаток знаний о процессах, происходящих на молекулярном уровне в клетке, лимитирует создание более сложных систем. По этим причинам, использование существующих генетических элементов или синтезированных конструкций и внедрение их в живые системы — задача далеко не тривиальная.
Главная проблема, возникающая при дизайне генетических сетей — перекрестное взаимодействие между естественными клеточными системами и искусственно встроенными [3]. Возникающие помехи мешают функционированию синтезированной системы по задуманной программе. Возможное решение — создание ортогональных систем, то есть систем, не влияющих на работу существующих клеточных компонентов. Так, в 2010 году английским исследователям удалось расширить генетический код, добавив в клетку ортогональную систему трансляции [4].
В чем разница между синтетической биологией и генной инженерией?
Таким образом из синтеза инженерии и биологии появилось новое научное направление — синтетическая биология. Эта междисциплинарная наука объединяет молекулярных биологов, химиков, физиков и инженеров для достижения общих целей. На первый взгляд, синтетическая биология мало отличается от генетической инженерии. Обе науки бросают вызов эволюции и ставят своей целью создание новых биологических систем. Где же проходит граница между этими двумя близкими дисциплинами?
Главное отличие синтетической биологии от генетической инженерии заключается в том, что генетическая инженерия — это набор методов, а синтетическая биология — это подход к выполнению задачи. Биоинженеры используют ПЦР для создания мутантов и амплификации ДНК [5]; метод рекомбинантных ДНК для создания новых комбинаций генов [6]; секвенирование для чтения последовательностей [7]. Синтетическая биология дополняет молекулярные основы генетической инженерии и предлагает применить инженерные принципы при решении задач биоинженерии. Один из создателей синтетической биологии, Дрю Энди, выделяет следующие принципы [2], лежащие в основе синтетической биологии — автоматизация, стандартизация, абстракция и декаплинг (видео 1).
Видео 1. Дрю Энди, профессор Стэнфордского университета, объясняет разницу между генетической инженерией и синтетической биологией
Еще про инженерные принципы
Автоматизация
Облачные лаборатории, такие как Emerald Cloud Lab (рис. 2), предлагают полностью автоматизировать работу мокрых биологов. Выбирая готовый протокол из предложенных на сайте, ученые задают последовательность операций, которую необходимо выполнить с образцами. После этого полностью роботизированная система исполняет все команды и исследователям остается только проанализировать полученные результаты. Такой подход поможет справиться с кризисом воспроизводимости и освободит руки исследователей для работы над задачами, требующими творческого подхода.

Рисунок 2. Роботизированная система из облачной лаборатории Emerald Cloud Laboratory. Полная автоматизация лабораторной работы позволит увеличить воспроизводимость полученных результатов и значительно сократить время на выполнение научных экспериментов.
Абстракция
Сложность биологических систем, упомянутая ранее, не позволяет сделать рутинным процессом конструирование многокомпонентных биологических систем, способных вести себя согласно задумке дизайнера. Широко используемый инструмент для манипуляции со сложными системами — абстракция. Информация, описывающая биологические системы, должна быть распределена по нескольким уровням, подчиняющимся иерархии (рис. 3). Чтобы такой подход имел смысл, эта иерархия уровней должна сделать возможным рассмотрение каждого отдельного уровня вне контекста вышележащих и нижележащих уровней, и связь между уровнями абстракции должна быть обеспечена передачей информации.
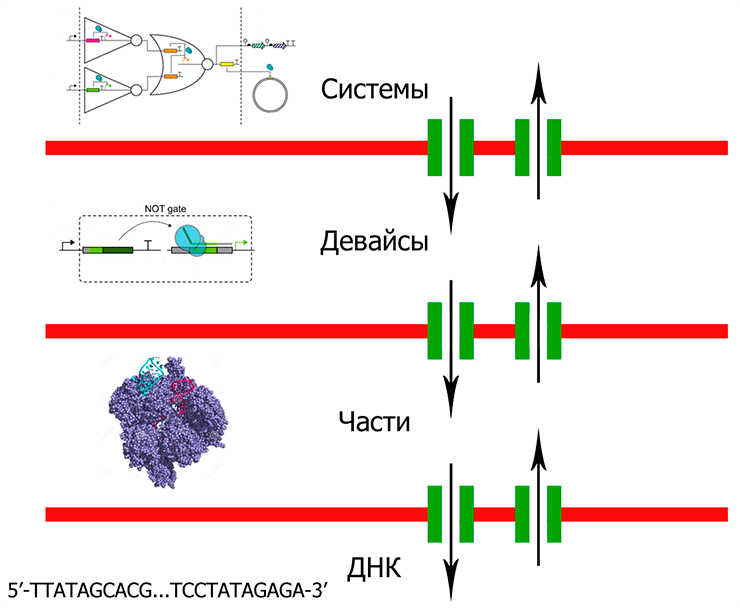
Рисунок 3. Схема, иллюстрирующая способ представления инженерных генетических сетей с помощью уровней абстракции, подчиненных определенной иерархии. Цель подобного представления — упрощение работы со сложными биологическими системами. Здесь уровни абстракции — «ДНК», «Части» (молекулы, выполняющие базовые биологические функции: ДНК-связывающие белки и другие), «Девайсы» (любые комбинации «частей», выполняющие функции, определенные дизайном) и «Системы» (любые комбинации «девайсов»). Барьеры (красные блоки) разделяют уровни абстракции. Связь между уровнями обеспечивается интерфейсами (зеленые блоки). Подобная система должна обеспечить независимую работу на каждом уровне. Исследователь, работающий на уровне «частей», должен знать, какие части потребуются исследователю на уровне «девайсов», как именно эти части работают (например, взаимодействия между аминокислотами и большой бороздкой ДНК) и как можно заказать необходимую последовательность ДНК. Но этому же исследователю вовсе не обязательно знать что-либо о фосфорамидитной химии, о том, как короткие олигонуклеотиды собираются в длинные последовательности ДНК или что-либо о работе генетических осцилляторов.
[2], рисунок адаптирован
Декаплинг
В биоинженерии одни и те же специалисты продумывают дизайн генетической конструкции и осуществляют сборку биологических систем. В любой инженерной дисциплине сборка и дизайн разделены между разными специалистами. Например, в архитектуре строители не проектируют здания, а инженеры не работают на стройке. Одна из задач синтетической биологии — разделить сборку генетических сетей и их дизайн. Требуются профессионалы, которые будут заниматься исключительно созданием базовых элементов генетических сетей для сборки более сложных композиций, и исследователи, специализирующиеся исключительно на дизайне новых биологических систем.
Стандартизация
В молекулярной биологии уже существуют стандарты для некоторых широкоиспользуемых типов данных. Например, общепринятые стандарты хранения и обмена данными экспрессии генов, последовательностей ДНК, биологических моделей. Стандартизация позволяет экономить время при исследовании и повышать воспроизводимость результатов исследований. Однако не существует стандартов для большинства классов биологических функций (например, для активности промотора), экспериментальных измерений, условий эксплуатации штаммов бактерий (параметров окружающей среды, скорости роста, питательной среды и так далее). Синтетическая биология ставит перед собой задачу создать четкие правила, руководствуясь которыми биологи смогли бы повысить надежность синтетических систем и воспроизводимость экспериментов по их созданию. Для упрощения рутинных операций биоинженерии, таких как проведение реакций рестрикции-лигирования, Том Найт, именуемый отцом синтетической биологии, создал первый широкоиспользуемый стандарт в синтетической биологии — стандарт BioBricks [8].
Стандарт BioBricks
Это способ стандартизовать компоненты, используемые при сборке более масштабных конструкций. Каждый «биокирпичик» состоит из основы (backbone), префикса, суффикса и кодирующей части и представляет собой небольшую плазмиду. В префиксе и суффиксе содержатся по два сайта узнавания рестриктаз-изокаудамеров. Такие рестриктазы узнают разные сайты, но образуют совместимые липкие концы. После лигирования образуется гибридный сайт, не узнаваемый ни одной из рестриктаз — так исключается возможность неправильной последовательности сборки, что значительно упрощает процесс создания рекомбинантов. Кирпичики, содержащие составные части системы (например, промотор или сайт связывания рибосомы), называют вектором ввода, а итоговую плазмиду, готовую к трансфекции или трансформации, — вектором экспрессии.
iGEM & Registry of standard biological parts
Трудами многих исследователей была создана библиотека стандартных биологических частей (Registry of standard biological parts). В ней в открытом доступе хранятся последовательности ДНК созданных элементов и вся информация, доступная о них. В библиотеке на сегодняшний день зарегистрировано уже более 20 000 частей и с каждым годом их количество пополняется участниками конкурса iGEM (The International Genetically Engineered Machine Competition). Цель конкурса — привлечь больше молодых исследователей к работе над проектами в области синтетической биологии. Один из прошлогодних проектов-победителей — синтетическая система бактериального чувства кворума, которая может стать звеном в целой искусственной экосистеме [9].
Простейшие устройства: логические вентили, триггер, осциллятор
Применение инженерных принципов и дискретизации биологических сигналов обещает возможность биоинженерам создавать более сложные системы, с высокой точностью исполняющие свои функции. Для создания многокомпонентных систем необходимо существование четко работающей элементной базы (рис. 4). Обработка цифровых сигналов происходит с помощью логических операций, многие из которых (буфер (повторитель), И, ИЛИ, НЕ (инвертор) и другие) уже имеют свое физическое воплощение в виде регуляторных контуров (английский термин genetic circuit более наглядно демонстрирует аналогию с электрическими сетями), созданных из последовательности ДНК.
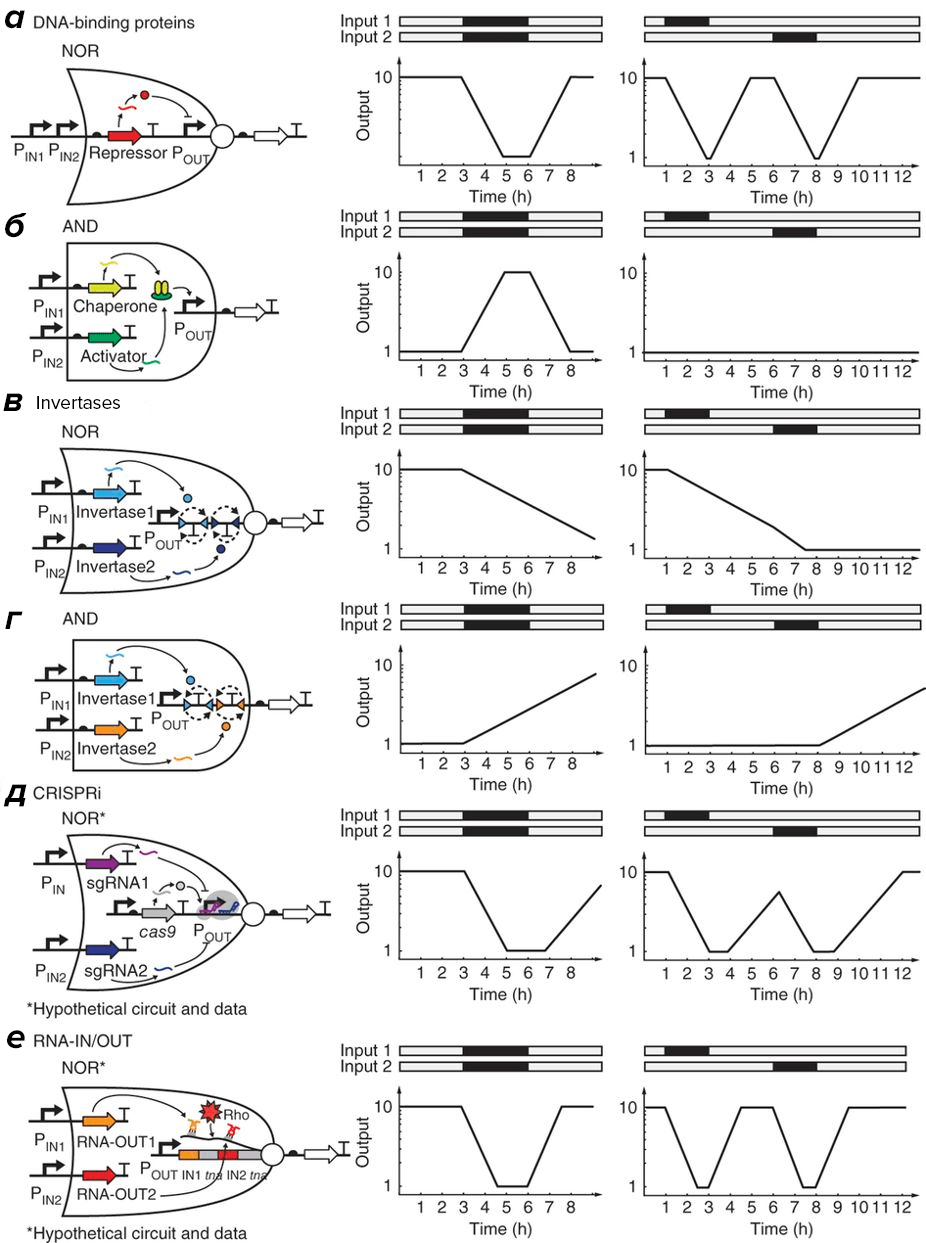
Рисунок 4. Примеры возможной реализации логических вентилей, используемых при дизайне генетических сетей Слева — схемы генетических сетей, задействующие ДНК-связывающие белки (а и б), инвертазы (в и г), CRISPRi (д) и малые некодирующие РНК (RNA-IN и RNA-OUT) (е) при работе логических вентилей. Справа — графики, демонстрирующие поведение системы при одновременном и последовательном поступлении двух сигналов.
Из простейших элементов были созданы более сложные устройства (рис. 5). В начале 2000-х Джеймс Коллинз и Тим Гарднер стали создателями первого искусственного генетического триггера, обладающего поведением, сходным с триггером в электроинженерии [10]. Он представлял собой систему из двух генов (ген A и ген B), репрессирующих друг друга. При этом за синтезом одного из репрессоров следовал синтез флуоресцентного белка, для возможности отследить извне динамику системы. После краткой индукции система переходила из первого состояния (ген А включен, ген B выключен) во второе (ген B включен, ген А выключен), что сопровождалось исчезновением или появлением сигнала. Такая система обладает памятью: после исчезновения воздействия индуцирующего сигнала система сохраняет способность поддерживать свое состояние.
Чуть позже Майкл Эловиц и его коллега Станислав Лейберг сконструировали первый осциллятор [11], представляющий собой систему из трех генов, связанных между собой петлями связи. Продукт первого гена подавляет действие второго, второй — третьего, и, замыкая круг, продукт третьего гена репрессирует экспрессию первого. Концентрации трех белков представляли собой гармонические колебания с заданными значениями амплитуды. Осцилляция детектировалась с помощью репортерной плазмиды с геном GFP.
Несмотря на то, что идея для создания биологических аналогов осциллятора и триггера пришла из электронной инженерии, за основу воплощения на уровне ДНК этих устройств были взяты системы, реально существующие в природе. Каркасом для осциллятора стали биологические часы цианобактерий, а для триггера — система бактериофага лямбда, ответственная за вступление бактериофага в литический цикл.
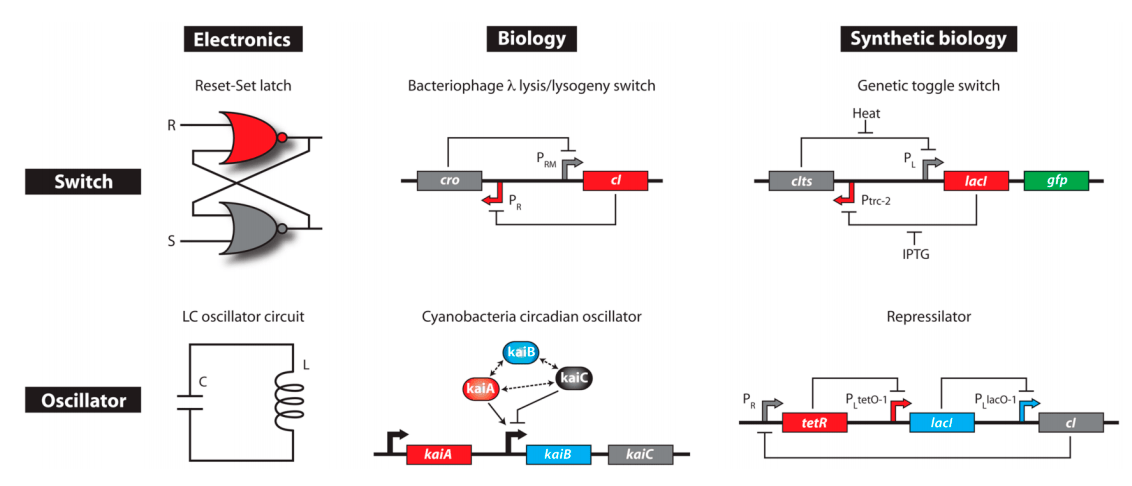
Рисунок 5. Простейшие устройства электроинженерии — переключатель (минимальное запоминающее устройство) и осциллятор (генератор ритма), их аналоги в естественных системах и схемы искусственных генетических сетей
Сегодня прогресс в синтетической биологии достиг такого уровня, что ученые трудятся над конструированием инженерных тканей и органов, а особо амбициозные специалисты замахнулись на создание живых организмов de novo с полностью искусственно синтезированным геномом [12]. Одной из главных проблем остается создание сложных систем, принимающих на вход много сигналов. Из-за высокого уровня шума и сложных связей, объединяющих клеточные компоненты, предсказать поведение системы, состоящей из большого числа элементов, практически невозможно. Перспективные направления, которые позволят решить эту проблему — системы на основе РНК—РНК взаимодействий и CRISPR. Использование малых РНК в качестве механизма передачи сигнала позволяет нивелировать задержки, типичные для систем, основанных на взаимодействии ДНК—белок, и повышает точность клеточных «вычислений». Другое немаловажное преимущество — возможность применения одних и тех же регуляторных элементов как в прокариотических, так и в эукариотических клетках. Самая большая на сегодняшний день генетическая конструкция, способная функционировать in vivo, создана из нескольких взаимосвязанных РНК-переключателей и принимает на вход 12 сигналов [13].
Методы синтетической биологии
Задачи синтетической биологии включают в себя сборку масштабных конструкций из ДНК. На сегодняшний день самая длинная последовательность ДНК, синтезированная в лаборатории — собранный de novo геном Mycoplasma mycoides [14], [15]. Размер искусственно синтезированной последовательности — более 1 000 000 пар оснований. Если бы сборку такой масштабной конструкции, состоящей из большого числа отдельных последовательностей, проводили с помощью реакций рестрикции и лигирования, возможно, мы бы никогда не дождались первого искусственно синтезированного генома. Чтобы собрать по кусочкам геном микоплазмы, потребовалось изобрести новый метод сборки. Даниэль Гибсон, исследователь из Института Крейга Вентера, назвал его «метод изотермической рекомбинации in vitro в один шаг» (one step isothermal in vitro recombination method) [16], но все называют этот метод по имени его создателя — сборкой по Гибсону. Метод основывается на амплификации фрагментов с помощью ПЦР. При этом на концы последовательностей добавляются по нескольку десятков пар нуклеотидов от соседних фрагментов. После амплификации следует рекомбинация фрагментов и на выходе получается молекула ДНК, сшитая из нескольких кусочков. Преимущество данного метода состоит в том, что в одной пробирке одновременно можно соединить сразу большое число фрагментов, что значительно повышает скорость создания масштабных конструкций. Существует еще несколько методов, основанных на этом же принципе: SLIC и MoClo.
Технология получила свое дальнейшее развитие: Гибсон и его команда создали машину, позволяющую осуществить «биологическую телепортацию» (видео 2). Им удалось автоматизировать синтез последовательностей ДНК, РНК и белков. Передавая информацию о последовательности ДНК, на выходе можно получать запрограммированные живые бактерии. Возможное применение новой технологии — создание планет, пригодных для жизни, путем заселения модифицирующими их микроорганизмами.
Видео 2. Дэн Гибсон, создатель одного из методов высокопроизводительного клонирования, рассказывает о «биологической телепортации» и возможных перспективах синтетической биологии
Синтетическая биология: потенциальное применение
Как уже было сказано раньше, упрощение компонентной базы до простых, но робастных элементов открывает широкие возможности для дизайна сложно устроенных генетических сетей. Благодаря этому, синтетическая биология имеет широкое применение: от создания биокомпьютеров до управления дифференцировкой стволовых клеток (рис. 6).
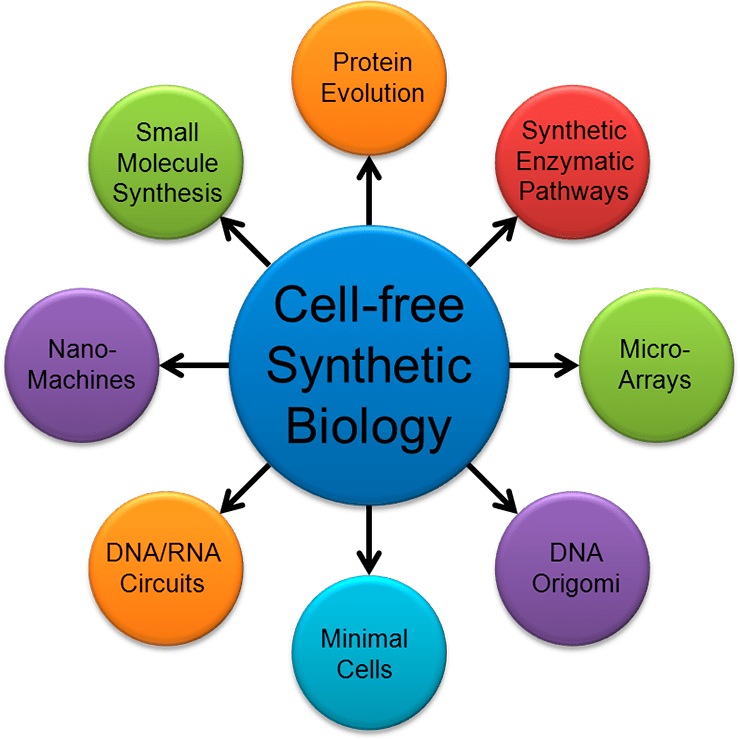
Рисунок 6. Потенциальное применение синтетической биологии
Искусственный морфогенез
Потенциал применения человеческих ИПСК (индуцированных плюрипотентных стволовых клеток) велик. Сегодня они главным образом используются как модельные объекты для скрининга лекарственных препаратов, изучения болезней и влияния токсичных веществ, но кроме этого, ведутся работы по применению ИПСК в клеточной терапии и создании искусственных органов.
Процесс перепрограммирования ИПСК в определенную клеточную линию — дорогостоящий и требующий длительного времени процесс. Большинство протоколов представляют собой обработку клеточных культур коктейлем из транскрипционных факторов, состав которого определяется экспериментально. Но даже при правильном подборе необходимых транскрипционных факторов, верном определении порядка добавления каждого их них и времени их воздействия, эффективность подобной процедуры не велика.
Альтернативный вариант — перепрограммирование клеток путем внедрения генетической информации о синтезе факторов транскрипции, необходимых для детерминации пути развития клетки (рис. 7). Подобным путем удалось воспроизвести достаточно сложные профили экспрессии. Например, с помощью РНК-сети, внедренной в ИПСК, были созданы инсулинсекретирующие бета-подобные клетки [17]. В развитии этого типа клеток ключевую роль играют три фактора: Ngn3, Pdx1 и MafA. Начало экспрессии и продолжительность синтеза регулировались при помощи нескольких регуляторных механизмов. В результате удалось воспроизвести уровень экспрессии генов транскрипционных факторов на протяжении всего периода дифференцировки и получить бета-подобные клетки.
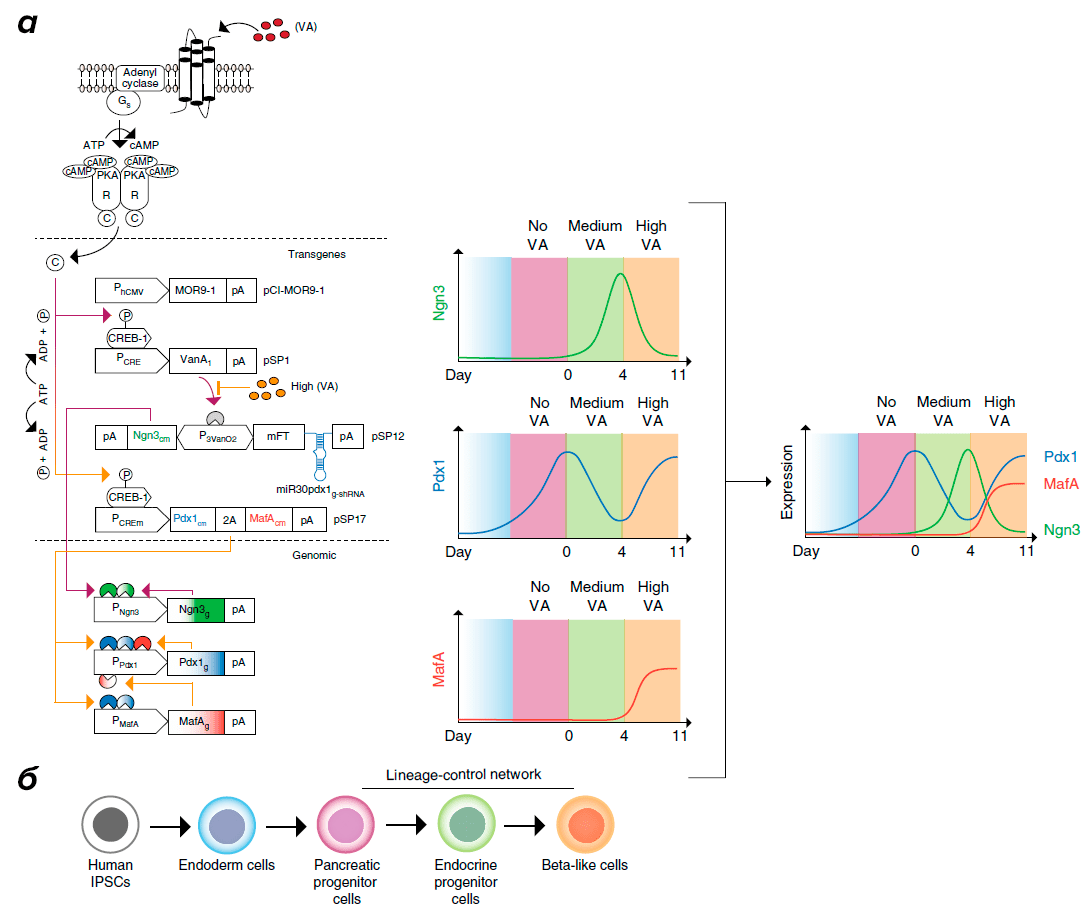
Рисунок 7. Управление дифференцировкой клеток с помощью генетических сетей. Слева — схема генетической сети, основанной на РНК и регуляторных белках, внедрением которой в клетки удалось направить развитие человеческих ИПСК в бета-подобные инсулинчувствительные клетки. Весь цикл развития занимает 11 дней, в регуляции развития участвуют три транскрипционных фактора: Pdx1, MafA и Ngn3. Справа — паттерны синтеза факторов транскрипции, участвующих в регуляции транскрипции. Внизу — схема последовательных превращений человеческих ИПСК в бета-подобные клетки.
Биосенсоры
Способность биосенсоров быстро и с высокой точностью детектировать широкий спектр молекул делает их незаменимыми в решении индустриальных, медицинских, экологических и научных задач. С совершенствованием дизайна генетических сетей появляется все больше вариантов сенсоров с самыми разными параметрами и условиями функционирования. Биосенсоры могут использоваться при контроле производства пищевых продуктов, качества воды и проверки почвы на контаминацию. В медицинских исследованиях подобные биосенсоры могут заменить целую лабораторию в полевых условиях и оказаться ценным инструментом, позволяющим детектировать наличие патогена. Так в прошлом году метод с затейливым названием SHERLOCK поразил всех своей способностью детектировать ничтожно малые концентрации патогена и выявлять различия геномов с точностью до одного нуклеотида [18].
Лекарства
Малярия до сих пор остается одним из опаснейших заболеваний и ежегодно уносит десятки тысяч жизней. Китайский фармаколог Ту Юю (Tu Youyou) (рис. 8, слева) в поисках противомалярийного препарата обратилась к средствам народной медицины. Ее поиски увенчались успехом: был открыт артемизинин, выделенный из полыни однолетней [19]. Тогда возникла новая проблема — доставка необходимого лекарственного препарата начала зависеть от всходов полыни и в случае неурожайного года препарат, спасающий жизни, оказывался в недостатке. Альтернативу выделению экстракта из полыни предложил Джей Кислинг (Jay Keasling) (рис. 8, справа) — профессор химической инженерии и биоинженерии Калифорнийского университета в Беркли. Его команде удалось с нуля создать метаболический путь синтеза предшественника артемизинина в клетках дрожжей. Таким образом был создан дополнительный источник этого лекарственного препарата, который может стать основным в случае необходимости.

Рисунок 8. Исследователи, внесшие наибольший вклад в разработку противомалярийного препарата — артемизинина. Слева: Ту Юю — китайский фармаколог, лауреат Нобелевской премии по физиологии и медицине (2015). Изучая народную китайскую медицину, открыла артемизинин — препарат из полыни однолетней (Artemesia annua), помогающий в борьбе с малярией. Справа: Джей Д. Кислинг — изобрел альтернативный способ получения предшественника артемизинина, создав новый метаболический путь в дрожжах.
Биокомпьютеры
В отличие от привычных нам компьютеров, биокомпьютеры потребляют удивительно мало энергии, что делает их чрезвычайно экономически выгодными вычислительными устройствами. Были созданы компьютеры на основе бактерий, которые способны выполнять простейшие логические операции, такие как логическое сложение, умножение и вычитание. Альтернатива клеточным системам — ДНК-компьютеры, способные выполнять свои функции вне клеток прямо в пробирке (рис. 9). Принцип работы таких систем основан на свойствах молекулы ДНК: в цепи ДНК закодирована информация в виде последовательности нуклеотидов, которую можно изменять с помощью ферментов. Таким образом, с помощью ДНК-компьютеров можно хранить и обрабатывать информацию. Потенциальное применение биокомпьютеров — встраивание вычислительных систем в человеческий организм и использование в генной терапии для детекции заболеваний и их лечения.
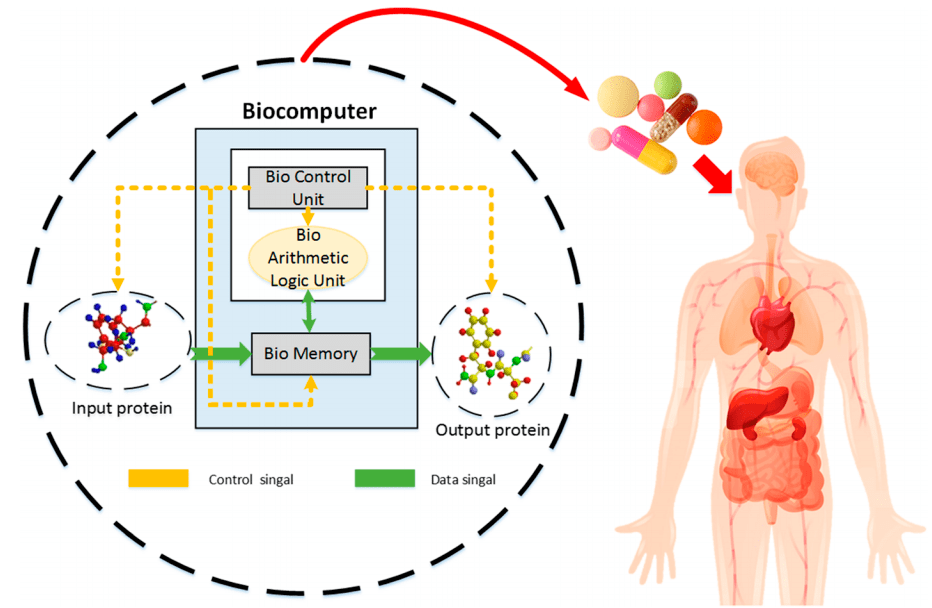
Рисунок 9. Потенциальное применение биокомпьютеров в лечении заболеваний: модуль памяти хранит информацию о перенесенных заболеваниях, вычислительный модуль по концентрациям определенных молекул (например, антигенов) оценивает состояние пациента и в случае необходимости запускает лечение.
Биотопливо
За счет возможности быстро нарастать, биомасса стала потенциальным альтернативным источником топлива. Для повышения содержания богатых энергией веществ в биомассе оптимальные условия жизни микроорганизмов сменяются на экстремальные, при которых в качестве защиты начинается накопление масел. Синтетические биологи разрабатывают альтернативу привычному способу получения биотоплива — перепрограммирование микроорганизмов путем изменения метаболических путей для создания штаммов, которые быстрее делятся и накапливают значительно больше богатых энергией соединений [20]. Объекты модификации — хорошо изученная кишечная палочка, продемонстрировавшая хорошую способность к экспрессии чужеродных генов, и многочисленные штаммы водорослей, которые, несмотря на недостаточную изученность устройства их генетического аппарата, остаются одними из самых многообещающих продуцентов биотоплива, поскольку для их роста требуется поразительно мало ресурсов.
Что дальше?
Новые задачи требуют новых подходов. За свое недолгое время существования синтетическая биология смогла доказать, что применение инженерных принципов к работе с биологическими объектами позволит эффективней находить решение задач из области биотехнологии, медицины и фармацевтики, а также повлечет за собой прогресс в области биоинженерии. Возможно, уже в скором будущем программирование живых организмов перестанет удивлять кого-либо, а биокомпьютеры достигнут такого уровня развития, что станут способными выполнять сложные программы.
Литература
- Hubert B. (2017). DNA seen through the eyes of a coder. Сайт ds9a.nl;
- Drew Endy. (2005). Foundations for engineering biology. Nature. 438, 449-453;
- Jennifer A N Brophy, Christopher A Voigt. (2014). Principles of genetic circuit design. Nat Methods. 11, 508-520;
- Слово из четырёх букв;
- 12 методов в картинках: полимеразная цепная реакция;
- 12 методов в картинках: генная инженерия. Часть II: инструменты и техники;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Reshma P Shetty, Drew Endy, Thomas F Knight. (2008). Engineering BioBrick vectors from BioBrick parts. J Biol Eng. 2, 5;
- Синтетическая биология: от наблюдения к вмешательству;
- Timothy S. Gardner, Charles R. Cantor, James J. Collins. (2000). Construction of a genetic toggle switch in Escherichia coli. Nature. 403, 339-342;
- Michael B. Elowitz, Stanislas Leibler. (2000). A synthetic oscillatory network of transcriptional regulators. Nature. 403, 335-338;
- Жизнь в эпоху синтетической жизни;
- Jongmin Kim, Peng Yin, Alexander A. Green. (2018). Ribocomputing: Cellular Logic Computation Using RNA Devices. Biochemistry. 57, 883-885;
- D. G. Gibson, G. A. Benders, C. Andrews-Pfannkoch, E. A. Denisova, H. Baden-Tillson, et. al.. (2008). Complete Chemical Synthesis, Assembly, and Cloning of a Mycoplasma genitalium Genome. Science. 319, 1215-1220;
- Синтетическая жизнь;
- Daniel G Gibson, Lei Young, Ray-Yuan Chuang, J Craig Venter, Clyde A Hutchison, Hamilton O Smith. (2009). Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6, 343-345;
- Pratik Saxena, Boon Chin Heng, Peng Bai, Marc Folcher, Henryk Zulewski, Martin Fussenegger. (2016). A programmable synthetic lineage-control network that differentiates human IPSCs into glucose-sensitive insulin-secreting beta-like cells. Nat Comms. 7, 11247;
- SHERLOCK — молекулярный сыщик спешит на помощь!;
- Названы лауреаты Нобелевской премии-2015 по физиологии и медицине;
- Michael S Ferry, Jeff Hasty, Natalie A Cookson. (2012). Synthetic biology approaches to biofuel production. Biofuels. 3, 9-12;
- Ahmad S. Khalil, James J. Collins. (2010). Synthetic biology: applications come of age. Nat Rev Genet. 11, 367-379;
- Chun-Liang Lin, Ting-Yu Kuo, Wei-Xian Li. (2018). Synthesis of control unit for future biocomputer. J Biol Eng. 12.







