Компьютерные технологии против коронавируса: первые результаты
13 апреля 2020
Компьютерные технологии против коронавируса: первые результаты
- 3329
- 2
- 5
Новый коронавирус застал человечество врасплох, но к борьбе с ним подключились новые технологии: на тропу войны вышли хемоинформатика и искусственный интеллект. На фото — вирионы SARS-CoV-2, выходящие из зараженной клетки.
-
Авторы
-
Редакторы
Коронавирусы находятся в списке опасных патогенов с начала XXI в. В 2002 г. коронавирус вызвал эпидемию тяжелого острого респираторного синдрома (англ. severe acute respiratory syndrome, SARS), а в 2013 г. — ближневосточный респираторный синдром (англ. Middle East respiratory syndrome, MERS). В конце 2019 г. в Китае началась новая вспышка коронавирусной инфекции (англ. coronavirus disease, COVID-19), которая застала человечество врасплох. Вирус SARS-CoV-2, отличающийся довольно высокой инфицирующей способностью и смертностью, перекинулся на другие страны, и 11 марта ВОЗ объявила вспышку COVID-19 пандемией. В связи с этим перед учеными встала ключевая задача: в кратчайшие сроки разработать способы лечения и профилактики. На помощь в этом приходят передовые компьютерные технологии — молекулярное моделирование, виртуальный скрининг и искусственный интеллект. В продолжение ставшей уже такой популярной на «Биомолекуле» темы SARS-CoV-2 мы выкладываем (с некоторыми изменениями и дополнениями) исходно опубликованный в «Природе» обзор предварительных результатов разработки лекарственных препаратов против нового коронавируса с акцентом на применение компьютерных технологий.
С начала 2020 года весь мир следит за распространением коронавируса, который в декабре 2019-го вызвал эпидемию в Китае, а затем перекинулся и на другие страны. Поскольку эпидемия охватила весь мир , 30 января Всемирная организация здравоохранения объявила вспышку нового коронавируса (который поначалу именовался 2019-nCoV) чрезвычайной ситуацией в области общественного здравоохранения, имеющей международное значение, а 11 марта — пандемией . Серьезность ситуации требует искать решение проблемы всеми возможными путями .
Эпидемиологическая обстановка в мире быстро меняется. За актуальностью информации следит Университетский центр системных исследований и инженерии имени Джонса Хопкинса, практически в реальном времени выкладывая статистику на GitHub, на основе которой создана визуальная панель мониторинга распространения SARS-CoV-2.
«Биомолекула» также пристально следит за развитием ситуации: «Хроника распространения SARS-CoV-2» [1].
Помимо хроники, на «Биомолекуле» опубликовано еще несколько статей про новый коронавирус, которые уже снискали большую (и, увы, печальную) популярность: «2019-nCoV: очередной коронованный убийца?» [2] и дилогия «COVID-19: отвечаем на вопросы» [3] и «COVID-19: что мы знаем и чего не знаем» [4].
Китайские исследователи оперативно расшифровали и охарактеризовали геном нового коронавируса, обнаружив большое сходство с вирусом SARS-CoV (аббревиатура от англ. severe acute respiratory syndrome — коронавирус тяжелого острого респираторного синдрома), который был причиной эпидемии в 2002–2003 гг., а также с двумя коронавирусами летучих мышей, что стало основанием считать их природным резервуаром нового коронавируса, переименованного, соответственно, в SARS-CoV-2 [5–7]. Еще до публикации этих результатов его нуклеотидная последовательность была депонирована в базу данных GenBank. К 8 апреля на этом и подобных порталах собрали уже 566 геномов пандемического коронавируса из образцов, полученных в разных странах. Геномные исследования SARS-CoV-2 позволяют разрабатывать тесты для его выявления, строить филогенетические деревья, чтобы проследить его происхождение [66] и эволюцию и т.д. Кроме того, исследователи изучают 3D-структуры белков, механизмы инфицирования, ищут подходы для создания вакцин и лекарств — моноклональных антител и малых молекул. ВОЗ ведет базу данных публикаций и размещает на своем сайте новости по «противокоронавирусным» исследованиям.
Все результаты исследований незамедлительно публикуются в открытом доступе на сайтах научных журналов (Nature, Science, The Lancet, Journal of Medical Virology) и препринтов (bioRxiv, medRxiv и chemRxiv). За последние два месяца только на bioRxiv опубликовано более 80 статей о самых разных аспектах изучения SARS-CoV-2 и борьбы с ним. Важно подчеркнуть, что на всех сервисах размещено объявление о том, что статьи не прошли рецензирование и не должны рассматриваться как окончательные выводы, рекомендации по лечению или профилактике, а также не должны освещаться в СМИ как доказанная информация. Мы поддерживаем эти предостережения и призываем рассматривать нашу публикацию как научно-просветительский материал, изложенные в нем данные как предварительные и требующие дополнительного подтверждения, а сведения по лечению и профилактике COVID-2019 черпать только из официальных рекомендаций.
Откуда взялся новый коронавирус?
Впервые коронавирус человека выделили в 1965 г. из больного ОРВИ, а вскоре выяснилось, что коронавирусов много, и циркулируют они не только среди людей, но и среди домашних и диких животных [8]. Коронавирусы не считались опасными патогенами человека до тех пор, пока в 2002 г. мир не столкнулся с эпидемией SARS, которая началась в Китае и распространилась на 37 стран, поразив более 8 тыс. человек и погубив 774 (смертность более 9%) [9], [10]. В 2012 г. в Саудовской Аравии вспыхнула вторая эпидемия коронавируса, связанного с ближневосточным респираторным синдромом (англ. Middle-East respiratory syndrome, MERS), которым переболели почти 2,5 тыс. человек, 858 из которых погибли (смертность более 34%) [8]. После этих двух эпидемий стало ясно, что коронавирусы способны преодолевать межвидовые барьеры и переходить от животных к людям, а также что они могут представлять для людей серьезную опасность [11]. Эксперты предупреждали, что рано или поздно может возникнуть новая вспышка [12]. Это и случилось в 2019 г. — и снова в Китае, но никто не ожидал, что она охватит весь мир, то есть дорастет до размеров пандемии.
Во всех трех случаях естественным резервуаром коронавирусам послужили летучие мыши. Известно, что SARS-CoV изначально циркулировал среди подковоносых летучих мышей (Rhinolophus sinicus), а на человека перекинулся от небольшого лазающего млекопитающего из отряда хищных — малайской пальмовой циветты (Paradoxurus hermaphroditus) [13].
MERS-CoV также возник среди летучих мышей, а на пути передачи к человеку оказались одногорбые верблюды (Camelus dromedarius). Промежуточный хозяин SARS-CoV-2 пока точно не установлен. В конце февраля вышло несколько препринтов, в которых говорилось о довольно большом генетическом сходстве (85,5–92,4%) коронавирусов яванского панголина (Manis javanica) с вирусом, вызывающим COVID-19 [14]. Однако этого явно недостаточно, чтобы уверенно обвинить панголина (например, SARS-CoV имел 99,8% общих с коронавирусом циветты последовательностей, поэтому это животное стали считать источником вспышки «атипичной пневмонии»).
Интенсивные исследования нового коронавируса позволили Международному комитету по таксономии вирусов определить место SARS-CoV-2: его отнесли к роду Betacoronavirus семейства Coronaviridae отряда Nidovirales царства Riboviria (рис. 1) [7].
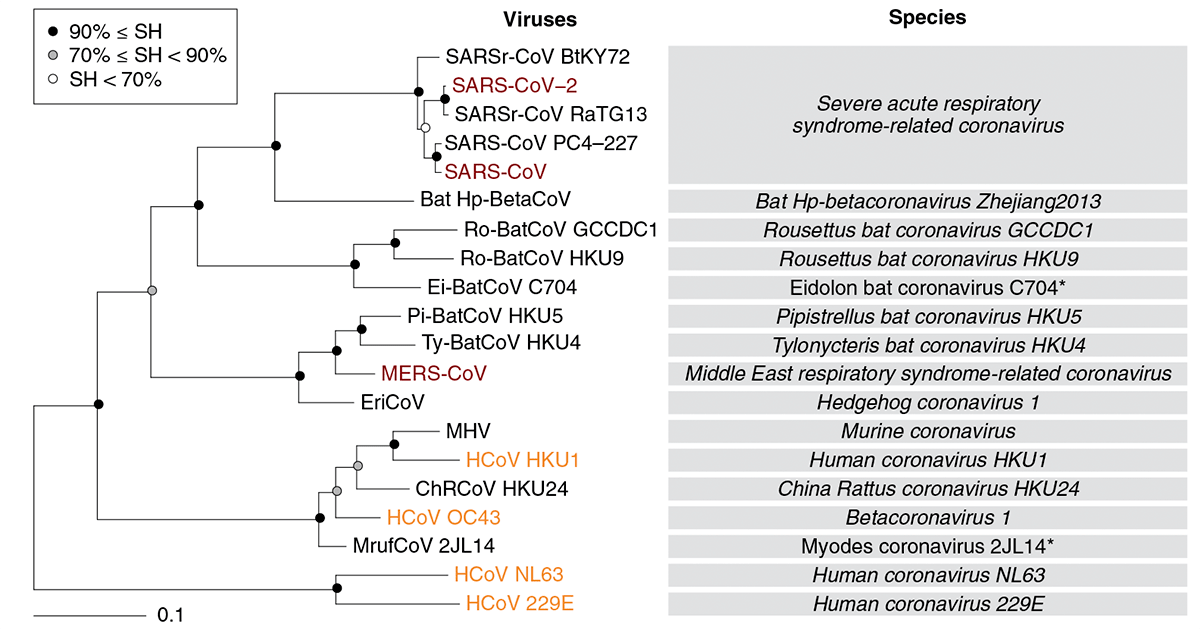
Рисунок 1. Филогенетическое древо коронавирусов, циркулирующих среди летучих мышей или людей, в том числе пяти видов рода Betacoronavirus, связанных с тяжелым острым респираторным синдромом. Красным цветом выделены зоонозные вирусы, проявившие патогенность к людям; коричневым — вирусы, циркулирующие среди людей и вызывающие у них незначительные респираторные проявления. Звездочками обозначены два вида коронавирусов, демаркация и названия которых ожидают одобрения Международного комитета по таксономии вирусов, так что они не выделены курсивом. SH — достоверность ветви, определенная по тесту Симодайра—Хасэгава (Shimodaira—Hasegawa).
Как и у всех представителей Coronaviridae, вирион SARS-CoV-2 обрамляют булавовидные шипы гликопротеина, которые придают оболочке вируса сходство с солнечной короной (отсюда название), что хорошо заметно на электронной микрофотографии (рис. 2а). Этот поверхностный гликопротеин, или S-белок (от англ. spike — шип), отвечает за проникновение вируса в клетку путем имитации молекул, на которые реагируют трансмембранные рецепторы клеток. Помимо липидной оболочки, в которую заякорены еще два структурных белка — E (Envelope, оболочка) и М (Membrane), — у коронавируса есть внутренний нуклеокапсид из N-белка (Nucleocapsid), внутри которого скрыт геном вируса (плюс-цепь РНК) (рис. 2б).
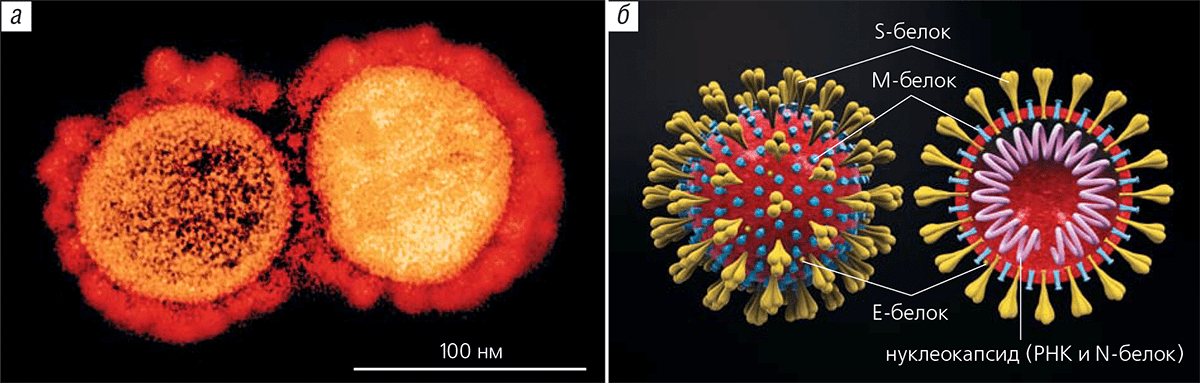
Рисунок 2. Электронная микрофотография зрелых вирионов SARS-CoV-2 (а) и его 3D-модель (б)
Центры по контролю и профилактике заболеваний и Национальный институт аллергологии и инфекционных заболеваний США
SARS-CoV отличался от своих сородичей тем, что его «шипы» связываются с АПФ2 — ангиотензинпревращающим ферментом 2 (англ. angiotensin converting enzyme 2, ACE2), — «конкурентом» известного АПФ, являющегося мишенью лекарств для снижения давления. В то время как АПФ превращает неактивный ангиотензин I в повышающий давление ангиотензин II, АПФ2 расщепляет ангиотензин II до снижающего давление ангиотензина 1-7, тем самым уравновешивая действие АПФ. АПФ находится в крови, а АПФ2 – на мембранах клеток легких. Умение связываться с ним, по-видимому, и придает вирусу такую высокую способность вызывать пневмонию [8]. MERS-CoV пошел иным путем, нежели его «старший брат» из Китая, — S-белок MERS-CoV связывается с дипептидилпептидазой-4 (англ. dipeptidyl-peptidase, DPP4) [15], которая расщепляет инкретиновые гормоны и служит терапевтической мишенью для лечения сахарного диабета II типа. Помимо легких, DPP4 в большом количестве содержится в почках — поэтому MERS-CoV вызывает пневмонию с почечной недостаточностью одновременно [8].
В отличие от MERS-CoV, SARS-CoV-2 пошел по проторенной дорожке: его «шипы», как и у SARS-CoV, связываются с ACE2 клетки хозяина [16]. По геномной последовательности SARS-CoV-2 — близкий родственник SARS-CoV, но отличается от него и MERS-CoV меньшей смертностью (к нашему счастью), хотя и гораздо большей заразностью. Это и позволило ему распространиться гораздо шире, чем в две предыдущие коронавирусные эпидемии. Очевидно, что против SARS-CoV-2 нужны специфические лекарства.
Как найти лекарство?
Несмотря на 15-летнюю историю контакта с опасными коронавирусами и ожидание новых вспышек, человечество не успело разработать специфического противокоронавирусного средства [17]. Даже более или менее испытанных терапевтических стратегий мало. Во время прежних вспышек применяли антивирусные препараты широкого действия рибавирин и интерферон-альфа [10]. В случае вирусов это означает, что эффект не гарантирован, а побочные действия не вполне предсказуемы. Впрочем, рибавирин продемонстрировал эффективность против SARS-CoV [18], но нужны более крупные и тщательные исследования .
Речь идет о клинических исследованиях, с помощью которых строго доказывают эффективность и безопасность всех лекарственных средств, прежде чем применять их на практике. Как это делается, можно прочесть в спецпроекте о клинических исследованиях.
На животных, зараженных SARS-CoV и MERS-CoV, показал эффективность другой противовирусный препарат широкого спектра — ремдесивир, который изначально был разработан для лечения лихорадки Эбола. О старте первых клинических исследований ремдесивира объявили в конце февраля Национальные институты здоровья США с планируемой датой окончания к 2023 году; однако сейчас таких исследований зарегистрировано уже 10, и первые результаты вскоре должны появиться. Неплохо себя зарекомендовал и другой противовирусный препарат широкого спектра действия — фавипиравир (рис. 3).
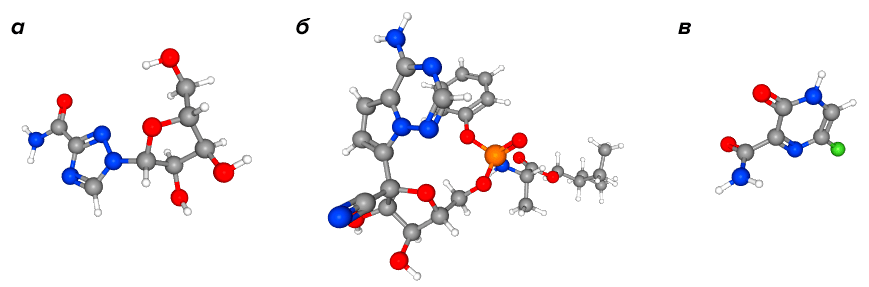
Рисунок 3. Препараты, применяемые для лечения коронавирусной инфекции. а — рибавирин, б — ремдесивир, в — фавипиравир.
Комбинация препаратов для лечения СПИДа (ингибиторы протеазы ВИЧ) — лопинавира с ритонавиром — оказалась эффективна в эксперименте на клеточной культуре и при лечении SARS [18], поэтому китайские ведомства, ответственные за борьбу с коронавирусом, некоторое время официально рекомендовали использовать ее для лечения COVID-19 [19]. Впрочем, уже в марте 2020 года показали, что для людей эта комбинация неэффективна.
Очевидно, что имеющегося арсенала недостаточно. Фармацевтическая отрасль оказалась застигнута врасплох, хотя ученые и прогнозировали появление нового коронавируса. Необходимо срочно разрабатывать новые лекарства для борьбы с инфекцией. На какие мишени они должны быть нацелены?
Интересующихся фармакологией приглашаем прочесть нашу статью про «Поиск лекарственных мишеней» [20].
Геномы коронавирусов — самые большие среди РНК-содержащих вирусов, но мало того, что размер генома SARS-CoV-2 — около 30 тыс. нуклеотидов [21], так у него еще и довольно сложный жизненный цикл (рис. 4). Однако сложное устройство легче «сломать», то есть потенциальных мишеней для лекарственного воздействия в SARS-CoV-2 достаточно (рис. 5). Но лекарство должно защищать не только от текущей вспышки, но и других инфекций, вызванных другими штаммами коронавируса, которые могут появиться в любой момент. Здесь приходится включать эволюционную логику и «прицеливаться» в белки, которые в ходе эволюции коронавирусов меняются мало (так называемые консервативные мишени). Одним из таких белков является главная протеаза вируса (Mpro) — ее и используют как главную мишень для разработки низкомолекулярных антикоронавирусных лекарств [22], [23].
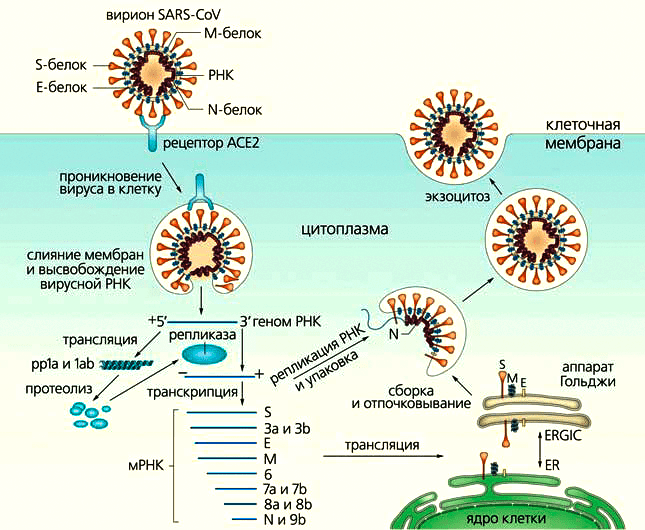
Рисунок 4. Жизненный цикл SARS-CoV. Его вирион проникает в клетку путем эндоцитоза: S-белок, прикидываясь «своим», связывается с ее рецептором (ACE2), затем расщепляется, вследствие чего мембраны вируса и эндосомы сливаются, и РНК выходит в цитоплазму. Геном транслируется в полипротеины (pp1a и 1ab), с помощью которых формируется копия РНК вируса, а также транскрибируется восемь мРНК-шаблонов для генерации его белков в просвете (ERGIC) между эндоплазматическим ретикулумом (ER) и аппаратом Гольджи [24]. Вирионы собираются в цитоплазме и выходят из клетки путем экзоцитоза [9], [11], [17].
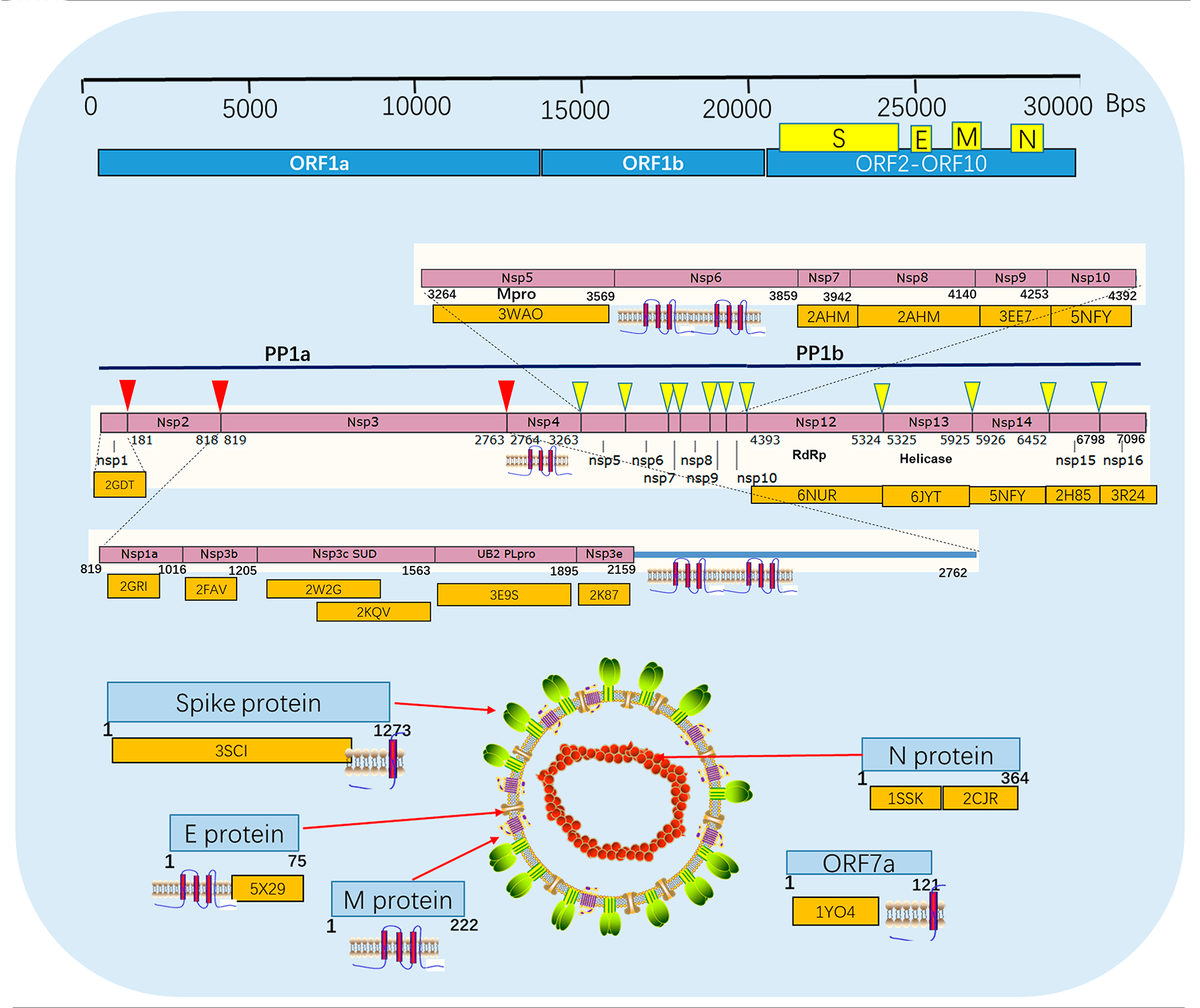
Рисунок 5. Наглядное изображение генома коронавируса и его структурных элементов — возможных терапевтических мишеней. Главная протеаза обозначена 3WAO.
Компьютерный дизайн лекарств
Главная проблема дизайна молекул против коронавируса заключается в том, что времени на создание принципиально нового лекарства попросту нет. Даже если молекулу-кандидата удастся найти очень быстро (что далеко не факт), доклинические и клинические испытания продлятся годы.
К счастью, смертность от COVID-19, по сравнению с SARS и MERS, весьма невелика, но это накладывает существенные требования к безопасности потенциального лекарства. Риск от его применения должен быть существенно ниже, чем от самой болезни. А значит, лекарство должно быть безопасным. Это сильно осложняет поиск: есть вероятность, что на этапе клинических испытаний будет выявлен слишком большой риск от приема препарата, несоизмеримый с опасностью болезни.
Отсюда возникла идея — не надо разрабатывать новые лекарства! Нужно использовать старые, то есть найти такие соединения, доклинические испытания которых проведены, безопасность доказана, и остается только проверить активность против SARS-CoV-2. Это называется перепрофилированием лекарств. В связи с этим можно выделить три стратегии разработки лекарства:
- Перепрофилирование других противовирусных препаратов. Как мы уже писали, можно попытаться использовать против SARS-CoV-2 препараты с широким противовирусным потенциалом, которые показали эффективность против РНК-вирусов (например, ВИЧ). Это самый быстрый и надежный способ с высоким шансом на успех, правда, эффективность может быть не очень велика, поскольку препарат не «настраивался» специально против коронавируса. Итак, можно попытаться использовать ингибиторы протеазы Mpro или РНК-полимеразы вируса, и именно по этому пути в настоящее время идут клинические испытания. Кроме того, этот подход не расширяет инструментария против вирусов. Он позволяет добиться тактической победы, но в долгосрочной перспективе — не самый лучший. А мы уже убедились, что новых коронавирусов нужно ждать постоянно. Да и не они одни образуют проблему «новых патогенов» — ранее незнакомых нам возбудителей с высоким эпидемическим потенциалом.
- Перепрофилирование других лекарств, не показавших активность против вирусов. Они хороши тем, что их профиль безопасности известен. Если имеются сведения, что какое-либо лекарство может иметь активность против коронавируса, можно пропустить стадию изучения его безопасности (пожалуй, самая долгая и дорогая часть доклинических испытаний и первая стадия клинических) — необходимо только изучить его эффективность (разумеется, с оговоркой, что профиль токсичности у больных коронавирусом может отличаться от такового у других пациентов: так, коронавирусу особенно подвержены люди с гипертензией и сахарным диабетом, и для них могут оказаться токсичными или плохо сочетаться с другими принимаемыми препаратами лекарства, которые подходят всем остальным; поэтому риск все равно есть). Шанс на успех в этом случае меньше. К примеру, не раз было показано, что противомалярийный препарат хлорохин эффективен против вирусов in vitro. Сейчас проводятся клинические испытания против COVID-19, но этот препарат уже не раз проваливался в испытаниях на людях [26].
- Поиск в существующих базах данных химических соединений (реальных или виртуальных). Можно пытаться придумать структуру, эффективную против коронавируса, на основе структуры биологической мишени (какой-нибудь макромолекулы коронавируса, которую надо заблокировать), или использовать методы искусственного интеллекта, способные генерировать структуры молекул, потенциально обладающих нужными свойствами. В любом случае эти молекулы будут новыми, с неизвестной фармакокинетикой и фармакодинамикой, которые нужно изучать и оптимизировать. С одной стороны, на исследования уйдут годы, но с другой — будет создан препарат с принципиально новым видом активности, «настроенной» против коронавируса. В будущем от него может быть и другая польза. Так, препарат N3, который разрабатывался против коронавируса SARS-CoV 2003 г., пригодился при расшифровке структуры протеазы коронавируса 2019 г. Это лучший путь в долгосрочной перспективе, хотя он самый дорогой и долгий.
Таким образом, единственно правильного пути нет. Каждый имеет достоинства и недостатки, но в целом эти три стратегии дополняют друг друга и способны помочь человечеству побороть COVID-19. По этой причине работы ведутся во всех направлениях.
Чаще всего в поиске новых лекарств используется методика докинга (или молекулярной стыковки) . В докинге оценивается, возможно ли проникновение молекулы в «связывающий карман» биологической мишени и эффективное взаимодействие с ней. Для этого используется специальная оценочная функция, обычно основанная на методике эмпирических силовых полей. Это один из самых распространенных и эффективных методов компьютерного моделирования связывания лекарства с рецептором. Чаще всего при поиске новых лекарств используется библиотека соединений-кандидатов и трехмерная структура молекулярной мишени. В дальнейшую разработку поступают лидерные молекулы, показавшие по результатам моделирования максимальную энергию связывания. Такой подход называется виртуальным скринингом.
Докинг и другие компьютерные методики рассмотрены в публикациях «Виртуальные тропы реальных лекарств» [27] и «12 методов в картинках: “сухая” биология» [28], а сама область рационального конструирования лекарств — в статье «Драг-дизайн: как в современном мире создаются новые лекарства» [29] и книге «От оргазма до бессмертия. Записки драг-дизайнера» [30].
Беда в том, что SARS-CoV-2 появился внезапно, а для расшифровки трехмерной структуры его белков требовалось время. Но геном уже расшифрован, а следовательно, известна и последовательность аминокислот в его белках! В таком случае можно получить приблизительную 3D-структуру путем моделирования по гомологии . С помощью специальных программ по базам данных находят белки, сходные по последовательности с исследуемым, имеющие общее с ним эволюционное происхождение (это и называется гомологией), и, что важно, для которых уже расшифрована трехмерная структура. Ее используют как шаблон, на который с помощью других программ буквально «натягивают» исследуемый белок. Затем может следовать процедура небольшой «подгонки», чтобы смоделированный белок «улегся поудобнее» — скорее всего, так он и будет сворачиваться в естественных условиях.
Как это делается, читайте в статье «Торжество компьютерных методов: предсказание строения белков» [31].
Именно этот подход применили китайские ученые, которые использовали в качестве шаблона для моделирования протеазу Mpro SARS-CoV — ту самую «первую ласточку» из 2002 г. [23]. Эта структура доступна всем в банке данных PDB под кодом 2GTB [22]. Библиотека для докинга включала 1900 лекарств. Но более трети из них имели высокие значения расчетной энергии связывания. Сократить круг поиска помог отбор молекул, связывающихся с протеазой аналогично известным веществам. Этот поиск также проводили с помощью вычислительной техники. Так список сократился до 159.
Далее исследователи визуализировали модели полученных комплексов и «на глаз» выбрали 15 структур. Их использовали для более точного метода — моделирования движений молекул во времени (молекулярной динамики ). В результате «победителем» оказался нелфинавир — противовирусное средство, которое уже используется против ВИЧ. Кроме того, исследователи посчитали потенциально интересными препарат для снижения холестерина питавастатин, антипсихотический препарат перампанел и празиквантел, применяющийся против гельминтов. Однако последние три интересны скорее как стартовые соединения для дальнейшей оптимизации.
И об этом методе есть статья на «Биомолекуле»: «Молекулярная динамика биомолекул. Часть I. История полувековой давности» [32].
26 января 2020 г. «на подмогу» исследователям подоспела экспериментально определенная структура Mpro самого SARS-CoV-2, да еще и в комплексе с известным блокатором N3! Она также доступна в банке данных PDB для «всеобщего обозрения» под кодом 6LU7 (рис. 6). Виртуальный скрининг стал проще. И снова в исследовании появляются препараты против ВИЧ: ученые использовали их как инструмент для сравнения связывающих участков Mpro «старого» (SARS-CoV) и «нового» (SARS-CoV-2) коронавирусов [33]. Моделирование показало, что исследуемые «карманы» в белке связывают лекарство сходным образом. Далее ученые предприняли виртуальный скрининг, используя в качестве библиотеки базу данных DrugBank. Они выявили 10 препаратов-«лидеров», но результаты несколько разочаровали. Бóльшая часть этих соединений не имело никакого отношения к вирусам, а сам спектр соединений довольно пестрый: от антипсихотиков до противогрибковых агентов. Ни проверки идеи, ни даже оптимизации «находок» не последовало. Впрочем, данная работа — препринт, как и предыдущая. А значит, пока это лишь предварительные данные.
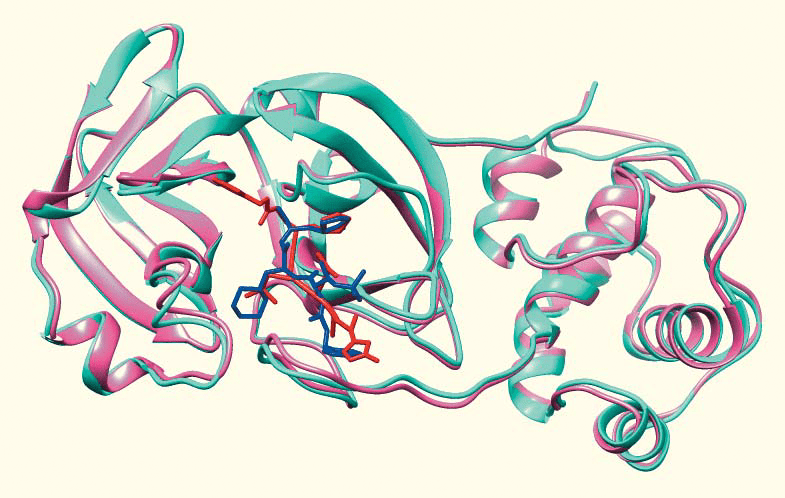
Рисунок 6. Наложение недавно расшифрованной протеазы SARS-CoV-2 (розовая, связана с красным блокатором) на гомологичную протеазу SARS-CoV (голубая, связана с синим ингибитором). Обратите внимание на сходство структуры протеаз и положения блокаторов в активном центре (хотя это разные блокаторы). Изображение сделано авторами статьи [35] на основе структур PDB 6lu7 (DOI: 10.2210/pdb6LU7/pdb) и 2gx4 (DOI: 10.2210/pdb2GX4/pdb).
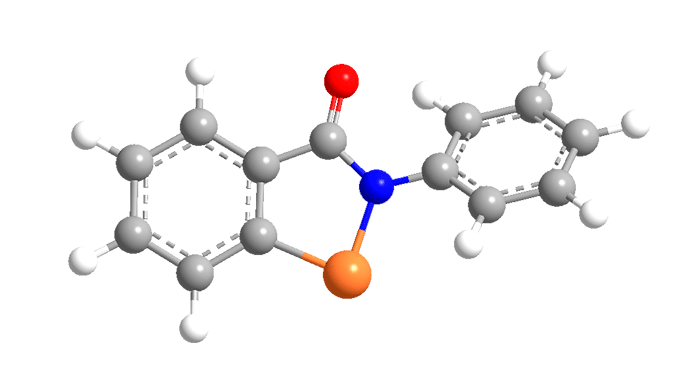
Другая группа китайских ученых получила более надежные результаты [34]. Их библиотека включала 8 тыс. лекарств. В результате скрининга обнаружилось 690 «лидеров». Однако эта группа исследователей, в отличие от предыдущей, исключила «неинтересные» молекулы: красители, токсичные вещества, препараты, применяемые в неврологии (в вирусологии им делать нечего) и противоопухолевые лекарства с высокой токсичностью. Это помогло не только снять один лишний вопрос к своей работе, но и сократить «шорт-лист» до 50 кандидатов. Из этого вполне разумного количества авторы выделили четыре соединения: прулифлоксацин (фторхинолоновый антибиотик), биктегравир (ингибитор интегразы ВИЧ), нелфинавир (уже упоминавшийся ингибитор протеазы ВИЧ) и тегобувир (препарат от гепатита С) (рис. 7).
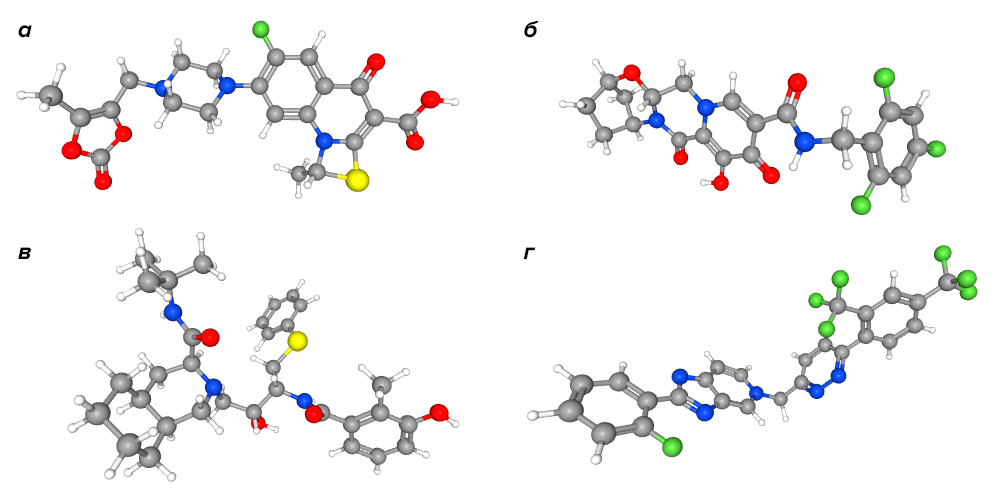
Рисунок 7. Вещества-«победители» в докинге. а — прулифлоксацин, б — биктегравир, в — нелфинавир, г — тегобувир.
В общем, виртуальный скрининг и докинг показали свою привлекательность в деле борьбы с коронавирусом. Работы с использованием этого инструмента продолжаются очень активно, правда есть и проблема: пока у этих работ нет экспериментального подтверждения, но это дело времени. Нужно помнить, что в большинстве лабораторий этого штамма еще просто нет, и обучение работе с ним займет время (а также, в связи с опасностью вируса, такие лаборатории должны быть особо сертифицированы). В этом смысле создание международных консорциумов представляется особенно перспективным, и они в настоящее время уже формируются.
Перепрофилирование в пробирке
Насколько бы докинг ни был мощным, только эксперимент есть критерий истины. Молекулярная динамика более точна, но это крайне ресурсоемкий метод, и он не годится для скрининга больших библиотек. А что, если провести аналогичный скрининг не в компьютерной модели, а in vitro? На настоящей протеазе вируса? Одна большая группа специалистов провела такой масштабный эксперимент [21]. Мишенью вновь была та же самая Mpro. Скрининг проводили измерением связывания соединения с Mpro методом на основе ферстеровского резонансного переноса энергии (FRET) [36], часто применяющимся в молекулярной биологии. Причем размеру библиотеки могли бы позавидовать даже исследователи, проводившие куда менее затратные компьютерные исследования: более 10 тыс. соединений, среди которых одобренные лекарства, препараты, проходящие клинические испытания, и природные соединения. Так что по производительности этот биологический скрининг не уступал некоторым работам в области виртуального скрининга.
В этом испытании было выявлено 7 молекул-«победителей». Вторым этапом эти соединения протестировали на способность предотвращать заражение клеточной культуры вирусом SARS-CoV-2. «Победил» антиоксидант эбселен — селенорганическое вещество. Результаты похуже показал TDZD-8, который тестируют в настоящее время против болезни Альцгеймера, а также разработанный против SARS-CoV-2 ковалентный ингибитор N3 (рис. 8).
Любопытно, что компьютерные методы на основе докинга не выделили ни одну из этих молекул! Но их вполне могло не быть в базах данных, использованных исследователями. А эбселен и N3 вдобавок могут быть докингу просто не по зубам. В состав первого входит селен, параметры которого в силовых полях для расчета энергии докинга недостаточно надежны. А молекула N3 представляет собой ковалентный ингибитор, связывающийся с белком по реакции Михаэля. Использованные исследователями методы докинга этого учесть не могут, хоть и существуют специальные алгоритмы для ковалентного докинга.
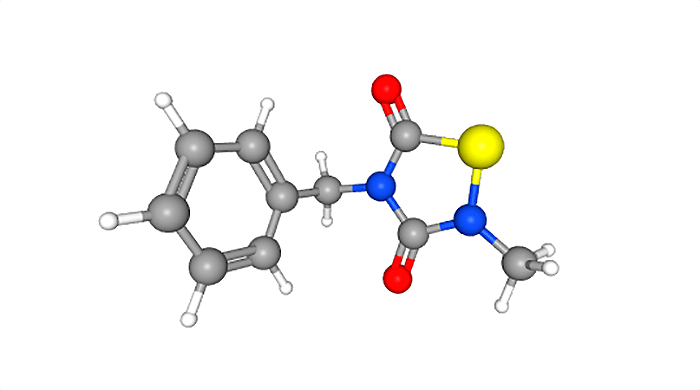
Однако та же группа исследователей провела и компьютерные «эксперименты», несколько «реабилитировав» их. Ученые сделали компьютерный скрининг, в ходе которого победил цинансерин — антагонист серотонина, изначально разрабатывавшийся для фармакологического воздействия на нервную систему (рис. 9). Любопытно, что он уже зарекомендовал себя как ингибитор протеазы опасного коронавируса из 2002 г. — SARS-CoV [37]. В уже упоминавшемся тесте на клеточной культуре цинансерин также доказал свою эффективность [21].
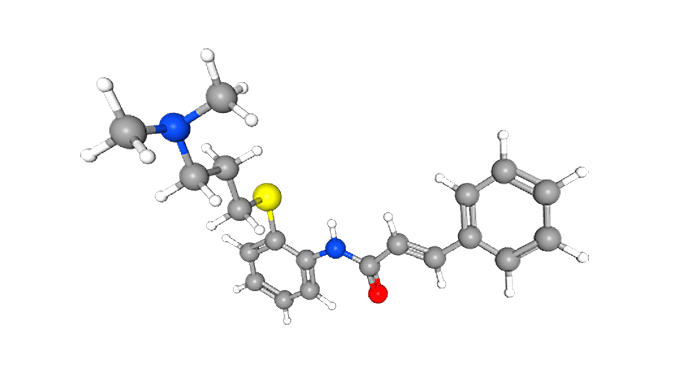
Рисунок 9. Цинансерин
Так что работа этой группы скорее убеждает в необходимости должного подхода к компьютерному скринингу и его комбинации с экспериментальными исследованиями.
Искусственный интеллект на тропе войны
Докинг — это все же просто вычислительная методика, работающая по явно заданному компьютерному алгоритму. А как насчет более передовых технологий?
Горячей темой в разработке лекарств являются машинное обучение и искусственный интеллект. Несмотря на свою молодость, такие разработки уже достигли неплохих успехов: искусственный интеллект предсказывает пути синтеза неорганических материалов [38], определяет, как «свернется» белок [39], разрабатывает новые ингибиторы DDR-киназ [40]. Это будущее, стремительно входящее в практику фармацевтических компаний. Однако коронавирус ждать не будет — нужно, чтобы будущее наступило экстренно.
Компания InSilico Medicine, которая специализируется на применении искусственного интеллекта в медицине, предложила использовать свои протоколы для разработки новых антикоронавирусных лекарств. В отличие от предыдущих работ, это не перепрофилирование, а «придумывание» новой молекулы. Изюминка в том, что «придумать» (сгенерировать) структуру должен был компьютер [41]. А генерировать предполагалось на основе уже знакомой нам протеазы Mpro.
Так как эта компания начала работу в числе первых, они «не успели» дождаться кристаллографической структуры и использовали модель по гомологии, «вставив» в нее лиганд, содержащийся в шаблоне, и оптимизировав его положение молекулярным моделированием. К этой информации было добавлено почти 6 тыс. известных антипротеазных соединений. Компьютер должен был «придумать» соединения как на основе структуры связывающего кармана, так и на основе известных блокаторов.
В программе для «придумывания» была реализована особая архитектура нейронной сети (имитирующая нейронные сети мозга). Здесь уже нет явного алгоритма — как и живой мозг, программа учится. Компания использовала весь имеющийся у них арсенал методик: сети-автокодировщики, состязательные сети (в таком методе две нейронные сети соревнуются между собой), языковые модели (работающие по принципу предсказания следующего слова в предложении).
«Придуманные» компьютером молекулы далее подвергались фильтрации и отсеву по нескольким критериям: подходящие физико-химические параметры, отсутствие «нехороших» групп, предсказанная активность, качество докинга в модели протеазы, показатели новизны и разнообразия.
Методология довольно впечатляющая и очень напоминает таковую в предыдущем исследовании этой группы по созданию ингибиторов DDR-киназ [40], опубликованном в журнале Nature Biotechnology (что говорит само за себя). Сейчас группа занимается синтезом соединений для дальнейшей экспериментальной проверки, так что — ждем окончательных результатов и надеемся, что они будут хорошими!
Совсем другим путем пошли их конкуренты из компании BenevolentAI [42]. Они сконцентрировали внимание на перепрофилировании. Но на этот раз не использовали скрининг и докинг, а предложили искусственному интеллекту заняться процессом познания. Из научной литературы был извлечен граф знаний. На нем видно, что белок ACE2, с которым связывается коронавирус, участвует в эндоцитозе. Важным регулятором эндоцитоза является AP2-ассоциированная протеинкиназа-1 (AAK1). Соответственно, ее подавление может предотвратить эндоцитоз вируса.
Далее из 300 существующих ингибиторов ААК1 отобрали 40 одобренных для применения в клинической практике. Исключили препараты, обладающие выраженными нежелательными реакциями или требующие высоких дозировок лекарств. Среди шести высокоактивных ингибиторов особенно интересен ингибитор янус-киназ барицитиниб (изначально разработанный для лечения ревматоидного артрита). Он ингибирует не только ААК1, но и циклин-G-ассоциированную киназу (GAK), которая также участвует в эндоцитозе. Авторы полагают, что эта молекула может использоваться для профилактики и лечения коронавирусной инфекции. Идея хорошая, и — в кои-то веки — связана не с протеазой вируса, а с другой мишенью. Но это лишь модельные данные — их еще надо проверить.
По этим работам был написан весьма интересный критический разбор [43]. Обе работы критиковались в основном за очевидность. Так, критики считали, что работа сотрудников BenevolentAI — это скорее продвинутый научный поиск, доступный и человеку. Зачем здесь искусственный интеллект? Его триумфа здесь особо не просматривается. Но, на наш взгляд, сама по себе разработка, где искусственный интеллект собирает граф научных знаний с потенциальным медицинским применением, заслуживает внимания. Хотя бы с технической точки зрения. Такой подход может оказаться полезным в будущем — время покажет. Повторимся, что все данные пока предварительные.
InSilico критикуют за то, что их генератор создает молекулы, собирая «кусочки» известных лекарств. Например, одна из них содержит кусок противогельминтного препарата празиквантела (который в одной из упомянутых выше работ «выиграл» в докинге). Но основная претензия критиков заключается в том, что сотрудники InSilico не использовали перепрофилирование. Критики посчитали, что вряд ли кто-нибудь захочет вложиться в реальную разработку абсолютно новых лекарств. Но, в нашем понимании, это не проблема искусственного интеллекта и научного подхода коллектива InSilico.
Но не бизнесом единым движется область искусственного интеллекта в дизайне лекарств. Исследователи из Университета Мичигана использовали нейронную сеть-автокодировщик для генерации новых молекул, активных против Mpro [44]. Было сгенерировано несколько молекул, которые имеют неплохие физико-химические характеристики, а также доступны для синтеза (синтетическая доступность оценивалась с помощью SAScore [45]). Они сравнили созданные молекулы с рекомендованным на момент исследования сочетанием лопинавира с ритонавиром и нашли, что в целом характеристики биодоступности и синтетической доступности сгенерированных молекул были лучше. Но даже беглый взгляд на эти молекулы позволяет заметить, что тут еще нужно поработать над медико-химической составляющей. Многие структуры содержат очень реакционноспособные фрагменты, и до вируса в таком виде молекулы точно не дойдут. Это исследование еще очень «сырое».
В другой работе [46] той же команды использовались свёрточные нейронные сети. Они работают по принципу нейронных сетей в зрительных центрах коры головного мозга. Сети предсказывали энергии связывания молекул с Мpro-белком коронавируса. Сначала авторы собрали данные по молекулам, активным против SARS, потом «сдокировали» их в расшифрованную протеазу SARS-CoV-2 и на основе этого создали обучающую выборку. Отдельно собрали данные из базы BindingDB уже по отношению к разным белкам. С помощью полученной модели протестировали около 1400 известных лекарств для поиска возможности их перепрофилирования под лечение коронавируса. На их основе нашли группу молекул, которые можно перепрофилировать. По активности в этой группе лидировали: противоопухолевое таргетное средство, ингибитор протеасом бортезомиб, противотревожный и седативный препарат флуразепам и таргетный препарат для лечения лейкемии понатиниб.
Интересны данные исследователей по ингибиторам других протеаз как кандидатам в блокаторы протеазы коронавируса (табл. 1). «Лидером» оказался ингибитор протеазы вируса гепатита С боцепревир — только его авторы отмечали как имеющего высокую предсказанную аффинность к протеазе коронавируса. Его предсказанная аффинность высока: –9,36 ккал/моль. «Аутсайдером» оказался фосфорорганический яд изофлурофат — всего –4,94 ккал/моль, что почти ничто по меркам молекулярного моделирования.
| Класс веществ | Препараты |
|---|---|
| Ингибиторы вирусных протеаз | Ампренавир, атазанавир, боцепревир, дарунавир, индинавир, лопинавир, нелфинавир, ритонавир, саквинавир, симепревир, телапревир, типранавир, фосампренавир |
| Ингибиторы факторов свертывания крови (антикоагулянты) | Апиксабан, аргатробан, дабигатрана этексилат, ксимелагатран, ривароксабан |
| Ингибиторы АПФ | Беназеприл, каптоприл, квинаприл, лизиноприл, моэксиприл, периндоприл, рамиприл, спираприл, трандолаприл, фозиноприл, цилазаприл, эналаприл |
| Ингибиторы DPP4 | Алоглиптин, видаглиптин, линаглиптин, саксаглиптин, ситаглиптин |
| Ингибиторы других протеаз человеческого организма | Кандоксатрил, ремикирен, циластатин |
| Другие препараты/вещества | Изофлурофат, экабет |
Несмотря на то, что авторы выделили только боцепревир, ему «наступают на пятки» несколько ингибиторов вирусных протеаз, используемых для лечения вирусного гепатита и ВИЧ-инфекции, а также — немного неожиданно — ингибиторы факторов свертывания крови, которые также являются протеазами. В числе победителей также ингибиторы ангиотензинпревращающего фермента — того самого, к которому прикрепляется коронавирус. Это ведь тоже протеаза. Так что эти результаты также требуют проверки. Возможно, удастся создать препарат с двойным действием.
Исследователи группы Артема Черкасова, работающие в Университете Британской Колумбии (Ванкувер) [47], применили разработанный ими подход «глубокого докинга» [48] для поиска активных молекул с использованием кристаллической структуры Мpro уханьского коронавируса. «Глубокий докинг» представляет собой количественную модель «структура—свойство» на основе глубокого обучения, которая позволяет предсказывать результаты докинга. Методологически это исследование похоже на работу группы из Мичиганского университета.
Такой подход позволяет «просеять» гораздо больше соединений, чем при обычном докинге: ведь машина умеет предугадывать, какая энергия получится при докинге, и отбрасывать неподходящие молекулы. С использованием этого подхода авторы смогли провести виртуальный скрининг более 1,3 млрд (!) молекул из базы данных ZINC. В числе выявленных ими «лидеров» снова (уже в который раз!) оказался препарат для лечения ВИЧ — лопинавир. Также было отмечено «соединение 80» (имени у «новорожденного» вещества пока нет, только номер) — ингибитор протеазы SARS-CoV (из 2002 г.) (рис. 10). Однако 99% отмеченных соединений пока не синтезировано — это еще предстоит сделать.
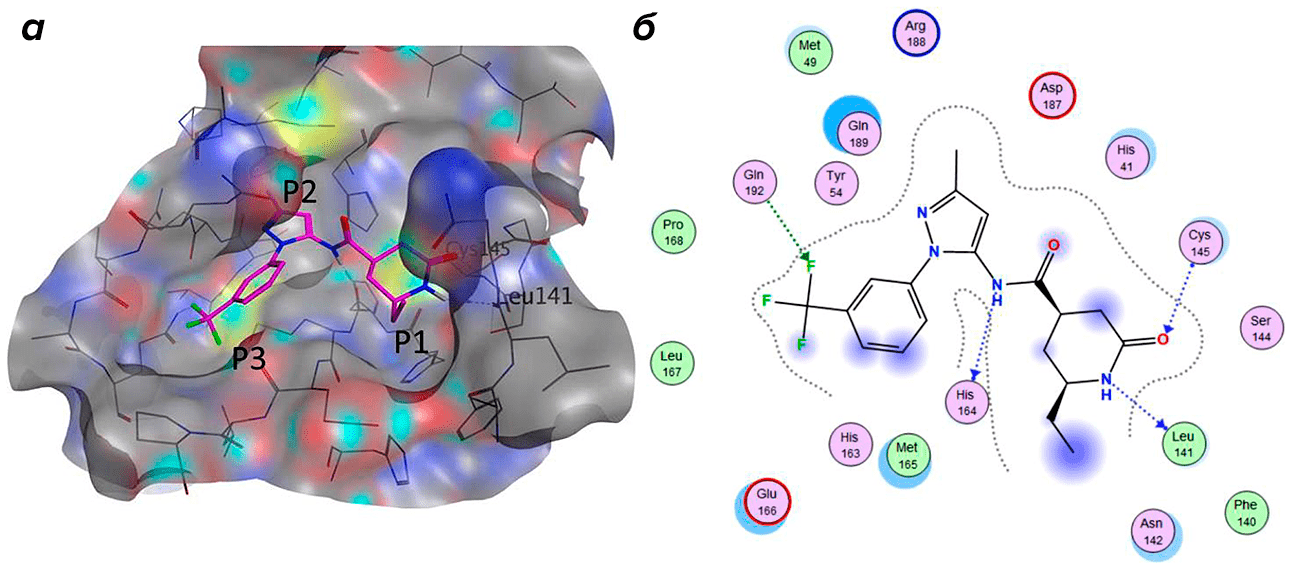
Рисунок 10. Предсказанное положение одного из веществ (а; пурпурная стержневая модель), найденных группой А. Черкасова, и взаимодействия (б) этого соединения в связывающем участке протеазы SARS-CoV-2 по итогам докинга. Синими пунктирными линиями показаны водородные связи с пептидным остовом, зелеными — с атомами боковой цепи.
Группа корейских ученых [49] обучила нейронную сеть предсказывать константу связывания молекулы с белком и провела поиск активных молекул среди коммерчески доступных. В результатах этого исследования оказались сразу три препарата против ВИЧ — ритонавир, который ранее был рекомендован для терапии коронавирусной инфекции (в сочетании с лопинавиром), а также атазанавир и эфавиренц. Немного разнообразия в эту подборку внесло противовирусное средство ганцикловир (но — от цитомегаловирусной инфекции).
Не протеазой единой...
Читатели могли обратить внимание (и мы сами неоднократно подчеркивали), что в компьютерных скринингах на блокирование Mpro часто «выигрывают» препараты для лечения ВИЧ-инфекции и гепатита C. Причем именно те, которые блокируют протеазы этих вирусов. Такое решение кажется очевидным — ведь протеазы могут быть похожи у разных вирусов. Кажется, что до этого можно догадаться и безо всякого моделирования! И исследователи уже догадались — об этом говорит применение комбинации лопинавира с ритонавиром против коронавирусных инфекций. А если так, то зачем вообще все эти высокопроизводительные вычисления и искусственный интеллект?
Не все так просто. Протеазы могут различаться по структуре и механизму катализа. Например, протеаза ВИЧ — аспартатная, протеаза вируса гепатита C — сериновая [50], а главная протеаза SARS-CoV-2 — цистеиновая [51], то есть они относятся к разным классам и имеют разный механизм действия. При таком разнообразии будет удачей, если ингибитор от одной протеазы подойдет к другой и будет эффективно ее блокировать. И искусственный интеллект здесь может послужить неплохим подспорьем, позволяя прикинуть шансы.
Кроме того, при недостатке экспериментальной информации самый очевидный путь может оказаться неверным. Например, если анти-ВИЧ-препараты эффективны против коронавирусов, связано ли это именно с протеазой?
Рассмотренные исследования давали положительный эффект, потому что в них протеаза была единственной мишенью. Этот момент может быть источником ошибки. А что, если сделать скрининг по всем возможным мишеням в составе коронавируса, которые изображены на рисунке 5? Этот рисунок взят из статьи [17], авторы которой уже провели такую масштабную работу. Построив по гомологии модели всех белков коронавируса, они провели скрининг существующих лекарств и натуральных соединений методом докинга.
По каждой мишени был получен список из 40 «лидеров», что в сумме, пожалуй, ставит рекорд по производительности среди статей, затронутых в обзоре. Весь список приведен в оригинальной статье — здесь же мы коснемся самых интересных находок.
Для ритонавира было найдено две возможные мишени, для лопинавира — даже четыре, но главной протеазы среди них нет! Зато рибавирин — аналог нуклеозида, для которого ожидаемо действие на репликацию и синтез белка, — оказался лидером по связыванию с папаин-подобной протеазой PLpro. Ему на пятки наступает валганцикловир — противогерпетическое средство, и тоже нуклеозидной природы! Правда, результаты скрининга против Mpro немного перекликаются с результатами команды из Университета Мичигана: там все же представлены препараты против ВИЧ — ампренавир и блокатор нейтральной эндопептидазы кандоксатрил. Но уверенность в протеазе как мишени перепрофилирования это исследование все же способно пошатнуть.
А коллектив исследователей из Польши исследовал главные протеазы SARS-CoV и SARS-CoV-2 методом молекулярной динамики и сообщил неприятное известие: оказывается, у этих белков разная форма и объем связывающего «кармашка» (рис. 11). «Карман» очень гибкий и, что самое плохое, подвержен мутациям [51]. Эта работа практически камня на камне не оставляет от представления о консервативности главной протеазы. Оказывается, это не лучшая мишень для драг-дизайна — по крайней мере, компьютерного.
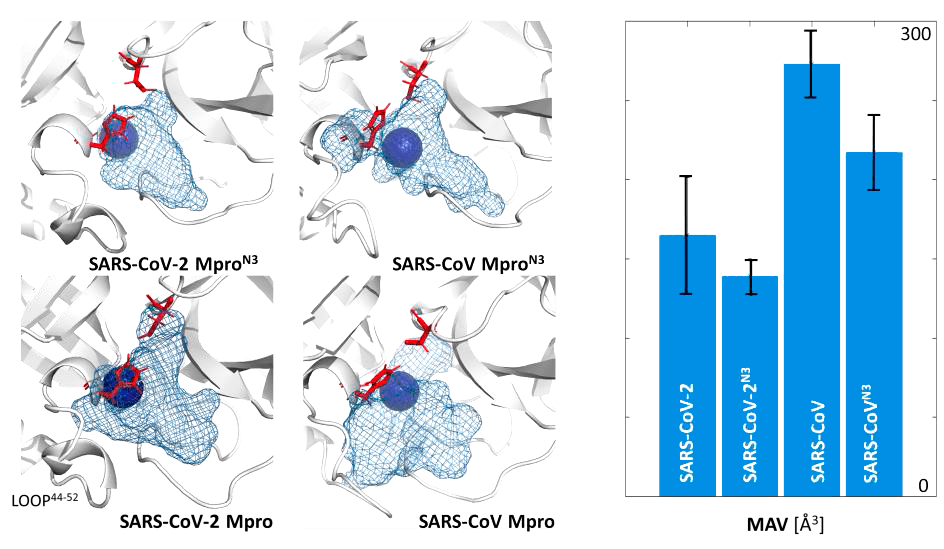
Рисунок 11. Форма сайта связывания главных протеаз вирусов 2002 (SASR-CoV) и 2019 годов (SARS-CoV-2) с ингибитором N3 и без него. На диаграмме справа — доступный объем сайта связывания. Видно, что «карман» протеазы SARS-CoV-2 меньше и имеет другую форму, нежели SARS-CoV.
Кроме этого, появились первые данные по клиническим испытаниям комбинации лопинавира с ритонавирсом в сравнении со стандартной терапией (кислород, ИВЛ и пр.) без приема этих лекарств. Результаты разочаровали: снижение смертности (19,2% vs 25,0%) и уменьшение продолжительности болезни (на 1,4 дня) оказались статистически незначимыми. При этом у 19% пациентов возникли побочные эффекты [52]. А ведь именно на эти препараты была большая надежда в плане блокирования Mpro.
Такое обилие плохих новостей несколько охладило интерес к главной протеазе. Скрининги по ней по-прежнему проводят многие исследовательские группы, но это направление стало своеобразным мейнстримом. И исследователи стали искать другие идеи.
Недавно появилось еще два масштабных скрининга против всех возможных мишеней [53], [54], причем один из них — даже с молекулярной динамикой [53]! Кроме этого, уже расшифрована структура белка «шипов» коронавируса и механизм его взаимодействия с АПФ на мембране клетки [55–58]. Этой перспективной мишенью уже воспользовались исследователи из Университета Теннесси — с помощью молекулярной динамики они смоделировали контакт между «шипом» вируса и АПФ и попробовали сделать масштабный докинг в него [59]. Получился довольно большой список «лидеров».
РНК-зависимая РНК-полимераза (RdRp) вируса стала объектом исследования ученых из Исследовательского центра IBM имени Томаса Уотсона и Университета Колумбии [60]. Они исследовали связывание уже упоминавшегося ремдесивира с этой полимеразой методом молекулярной динамики.
Ремдесивир исходно был разработан для лечения лихорадки Эбола, вирус которой также имеет РНК-зависимую РНК-полимеразу. В организме ремдесивир превращается в структурный аналог нуклеотидов, которыми полимераза достраивает цепь РНК (рис. 12). Схватив такую «молекулярную обманку», полимераза блокируется, а цепь РНК обрывается [61].
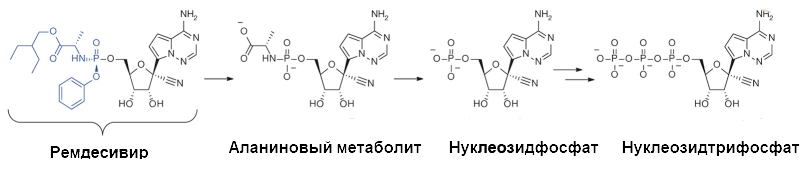
Рисунок 12. Ремдесивир превращается в нуклеозидтрифосфат, имитирующий «строительные блоки» РНК. Стоит РНК-полимеразе связать такой ложный «кирпичик», как цепь РНК оборвется.
С помощью молекулярной динамики исследователи хотели предсказать, как ремдесивир может связаться с полимеразой нового коронавируса. По их расчетам, его связывание должно быть примерно в 800 раз сильнее, чем связывание АТФ! Впрочем, пока это только модельные данные. Но биологически правдоподобные: RdRp есть у всех РНК-содержащих вирусов, и их структура наиболее консервативна (рис. 13). Их даже используют для установления филогении РНК-содержащих вирусов, подобно тому, как для клеточных организмов используют 16S рибосомную РНК [61]. Так что в этом случае ингибитор от одной полимеразы вполне может подойти к другой, а это внушает надежду.
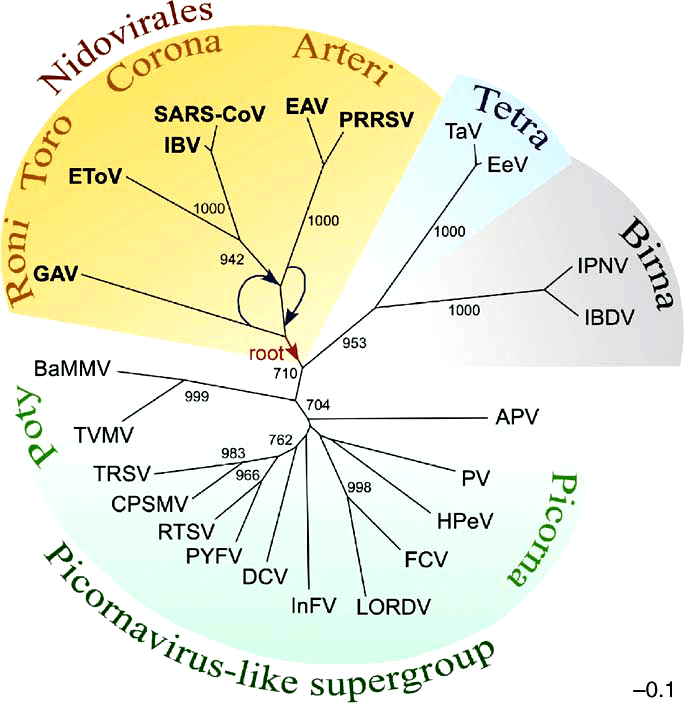
Рисунок 13. Филогенетическое дерево RdRp РНК-содержащих вирусов (коронавирусы вверху). Возможно, все RdRp — очень дальние родственники друг другу.
А пока все искали оружие против вируса, исследователи из компании GERO решили поискать препарат, который сделает хозяина невосприимчивым к вирусу! С помощью собственной нейронной сети они нашли молекулы, вызывающие изменения экспрессии генов, похожие на эффект нокаута гена COBP2. Последний, в свою очередь, отвечает за репликацию вируса SARS. Так они нашли некоторое количество противовирусных препаратов с активностью против SARS и MERS (с эффективной концентрацией на уровне микромоль).
Относительная доступность средств био- и хемоинформатики делает возможным поиск сразу во всех направлениях. Но экспериментов по найденным новым веществам пока очень мало, и поэтому неясно: помогут ли в итоге вычислительные методы и искусственный интеллект в борьбе с вирусом? Это покажет время, однако нынешняя пандемия является в некотором смысле «тренировочной» для био- и хемоинформатиков. Даже если в итоге «компьютерные» находки не потребуются — мы будем иметь бесценный опыт компьютерной «гонки вооружений» с новыми патогенами, который может пригодиться в будущем.
Тем временем... (вместо послесловия)
Согласно последней статье в Nature Biotechnology [62], началось несколько клинических испытаний по перепрофилированию лекарств в целях лечения COVID-19. Один из подходов заключается в перепрофилировании известных противовирусных препаратов, в основном из арсенала анти-ВИЧ-терапии. Имеются наблюдения, которые указывают на возможность перекрестного эффекта против коронавируса. Второй перспективный подход касается воздействия на организм человека, чтобы уменьшить «цитокиновый шторм», вызываемый иммунным ответом на вирус [63].
Разработка вакцин
В настоящее время около 70 компаний и научных коллективов по всему миру ведут разработку вакцин, список исследований опубликован ВОЗ. Три вакцины находятся на стадии клинических испытаний. А среди вакцин на стадии доклинической разработки — «предложения» на любой вкус. Много ДНК-вакцин и вакцин на основе рекомбинатных белков; при создании одной из них вирусные белки даже будут экспрессироваться в растениях. Есть несколько разработок с безыгольным впрыском в кожу и даже в виде капель или спрея в нос. Так что есть надежда, что у нас не только скоро будет вакцина, но прививка будет еще и безболезненной! Здорово, правда?
Исходный вариант этой статьи опубликован в журнале «Природа» №3 за 2020 г. [64].
Литература
- Хроника распространения SARS-CoV-2;
- 2019-nCoV: очередной коронованный убийца?;
- COVID-19: отвечаем на вопросы;
- COVID-19: что мы знаем и чего не знаем;
- Roujian Lu, Xiang Zhao, Juan Li, Peihua Niu, Bo Yang, et. al.. (2020). Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. The Lancet. 395, 565-574;
- Wu F., Zhao S., Yu B., Chen Y.-M., Wang W., Hu Y. et al. (2020). Complete genome characterisation of a novel coronavirus associated with severe human respiratory disease in Wuhan, China. bioRxiv;
- Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. (2020). The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol. 5, 536-544;
- McIntosh K. and Perlmanin S. Coronaviruses, including severe acute respiratory syndrome (SARS) and middle east respiratory syndrome (MERS). In: Mandell, Douglas, and Bennett's principles and practice of infectious diseases (eighth edition) / ed. by Bennett J.E., Dolin R., Blaser M.J. Elsevier Inc., 2014. P. 1928–1936;
- Carter J.B. and Saunders V.A. Virology: principles and applications. Chichester, 2007;
- Кулагина М.Г. Коронавирусная инфекция // Инфекционные болезни: национальное руководство / Н.Д. Ющук, Ю.Я. Венгеров. М., 2018. С. 759–768;
- . (2012). Nidovirales. Virus Taxonomy. 784-794;
- Vineet D Menachery, Boyd L Yount, Kari Debbink, Sudhakar Agnihothram, Lisa E Gralinski, et. al.. (2015). A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence. Nat Med. 21, 1508-1513;
- Zhengli Shi, Zhihong Hu. (2008). A review of studies on animal reservoirs of the SARS coronavirus. Virus Research. 133, 74-87;
- David Cyranoski. (2020). Mystery deepens over animal source of coronavirus. Nature. 579, 18-19;
- Anthony R. Fehr, Stanley Perlman. (2015). Coronaviruses: An Overview of Their Replication and Pathogenesis. Coronaviruses. 1-23;
- Markus Hoffmann, Hannah Kleine-Weber, Simon Schroeder, Nadine Krüger, Tanja Herrler, et. al.. (2020). SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor. Cell;
- Canrong Wu, Yang Liu, Yueying Yang, Peng Zhang, Wu Zhong, et. al.. (2020). Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods. Acta Pharmaceutica Sinica B;
- C M Chu. (2004). Role of lopinavir/ritonavir in the treatment of SARS: initial virological and clinical findings. Thorax. 59, 252-256;
- Ying-Hui Jin, , for the Zhongnan Hospital of Wuhan University Novel Coronavirus Management and Research Team, Evidence-Based Medicine Chapter of China International Exchange and Promotive Association for Medical and Health Care (CPAM), Lin Cai, Zhen-Shun Cheng, Hong Cheng, et. al.. (2020). A rapid advice guideline for the diagnosis and treatment of 2019 novel coronavirus (2019-nCoV) infected pneumonia (standard version). Military Med Res. 7;
- Поиск лекарственных мишеней;
- Zhenming Jin, Xiaoyu Du, Yechun Xu, Yongqiang Deng, Meiqin Liu, et. al.. (2020). Structure of Mpro from COVID-19 virus and discovery of its inhibitors. Nature;
- Ting-Wai Lee, Maia M. Cherney, Jie Liu, Karen Ellis James, James C. Powers, et. al.. (2007). Crystal Structures Reveal an Induced-fit Binding of a Substrate-like Aza-peptide Epoxide to SARS Coronavirus Main Peptidase. Journal of Molecular Biology. 366, 916-932;
- Xu Z., Peng C., Shi Y., Zhu Zh., Mu K., Wang X., Zhu W. (2020). Nelfinavir was predicted to be a potential inhibitor of 2019-nCov main protease by an integrative approach combining homology modelling, molecular docking and binding free energy calculation. bioRxiv;
- Silke Stertz, Mike Reichelt, Martin Spiegel, Thomas Kuri, Luis Martínez-Sobrido, et. al.. (2007). The intracellular sites of early replication and budding of SARS-coronavirus. Virology. 361, 304-315;
- Eric F. Pettersen, Thomas D. Goddard, Conrad C. Huang, Gregory S. Couch, Daniel M. Greenblatt, et. al.. (2004). UCSF Chimera?A visualization system for exploratory research and analysis. J. Comput. Chem.. 25, 1605-1612;
- Franck Touret, Xavier de Lamballerie. (2020). Of chloroquine and COVID-19. Antiviral Research. 177, 104762;
- Виртуальные тропы реальных лекарств;
- 12 методов в картинках: «сухая» биология;
- Драг-дизайн: как в современном мире создаются новые лекарства;
- Григорий Никифорович: «От оргазма до бессмертия. Записки драг-дизайнера». Рецензия;
- Торжество компьютерных методов: предсказание строения белков;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- Liu X. and Wang X.J. (2020). Potential inhibitors for 2019-nCoV coronavirus M protease from clinically approved medicines. bioRxiv;
- Li Y., Zhang J., Wang N., Li H., Shi Y., Guo G. et al. (2020). Therapeutic drugs targeting 2019-nCoV main protease by high-throughput creening. bioRxiv;
- Eric F. Pettersen, Thomas D. Goddard, Conrad C. Huang, Gregory S. Couch, Daniel M. Greenblatt, et. al.. (2004). UCSF Chimera?A visualization system for exploratory research and analysis. J. Comput. Chem.. 25, 1605-1612;
- Рулетка для спектроскописта;
- Lili Chen, Chunshan Gui, Xiaomin Luo, Qingang Yang, Stephan Günther, et. al.. (2005). Cinanserin Is an Inhibitor of the 3C-Like Proteinase of Severe Acute Respiratory Syndrome Coronavirus and Strongly Reduces Virus Replication In Vitro. JVI. 79, 7095-7103;
- Edward Kim, Zach Jensen, Alexander van Grootel, Kevin Huang, Matthew Staib, et. al.. (2020). Inorganic Materials Synthesis Planning with Literature-Trained Neural Networks. J. Chem. Inf. Model.. 60, 1194-1201;
- Andrew W. Senior, Richard Evans, John Jumper, James Kirkpatrick, Laurent Sifre, et. al.. (2020). Improved protein structure prediction using potentials from deep learning. Nature. 577, 706-710;
- Alex Zhavoronkov, Yan A. Ivanenkov, Alex Aliper, Mark S. Veselov, Vladimir A. Aladinskiy, et. al.. (2019). Deep learning enables rapid identification of potent DDR1 kinase inhibitors. Nat Biotechnol. 37, 1038-1040;
- Zhavoronkov A., Aladinskiy V., Zhebrak A., Zagribelnyy B., Terentiev V., Bezrukov D.S. et al. (2020). Potential COVID-2019 3C-like protease inhibitors designed using generative deep learning approaches. chemRxiv;
- Peter Richardson, Ivan Griffin, Catherine Tucker, Dan Smith, Olly Oechsle, et. al.. (2020). Baricitinib as potential treatment for 2019-nCoV acute respiratory disease. The Lancet. 395, e30-e31;
- Lemonick S. (2020). Two groups use artificial intelligence to find compounds that could fight the novel coronavirus. Chemical and Engineering News. 98, 6;
- Gao K., Nguyen D.D., Wang R., Wei G.W. (2020). Machine intelligence design of 2019-nCoV drugs. bioRxiv;
- Peter Ertl, Ansgar Schuffenhauer. (2009). Estimation of synthetic accessibility score of drug-like molecules based on molecular complexity and fragment contributions. J Cheminform. 1;
- Nguyen D.D., Gao K., Chen J. (2020). Potentially highly potent drugs for 2019-nCoV. bioRxiv;
- Anh-Tien Ton, Francesco Gentile, Michael Hsing, Fuqiang Ban, Artem Cherkasov. (2020). Rapid Identification of Potential Inhibitors of SARS‐CoV‐2 Main Protease by Deep Docking of 1.3 Billion Compounds. Mol. Inf.;
- Gentile F., Agrawal V., Hsing M., Ban F., Norinder U., Gleave M.E., Cherkasov A. (2019). Deep docking — a deep learning approach for virtual screening of big chemical datasets. bioRxiv;
- Beck B.R., Shin B., Choi Y., Choi Y., Park S., Kang K. (2020). Predicting commercially available antiviral drugs that may act on the novel coronavirus (2019-nCoV), Wuhan, China through a drug-target interaction deep learning model. bioRxiv;
- Kevin D. Raney, Suresh D. Sharma, Ibrahim M. Moustafa, Craig E. Cameron. (2010). Hepatitis C Virus Non-structural Protein 3 (HCV NS3): A Multifunctional Antiviral Target. J. Biol. Chem.. 285, 22725-22731;
- Bzówka M., Mitusińska K., Raczyńska A., Samol A., Tuszyński J.A., Góra A. (2020). Structural and evolutionary analysis Indicate that the SARS-CoV-2 Mpro is an inconvenient target for small-molecule inhibitors design. bioRxiv;
- Bin Cao, Yeming Wang, Danning Wen, Wen Liu, Jingli Wang, et. al.. (2020). A Trial of Lopinavir–Ritonavir in Adults Hospitalized with Severe Covid-19. N Engl J Med;
- Huang A., Tang X., Wu H., Zhang J., Wang W., Wang Z. et al. (2020). Virtual screening and molecular dynamics on blockage of key drug targets as treatment for COVID-19 caused by SARS-CoV-2. Preprints;
- Contini A. (2020). Virtual screening of an FDA approved drugs database on two COVID-19 coronavirus proteins. ResearchGate;
- Lan J., Ge J., Yu J., Shan S., Zhou H., Fan S. et al. (2020). Crystal structure of the 2019-nCoV spike receptor-binding domain bound with the ACE2 receptor. bioRxiv;
- Daniel Wrapp, Nianshuang Wang, Kizzmekia S. Corbett, Jory A. Goldsmith, Ching-Lin Hsieh, et. al.. (2020). Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 367, 1260-1263;
- Alexandra C. Walls, Young-Jun Park, M. Alejandra Tortorici, Abigail Wall, Andrew T. McGuire, David Veesler. (2020). Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell;
- Yan R., Zhang Y., Guo Y., Xia L., Zhou Q. (2020). Structural basis for the recognition of the 2019-nCoV by human ACE2. bioRxiv;
- Smith M. and Smith J.C. (2020). Repurposing therapeutics for COVID-19: supercomputer-based docking to the SARS-CoV-2 viral spike protein and viral spike protein-human ACE2 interface. chemRxiv;
- Zhang L. and Zhou R. (2020). Binding mechanism of remdesivir to SARS-CoV-2 RNA dependent RNA polymerase. Preprints;
- Travis K. Warren, Robert Jordan, Michael K. Lo, Adrian S. Ray, Richard L. Mackman, et. al.. (2016). Therapeutic efficacy of the small molecule GS-5734 against Ebola virus in rhesus monkeys. Nature. 531, 381-385;
- Charlotte Harrison. (2020). Coronavirus puts drug repurposing on the fast track. Nat Biotechnol. 38, 379-381;
- Puja Mehta, Daniel F McAuley, Michael Brown, Emilie Sanchez, Rachel S Tattersall, Jessica J Manson. (2020). COVID-19: consider cytokine storm syndromes and immunosuppression. The Lancet. 395, 1033-1034;
- Маджидов Т.И. и Куракин Г.Ф. (2020). Компьютерные технологии против коронавируса: первые результаты. «Природа». 3, 3–15;
- Alexander E. Gorbalenya, Luis Enjuanes, John Ziebuhr, Eric J. Snijder. (2006). Nidovirales: Evolving the largest RNA virus genome. Virus Research. 117, 17-37;
- Peter Forster, Lucy Forster, Colin Renfrew, Michael Forster. (2020). Phylogenetic network analysis of SARS-CoV-2 genomes. Proc Natl Acad Sci USA. 202004999.