
Такие разные синонимы
25 ноября 2014
Такие разные синонимы
- 2463
- 1
- 1
Нелёгкий выбор между кодонами серина, принадлежащими разным кодоновым семействам (подробнее см. раздел «Уникальный серин»).
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Аминокислота — «кирпичик» белковой последовательности — закодирована в геноме тремя нуклеотидами, при этом не всегда единственным образом. Здравый смысл подсказывает, что можно выбрать случайный триплет для данной аминокислоты и что от этого выбора ничего не зависит. Оказывается, здравый смысл в этом случае неправ.
Конкурс «био/мол/текст»-2014
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2014 в номинации «Биоинформатика и молекулярная эволюция».
Главный спонсор конкурса — дальновидная компания «Генотек».
Конкурс поддержан ОАО «РВК».
Спонсором номинации «Биоинформатика» является Институт биоинформатики.
Спонсором приза зрительских симпатий выступила фирма Helicon.
Свой приз также вручает Фонд поддержки передовых биотехнологий.
Вместо предисловия
Науке известно некое обобщение, которое называют «центральной догмой молекулярной биологии». Это правило описывает поток информации на молекулярном уровне: от молекулы дезоксирибонуклеиновой кислоты (ДНК) к рибонуклеиновой кислоте (РНК) и затем к белку. Конечно же, можно привести множество уточнений, усложнений, оговорок и замечаний к этой упрощённой модели, но тем не менее, сформулированная Фрэнсисом Криком в далёком 1958 году, она по сей день является ключом к понимаю того, как «знание», закодированное в последовательности ДНК, превращается в функциональный белок.
Для работы с информацией, содержащейся в ДНК, принято отображать её в виде последовательности мономеров (нуклеотидов), составляющих одну из двух цепей ДНК: A (аденин), G (гуанин), C (цитозин) и T (тимин). Такое представление даёт возможность анализировать информацию, заключённую, например, в гене человеке, с помощью знакомых программистам алгоритмов и методов для работы со строковым типом данных.
Аналогичный алфавит был предложен для записи последовательности белковой молекулы, мономерами которой являются аминокислоты. Для обозначения каждой протеиногенной аминокислоты используется или трёхбуквенное обозначение (на основе первых букв названия аминокислоты, например Ser для серина), или однобуквенное обозначение, разработанное Маргарет Оакли Дэйхофф. Последнее видится вполне разумной оптимизацией: это позволяет в три раза сократить размер пространства, необходимого для хранения последовательности белка, и является более удобным с точки зрения работы с последовательностью как со строкой. Имея последовательность «кирпичиков» ДНК, можно попытаться предсказать, какие элементы — аминокислоты — будут составлять молекулу белка.
Синтез молекулы информационной РНК (мРНК) является первым шагом в рассматриваемой цепи передачи информации: в процессе транскрипции информация, закодированная в последовательности дезоксирибонуклетидов, позволяет получить последовательность рибонуклеиновой кислоты с помощью принципа комплементарности. Это означает, что «буквы» РНК будут соответствовать «буквам» в смысловой цепи ДНК (A → U, C → G, и т.д.).
Сама по себе мРНК является сложной молекулой. Её структура (наличие функциональных элементов) может подвергаться модификации и зависит в том числе от организма, о котором идёт речь (значительны различия между мРНК прокариот и эукариот, — например, наличие протяженных нетранслируемых участков в эукариотических мРНК, практически отсутсвующих у прокариотических). Для нашего дальнейшего анализа будем считать, что нам известна та последовательность мРНК, которая транслируется в последовательность белка. Этот этап передачи информации не похож на транскрипцию, где ключевым был принцип комплементарности: здесь кодирование и декодирование данных осуществляется иначе.
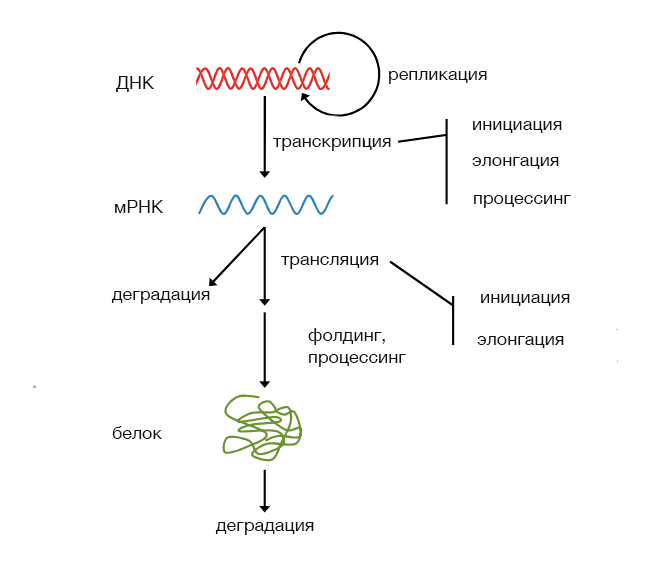
Рисунок 1. Экспрессия и некоторые факторы, влияющие на неё
В качестве деталей для конструирования белков природа выбрала 20 аминокислот. (Здесь стоит отметить существование организмов, некоторые белки которых включают селеноцистеин (Sec) и пирролизин (Pyl). Работы о появлении, эволюции и эволюционном значении этих 21-й и 22-й протеиногенных аминокислот могут показаться интересными и, пожалуй, заслуживают отдельного разговора.) Отсюда возникает требование, вполне естественное и очевидное: для записи в геноме информации о последовательностях белков при таком числе аминокислот необходимо, чтобы одна аминокислота была «зашифрована» в виде как минимум трёх нуклеотидов . На самом деле, именно три нуклеотида (триплет, или кодон — от английского code) кодируют одну аминокислоту. Логическим следствием этой идеи является избыточность генетического кода. Однако с «лишними» 44 (43 − 20 = 44) кодонами можно было бы поступить разными способами: к примеру, не использовать их вовсе или дать возможность нескольким разным кодонам кодировать одну аминокислоту. Последний путь и был избран природой .
Стóит уточнить, что пытливые умы ученых уже создали искусственным путем систему, в которой рибосома при помощи набора «ортогональных» тРНК распознает уже не триплет, а квадруплет, т.е. переключается на кодирование аминокислот не по схеме 43=64, а 44=256: «Слово из четырёх букв» [11]. — Ред.
Возникновение генетического кода почти наверняка не было случайным. По одной из современных гипотез, существует определенное сродство между аминокислотами и кодирующими их триплетами. Возможно, такое сродство лежит в основе механизма матричного синтеза, который в своей первоначальной форме не включал таких «адапторов», какими являются транспортные РНК: «У истоков генетического кода: родственные души» [12]. — Ред.
Информация, содержащаяся в цепи мРНК, считывается по три нуклеотида аппаратом белкового синтеза, при этом важным фактом является свойство неперекрываемости: один нуклеотид входит в состав только одного триплета. Не лишним здесь будет замечание о существовании так называемых стоп-кодонов: считывание UAG, или UGA, или UAA с мРНК даёт сигнал к завершению процесса трансляции. Кстати, эти кодоны имеют красочные названия: янтарь, опал и охра (amber, opal и ochre).
Итак, получается, что изменение некоторых букв генома в определённых позициях не повлечёт за собой замены аминокислот соответствующего белка, которая могла бы сказаться на его свойствах или функции. Существуют даже четырёхкратно вырожденные сайты (fourfold degenerate sites): например, кодоны GGU, GGC, GGA и GGG соответствуют глицину.

Рисунок 2. «Стандартный» генетический код. Отмечены четырёхкратно вырожденные сайты. Аминокислоты отсортированы в алфавитном порядке согласно их латинским названиям.
Синонимы и... не синонимы
Замена буквы в кодоне, конечно, возможна не только в текстовом редакторе, но и в настоящей ДНК. Такие мутации в белок-кодирующих областях генома подразделяют на синонимичные и не синонимичные (некоторые предпочитают использовать синонимы этих слов — «синонимические» и «не синонимические»). Если первые не изменяют аминокислоту, соответствующую кодону, где произошла мутация, то не синонимичные мутации приводят к замене аминокислоты. Такую классификацию мутаций можно использовать для характеристики сайтов (позиций) кодона: например, для кодона CUG первая позиция является на 1/3 синонимичной (UUG, как и CUG, кодирует лейцин) и на 2/3 не синонимичной (AUG и GUG кодируют метионин и валин, соответственно).
Идея о равноценности кодонов, кодирующих одну аминокислоту (иногда их именуют синонимичными кодонами), кажется не противоречащей логике. Действительно, если взглянуть на проблему с высоты центральной догмы молекулярной биологии, то вполне разумно будет заявить об отсутствии эффекта синонимичных мутаций на последовательность белка, а значит и на функцию его в клетке и его эволюционную судьбу. Однако такая гипотеза явно не соответствует реальности: если подсчитать доли синонимичных кодонов в каком-нибудь известном геноме, то вычисленные частоты наверняка будут значимо различаться. Таким образом, налицо предпочтение одних синонимичных кодонов другим, что не имеет, на первый взгляд, никакого смысла. Этот феномен назвали смещением частоты кодонов (codon-usage bias, далее СЧК), и суть его заключается именно в статистическом отклонении использования синонимичных кодонов от равномерного.
Зачем клетке редкие и частые кодоны?
Причины СЧК так или иначе затронуты в десятках (и даже сотнях) публикаций, поэтому важно рассмотреть основные идеи, лежащие в основе различных гипотез. Определённые паттерны использования кодонов наблюдаются в геномах организмов, принадлежащих различным систематическим группам: бактерий, архей, червей, млекопитающих и т.д. Для объяснения наблюдаемых паттернов можно привлечь две классические модели. Первая из них предлагает связывать СЧК с мутационным процессом; так, можно искать объяснение СЧК в смещённых частотах нуклеотидов, рождаемых точечными мутациями или системами репарации. Эту модель можно назвать «нейтральной», в отличие от второй, которая привлекает к объяснению понятие естественного отбора и утверждает, что синонимичные мутации могут влиять на приспособленность организма. И нейтральный, и селективный механизм, вероятно, играют роль в создании той картины СЧК, которую можно наблюдать как между различными видами, так и в пределах одного генома между генами.
Рассмотрение СЧК для генов одного генома становится особенно интересным в свете следующего факта: уровень экспрессии генов в ряде видов положительно коррелирует со степенью СЧК. Вероятно, явление СЧК наиболее выражено в последовательностях генов с высокими уровнями экспрессии для повышения эффективности или точности трансляции. Это заявление стоит рассматривать как гипотезу, которая остаётся темой дискуссий и споров. Так, подобное объяснение не согласуется с представлением, что именно инициация (а не элонгация) является основным фактором, ограничивающим скорость синтеза белка в клетке (подробнее см. [1]).
СЧК может частично объясняться и другими факторами, например вторичной структурой мРНК. В качестве примера можно привести гены алкогольдегидрогеназы плодовой мухи: вторичная структура их мРНК более стабильна для генов с более низкими уровнями экспрессии. Предполагается, что отбор в пользу «сильной» вторичной структуры может использоваться для снижения уровня экспрессии гена и наоборот.
Ещё одним примером того, как синонимическая мутация может влиять на стабильность мРНК, являются мутации в гене дофаминового рецептора человека DRD2. В последовательности этого гена обнаружено 6 разных полиморфизмов (SNP, single nucleotide polymorphism, или «снипов», как их называют), но лишь одна мутация, уменьшающая время жизни молекулы мРНК, связана с нарушением экспрессии гена DRD2, которая регулируется дофамином [2]. Эта мутация — C957T, т. е. замена девятьсот пятьдесят седьмой буквы последовательности с C на T, — приводит к заметным изменениям в модели вторичной структуры мРНК. Кстати, интересно, что при сравнении частоты кодонов в последовательностях гена DRD2 и его «соседей» по хромосоме обнаружилось смещение в частоте использования нуклеотидов в третьей позиции кодонов гена DRD2 в пользу G и C.
Как известно, мРНК эукариот, будучи синтезированной, подвергается процессингу, а лишь затем транслируется. Сплайсинг РНК является одним из этапов процессинга и состоит в вырезании «ненужных» участков пре-мРНК — интронов. Оказывается, науке известно большое число заболеваний, связанных с нарушением процесса сплайсинга в результате синонимических мутаций. В их списке можно найти, например, синдром Марфана, фенилкетонурию и множественный склероз. Предполагается, что эти мутации могут создавать новые сайты сплайсинга или влиять на элементы последовательности, важные для контроля процесса сплайсинга (так называемые энхансеры и сайленсеры сплайсинга — exonic splicing enhancers and silencers). Наблюдаемые явления СЧК, таким образом, могут частично объясняться отбором, действующим на последовательности этих модуляторов сплайсинга.
Традиционно сплайсинг считается особенностью именно молекул РНК. Однако оказывается, что у белков тоже бывает сплайсинг, причем в этом случае процесс протекает спонтанно (то есть, автокаталитически): «Белки против РНК — кто первым придумал сплайсинг?» [13]. — Ред.
Любопытно, что даже в пределах одного гена выявлено явление СЧК. Например, возможно использование менее адаптированных (к набору изоакцепторных тРНК) кодонов в тех сайтах, где для правильной ко-трансляционной укладки белка необходима врéменная остановка рибосомы. Обнаружено также явление автокорреляции кодонов в последовательности гена, позволяющее, как предполагают авторы этого наблюдения, повысить эффективность элонгации посредством механизма повторного использования молекул тРНК, «недавно» задействованных в процессе трансляции.
В поисках оптимальных кодонов
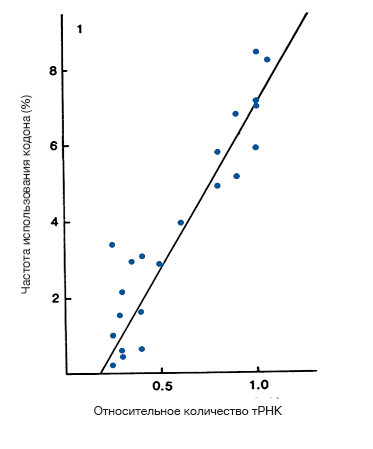
Рисунок 3. Взаимосвязь между частотой использования кодона и количеств тРНК для некоторых генов с сильным смещением использования кодонов от равномерного
Вполне естественной в свете всего вышесказанного выглядит идея организовать набор «хороших» кодонов, а заодно и определиться с критериями для этого. Согласно одной из теорий, преимущество таких кодонов перед их синонимичными собратьями заключается в оптимальной стабильности кодон-антикодонового взаимодействия: молекула тРНК будет оптимально подходить для кодона, если GC-состав (доля нуклеотидов G и C в последовательности) для такого взаимодействия будет промежуточным, т. е. находиться между экстремальными значениями слабых взаимодействий A—U (две водородные связи) и сильных пар G—C (три водородные связи). Однако такая теория не поддержана экспериментальными данными. Смещение частоты кодонов в сторону именно кодон-антикодоновых пар промежуточной стабильности не наблюдается, к тому же для разных организмов свойственны различные наборы «хороших» кодонов.
Но как же тогда природа выбирает оптимальные кодоны? Предполагают, что оптимизация эффективности трансляции возможна для любого выбранного кодона, например, с помощью подбора концентраций тРНК. В таком случае нет причин полагать, что свойства кодон-антикодоновых взаимодействий являются основой для выбора оптимальных кодонов.
Мухи и люди
Примечательно, что использование кодонов в геномах млекопитающих имеет много отличий в сравнении с другими таксонами. Различия использования кодонов между генами млекопитающих объясняются в первую очередь вариацией в GC-составе на уровне генома: оказывается, для генома млекопитающих свойственно наличие изохор — крупных (длиной >300 тысяч оснований) фрагментов хромосом, характеризующихся однородным GC-составом. При этом GC-состав между изохорами различается. Точнее всего предсказать нуклеотидный состав синонимичных сайтов и смещение частоты кодонов для определённого гена удаётся именно по нуклеотидному составу изохоры, которой этот ген принадлежит. Это можно считать подтверждением того, что отбор, действующий на гены или экзоны, не является главной движущей силой для синонимических мутаций.
Действие отбора на использование кодонов млекопитающими подвергается сомнению по теоретическим соображениям: эффективный размер популяции млекопитающих мал, что ограничивает эффективность отбора. В этой связи следует упомянуть теорию «почти нейтральных» мутаций. Если нейтральная теория предполагает скорость эволюции равной частоте нейтральных мутаций, то «почти нейтральная» теория основана на идее отрицательной корреляции между скоростью эволюции и эффективным размером популяции вида. Так, если негативный эффект s, оказываемый мутацией, невелик по сравнению с эффективным размером популяции Ne (s << 1/Ne), то её называют «эффективно нейтральной».Таким образом, мутация, «вредная» для плодовой мухи, может быть эффективно нейтральной для млекопитающих, так как для последних значение Ne много меньше. Поэтому предполагается маловероятным влияние естественного отбора на синонимические мутации для видов, популяции которых характеризуются Ne << 1 000 000, однако для некоторых биологических объектов, таких как бактерии и мухи, это влияние будет прослеживаться.
Уникальный серин
Давайте сделаем небольшое отступление и снова обратим взор на генетический код. Структура его такова, что лишь одна из аминокислот — серин — кодируется двумя наборами триплетов (TCN, где N — любое основание, и AGY, где Y — пиримидиновое основание, то есть C или T), для перехода между которыми недостаточно однонуклеотидной мутации. Так как вероятность одновременного мутирования двух позиций кодона считается малой, то предполагалось, что если остаток серина важен для функции белка, то соответствующий кодон будет «заперт» в одном из кодоновых семейств. Однако случаи «переключения» между этими кодоновыми семействами в кодирующих последовательностях были обнаружены, например, при изучении эволюции генов убиквитина — высококонсервативного белка, играющего ключевую роль в процессах деградации белков клетки [14], [15].
Было подсчитано, что если подобные переключения действительно имели место в эволюции, то происходили они с частотой, много превышающей частоту двух случайных мутаций в соседних сайтах. Так было предложено одно из возможных объяснений — динуклеотидные мутации. Другое объяснение не привлекает такие мутации, а основано на двух последовательных мутациях: «вредной», заменяющей сериновый кодон на треониновый (ACN) или цистеиновый (TGY), и «полезной», возвращающий серин «на место». При этом вторая мутация может привести к кодону другого семейства (AGY → ACY → TCY или TCY → TGY → AGY) или восстановить в последовательности прежний кодон. Эта гипотеза, однако, не отвечает на вопрос о том, как промежуточный (несериновый) кодон сохраняется в популяции.
Как взвесить тРНК?
Оптимальные кодоны для некоторых видов, в том числе для плодовой мухи Drosophila melanogaster и круглого червя Caenorhabditis elegans, соответствуют наиболее представленным тРНК, т.е. такие кодоны более всего адаптированы к набору изоакцепторных тРНК этих видов. Уместно отметить, что оценку количества тРНК можно производить по-разному. Наиболее прямой подход — использовать экспериментальные данные о количестве молекул тРНК в клетках ткани организма. Однако проведение таких экспериментов требует большого количества времени, материальных ресурсов и усилий исследователей. К счастью, есть способ косвенной оценки представленности тРНК: по количеству копий соответствующих генов в геноме интересующего организма. И хотя этот подход требует наличия последовательности всего генома, в наше время, когда количество данных полногеномного секвенирования возрастает экспоненциально, это вряд ли можно назвать проблемой [16–18].
Вычислительный аспект
Взгляд на феномен СЧК будет неполным, если не затронуть некоторые аспекты статистического и компьютерного анализа, связанные с тематикой вопроса. Так, необходимостью сравнения использования кодонов в последовательностях (разных генов и/или видов) продиктовано использование показателя RSCU (относительного использования синонимичных кодонов, the relative synonymous codon usage). RSCU может принимать значения от 0 (кодон отсутствует) до 6 (используется один кодон из 6 синонимичных). Если смещения частоты использования в семействе синонимичных кодонов не наблюдается, то значение RSCU равно единице. Формально RSCU определяют как отношение наблюдаемой частоты кодона к его частоте, ожидаемой в предположении равномерного использования синонимичных кодонов.
При вычислении RSCU игнорируются стоп-кодоны; для метионина и триптофана (каждую из этих аминокислот кодирует лишь один кодон) значение RSCU постоянно, поэтому RSCU для кодонов AUG и UGG также может быть опущено. Таким образом, для гена можно рассчитать RSCU по всем кодонам (так получится набор из 59 значений: 64 кодона − 3 стоп-кодона − 2 кодона [Met, Trp]). Если выполнить подобные вычисления для набора генов (например, некоторой выборки генов человека), можно изобразить полученные результаты в виде тепловой карты (рис. 4).
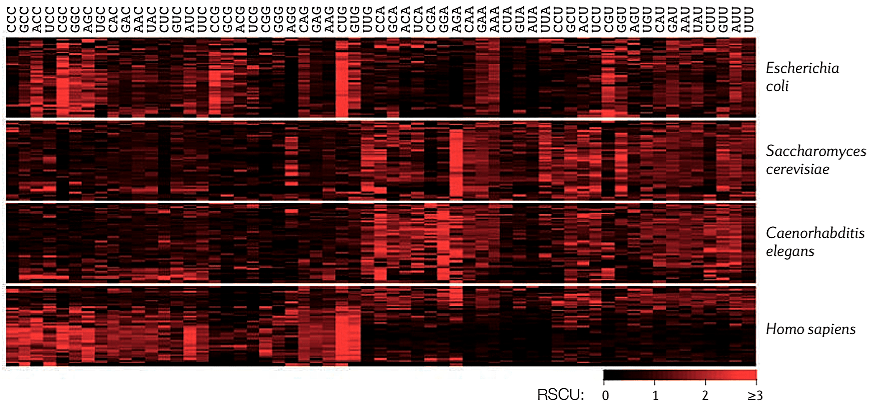
Рисунок 4. Относительное использование синонимичных кодонов (RSCU) для 50 случайно выбранных генов нескольких видов. Ясно прослеживается смещение частоты кодонов как в пределах одного генома, так и между геномами
На RSCU основано вычисление других показателей, например, относительной приспособленности кодона (w), которая вычисляется для определённых аминокислоты и кодона как отношение RSCU этого кодона к RSCU оптимального кодона для этой аминокислоты. Значение w позволяет получить индекс адаптации кодонов (the codon adaptation index, CAI), представляющий собой среднее геометрическое значений w для гена. Для Escherichia coli (кишечная палочка) было показано, что значения CAI генов рибосомных белков, как правило, высокие, в то время как для генов с низкими уровнями экспрессии характерны низкие значения CAI. Этот индекс может использоваться для предсказания уровней экспрессии генов определённого вида и гетерологичных генов (генов другого вида), сравнения использования кодонов разными организмами, идентификации рамок считывания, оценки скорости молекулярной эволюции или как мера адаптации использования кодонов в процессе эволюции (последнее представляет интерес, например, для вирусных генов).
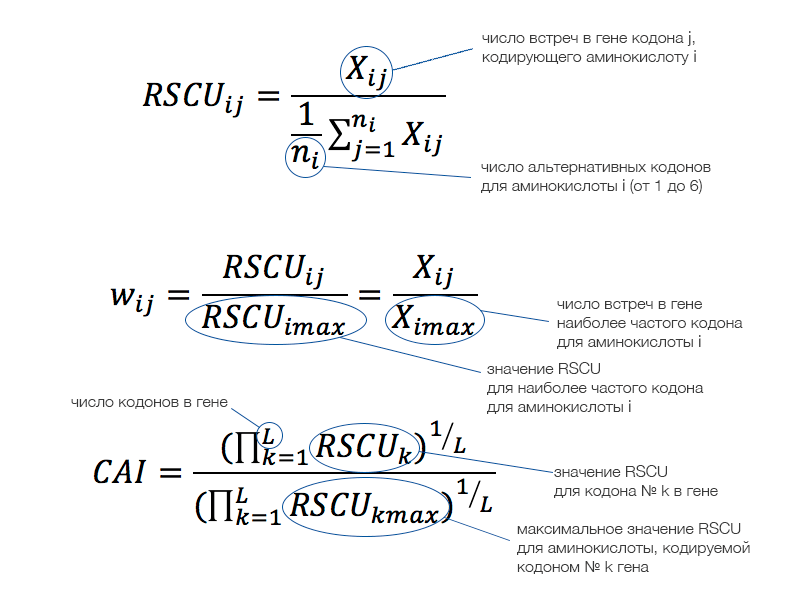
Рисунок 5. Величины, используемые для оценки смещения частоты кодонов, и способы их вычисления
Стоит упомянуть также, такие способы оценки СЧК, как FOP (частота оптимальных кодонов, the frequency of optimal codons) и tAI (индекс адаптации к тРНК, the tRNA adaptation index). FOP представляет собой долю оптимальных кодонов среди всех кодонов гена. Как было показано для кишечной палочки, более высокие значения FOP присущи генам с высокими уровнями экспрессии. Создатели tAI, развивая идею CAI, предложили оценку адаптации гена к пулу тРНК организма. Индекс адаптации к тРНК учитывает число изоакцепторных тРНК для каждого кодона, число копий гена каждой тРНК, а также эффективность кодон-антикодоновых взаимодействий. Значение tAI коррелирует с уровнями экспрессии генов: наибольшие значения tAI принимает для высоко экспрессируемых генов.
Прикладное значение
По традиции, в завершение стоит обозначить применимость затронутых теоретических аспектов (что-то вроде «СЧК вокруг нас»). Во-первых, как уже было упомянуто, синонимические мутации и отбор, действующий на них, связаны с различными заболеваниями (их неполный список можно найти, например, в [4]). При этом во многих случаях точный механизм, по которому синонимические мутации приводят к аберрантному сплайсингу, вызывающему заболевания, ещё не известен. Итак, синонимические мутации могут иметь, если говорить более строгим языком, патофизиологическое и фармакогенетическое значение.
Второй областью, где можно встретить рассматриваемое явление, является искусственное СЧК. Основой его является возможность «подгонки» кодонного состава гена под использование кодонов интересующего организма. Например, такая «подгонка» позволила добиться лучших результатов в экспрессии зелёного флуоресцентного белка GFP в клетках человека. «Одомашненный» CЧК даёт надежду на возможность улучшения характеристик трансгенов — чужеродных молекул ДНК, внедряемых в клетку, — без изменения белка, кодируемого ими. В данном случае, как и на протяжении истории человечества, ответ на вопрос «А как это делает природа?» позволяет добиться практического результата «своими руками»: так, знание направления действия отбора на синонимические позиции в модуляторах сплайсинга позволяет увеличить время жизни транскрипта. Оптимизацию последовательности трансгена можно проводить in silico — методами компьютерного моделирования и анализа (например, см. [5]). На данный момент, однако, их применение ограничено видами, для которых хорошо изучены отклонения в концентрациях тРНК, такими как бактерии и дрожжи .
О том, что такое GFP и об их разнообразии см. в статье «Флуоресцентные белки: разнообразнее, чем вы думали!» [19]. — Ред.
Современные исследования показывают, что СЧК позволяет контролировать экспрессию генов не только на уровне трансляции, но также транскрипции. В последнем случае, СЧК позволяет модулировать специфичность взаимодействия ряда транскрипционных факторов с экзонными (кодирующими) участками генов: «Таинственный код нашего генома» [20]. — Ред.
Литература
- Joshua B. Plotkin, Grzegorz Kudla. (2011). Synonymous but not the same: the causes and consequences of codon bias. Nat Rev Genet. 12, 32-42;
- J. Duan. (2003). Synonymous mutations in the human dopamine receptor D2 (DRD2) affect mRNA stability and synthesis of the receptor. Human Molecular Genetics. 12, 205-216;
- Andersson S.G. and Kurland C.G. (1990). Codon preferences in free-living microorganisms. Microbiol. Rev. 54, 198–210;
- J. V. Chamary, Joanna L. Parmley, Laurence D. Hurst. (2006). Hearing silence: non-neutral evolution at synonymous sites in mammals. Nat Rev Genet. 7, 98-108;
- Mark Welch, Alan Villalobos, Claes Gustafsson, Jeremy Minshull. (2009). You're one in a googol: optimizing genes for protein expression. J. R. Soc. Interface.. 6;
- Sharp P.M., Averof M., Lloyd A.T., Matassi G., Peden J.F. (1995). DNA sequence evolution: the sounds of silence. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 349, 241-247;
- Paul M. Sharp, Wen-Hsiung Li. (1987). The codon adaptation index-a measure of directional synonymous codon usage bias, and its potential applications. Nucl Acids Res. 15, 1281-1295;
- Toshimichi Ikemura. (1981). Correlation between the abundance of Escherichia coli transfer RNAs and the occurrence of the respective codons in its protein genes: A proposal for a synonymous codon choice that is optimal for the E. coli translational system. Journal of Molecular Biology. 151, 389-409;
- M. d. Reis. (2004). Solving the riddle of codon usage preferences: a test for translational selection. Nucleic Acids Research. 32, 5036-5044;
- L Duret. (2002). Evolution of synonymous codon usage in metazoans. Current Opinion in Genetics & Development. 12, 640-649;
- Слово из четырёх букв;
- У истоков генетического кода: родственные души;
- Белки против РНК — кто первым придумал сплайсинг?;
- Вездесущий убиквитин;
- «Вездесущий убиквитин» возвращается;
- Код жизни: прочесть не значит понять;
- Перевалило за тысячу: третья фаза геномики человека;
- Технология: $1000 за геном;
- Флуоресцентные белки: разнообразнее, чем вы думали!;
- Таинственный код нашего генома.







