12 методов в картинках: структурная биология
21 апреля 2017
12 методов в картинках: структурная биология
- 17034
- 4
- 54
рисунок Ольги Пташник.
-
Авторы
-
Редакторы
-
Иллюстратор
Науки о жизни идут по пути от крупного к мелкому. Совсем недавно биология описывала исключительно внешние черты животных, растений, бактерий. Молекулярная биология изучает живые организмы на уровне взаимодействий отдельных молекул. Биология структурная — исследует процессы в клетках на уровне атомов. Если хотите узнать, как «увидеть» отдельные атомы, как работает и «живет» структурная биология и какие использует приборы, вам сюда!
12 биологических методов в картинках

Генеральный партнер цикла — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Одна из главных миссий «Биомолекулы» — докопаться до самых корней. Мы не просто рассказываем, какие новые факты обнаружили исследователи — мы говорим о том, как они их обнаружили, стараемся объяснить принципы биологических методик. Как вытащить ген из одного организма и вставить в другой? Как проследить в огромной клетке за судьбой нескольких крошечных молекул? Как возбудить одну крохотную группу нейронов в огромном мозге?
И вот мы решили рассказать о лабораторных методах более системно, собрать воедино в одной рубрике самые главные, самые современные биологические методики. Чтоб было интереснее и нагляднее, мы густо проиллюстрировали статьи и даже кое-где добавили анимации. Мы хотим, чтобы статьи новой рубрики были интересны и понятны даже случайному прохожему. И с другой стороны — чтобы они были так подробны, что даже профессионал мог бы обнаружить в них что-то новое. Мы собрали методики в 12 больших групп и собираемся сделать на их основе биометодический календарь. Ждите обновлений!
Зачем нужна структурная биология?
Как известно, биология — это наука о жизни. Появилась она в самом начале XIX века и первые сто лет своего существования была сугубо описательной. Главной задачей биологии в то время считали найти и охарактеризовать как можно большее количество видов различных живых организмов, чуть позже — выявить родственные связи между ними. Со временем и с развитием других областей науки из биологии выделились несколько ветвей с приставкой «молекулярный»: молекулярная генетика, молекулярные биология и биохимия — науки, изучающие живое на уровне отдельных молекул, а не по внешнему виду организма или взаиморасположению его внутренних органов. Наконец, совсем недавно (в 50-х годах прошлого века) появилась такая область знания, как структурная биология — наука, изучающая процессы в живых организмах на уровне изменения пространственной структуры отдельных макромолекул. По сути, структурная биология находится на стыке трех различных наук. Во-первых, это биология, потому что наука изучает живые объекты, во-вторых, физика, поскольку используется широчайший арсенал физических экспериментальных методов, а в-третьих, химия, так как изменение структуры молекул — объект именно этой дисциплины.
Структурная биология изучает два основных класса соединений — белки (основное «рабочее тело» всех известных организмов) и нуклеиновые кислоты (главные «информационные» молекулы). Именно благодаря структурной биологии мы знаем, что ДНК имеет структуру двойной спирали, что тРНК нужно изображать в виде винтажной буквы «Г», а в рибосоме есть большая и малая субъединицы, состоящие из белков и РНК в определенной конформации.
Глобальная цель структурной биологии, как и любой другой науки, — «понять, как все устроено». В какую форму свернута цепь белка, который заставляет клетки делиться, как изменяется упаковка фермента во время химического процесса, который он осуществляет, какими местами взаимодействуют гормон роста и его рецептор — вот вопросы, на которые отвечает эта наука. Более того, отдельной целью является накопление такого объема данных, чтобы на эти вопросы (по еще неизученному объекту) можно было ответить на компьютере, не прибегая к дорогостоящему эксперименту.

Например, нужно понять, как работает система биолюминесценции у червей [1] или грибов — расшифровали геном, на основании этих данных нашли нужный белок и предсказали его пространственную структуру вместе с механизмом работы. Стоит, правда, признать, что пока такие методы существуют только в зачаточной стадии, и точно предсказать структуру белка, имея только его ген, еще невозможно [2]. С другой стороны, результаты структурной биологии имеют применение в медицине. Как надеются многие исследователи, знания о структуре биомолекул и о механизмах их работы позволят разрабатывать новые лекарства на рациональной базе, а не методом проб и ошибок (высокопроизводительного скрининга, если говорить строго), как это делается чаще всего сейчас. И это не научная фантастика: уже есть много лекарств, созданных или оптимизированных с применением структурной биологии [3].
История структурной биологии
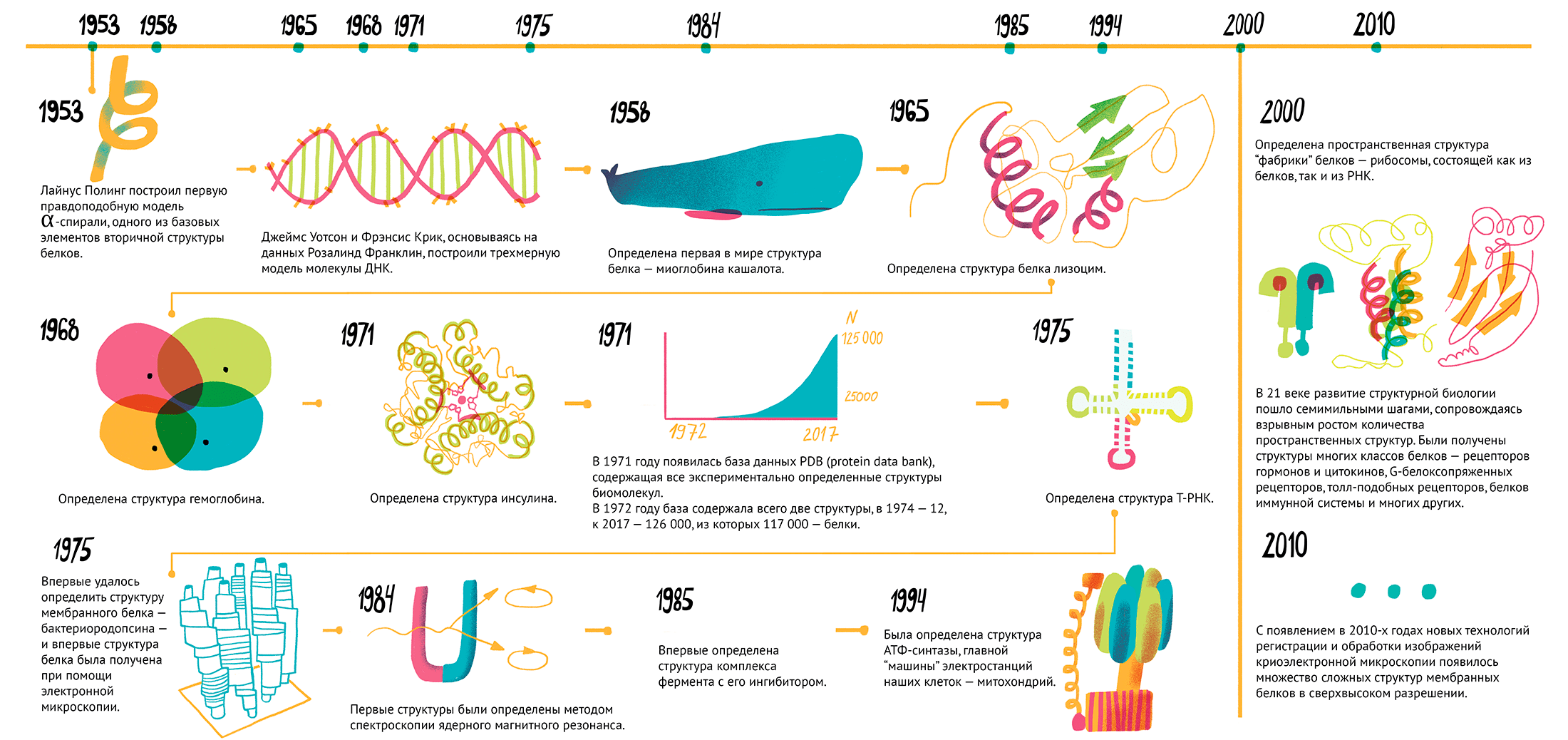
Рисунок 1. Важные этапы на пути структурной биологии. Чтобы увидеть рисунок в полном размере, нажмите на него.
Рисунок Ольги Пташник.
История структурной биологии (рис. 1) достаточно коротка и стартует в начале 1950-х, когда Джеймс Уотсон и Фрэнсис Крик, основываясь на данных Розалинд Франклин по дифракции рентгеновских лучей на кристаллах ДНК, собрали из винтажного конструктора модель известной сейчас всем двойной спирали [4]. Чуть раньше Лайнус Полинг построил первую правдоподобную модель α-спирали, одного из базовых элементов вторичной структуры белков (рис. 2) [5].
Спустя пять лет, в 1958 году, была определена первая в мире структура белка — миоглобина (белка мышечных волокон) кашалота (рис. 3). Выглядела она, конечно, не так красиво, как современные структуры, но это была значительная веха развития современной науки.
Следующая структура белка (лизоцима) появилась лишь через семь лет, но затем события ускорились. В 1968 году была получена структура гемоглобина, в 1971 — инсулина, в 1975 году получили структуру транспортной РНК (рис. 4).
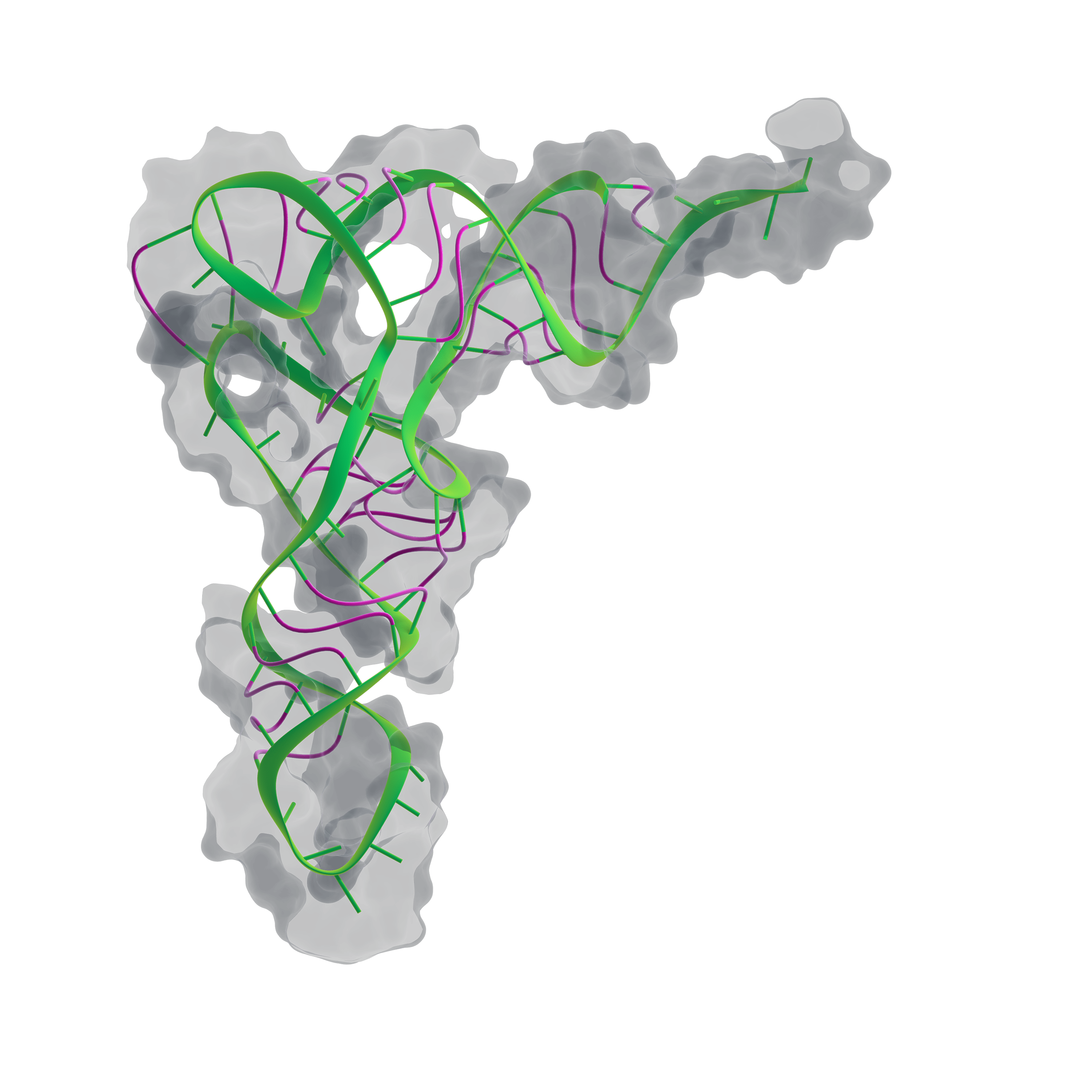
Рисунок 4. Пространственная структура транспортной РНК.
Visual Science
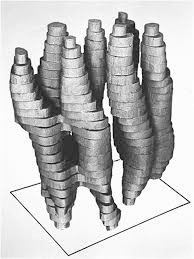
Рисунок 5. Структура бактериородопсина, полученная на основе данных электронной микроскопии.
Важнейшее событие для структурных биологов произошло в 1971 году: появилась база данных PDB (Protein Data Bank), которая сейчас содержит все пространственные структуры белков, которые были получены различными методами. В 1972 году база данных содержала всего две структуры, в 1974 — 12, сегодня в ней можно найти 126 000 различных пространственных структур, из которых 117 000 — белки. В 1975 году произошло целых два «впервые». Впервые удалось получить структуру мембранного белка — бактериородопсина, — и сделали это впервые при помощи электронной микроскопии (рис. 5) [7].
Через 10 лет, в 1984–1985 годах, первые структуры определили методом спектроскопии ядерного магнитного резонанса. С того момента произошло несколько ключевых открытий: в 1985 году получили структуру первого комплекса фермента с его ингибитором, в 1994 году определили структуру АТФ-синтазы [8], главной «машины» электростанций наших клеток (митохондрий), а уже в 2000 г. получили первую пространственную структуру «фабрики» белков — рибосомы, состоящей из белков и РНК (рис. 6) [9]. В 21 веке развитие структурной биологии пошло семимильными шагами, сопровождаясь взрывным ростом количества пространственных структур. Были получены структуры многих классов белков: рецепторов гормонов и цитокинов, G-белоксопряженных рецепторов, толл-подобных рецепторов, белков иммунной системы и многих других [10–15].
С появлением в 2010-х годах новых технологий регистрации и обработки изображений криоэлектронной микроскопии появилось множество сложных структур мембранных белков в сверхвысоком разрешении [16], [17]. Прогресс структурной биологии не остался незамеченным: за открытия в данной области были вручены 14 нобелевских премий, из них пять — уже в 21 веке.
Методы структурной биологии
Исследования в области структурной биологии ведут при помощи нескольких физических методов, из которых только три позволяют получать пространственные структуры биомолекул в атомарном разрешении. Методы структурной биологии основаны на измерении взаимодействия исследуемого вещества с различными видами электромагнитных волн или элементарных частиц. Все методики требуют значительных финансовых ресурсов — стоимость оборудования часто поражает воображение.

Рентгеноструктурный анализ

Рисунок 7. Общая схема рентгеноструктурного анализа.
Рисунок Ольги Пташник.
Исторически первый метод структурной биологии — рентгеноструктурный анализ (РСА) (рис. 7). Еще в начале 20 века выяснили, что по картине дифракции рентгеновских лучей на кристаллах можно изучать их свойства — тип симметрии ячейки, длину связей между атомами и пр. Если же в ячейках решетки кристаллов находятся органические соединения, то можно вычислить координаты атомов, и, следовательно, химическую и пространственную структуру этих молекул. Именно так была получена в 1949 году структура пенициллина, а в 1953 году — структура двойной спирали ДНК.
Казалось бы, все просто, но есть нюансы.
Во-первых, нужно как-то получить кристаллы, причем их размер должен быть достаточно большим (рис. 8). Если для не очень сложных молекул это выполнимо (вспомните, как кристаллизуются поваренная соль или медный купорос!), то кристаллизация белков — это сложнейшая задача, требующая неочевидной процедуры поиска оптимальных условий. Сейчас это делается при помощи специальных роботов, которые готовят и мониторят сотни различных растворов в поисках «проросших» кристаллов белков [18], [19]. Однако на заре кристаллографии получение кристалла белка могло занимать годы ценного времени.
Во-вторых, на основе полученных данных («сырых» дифракционных картин; рис. 8) нужно структуру «рассчитать». Сейчас это также рутинная задача, однако 60 лет назад, в эру ламповой техники и перфокарт, было далеко не так просто.
В-третьих, даже если получилось вырастить кристалл, то совсем не обязательно, что будет определена пространственная структура белка: для этого во всех узлах решетки белок должен иметь одну и ту же структуру, что далеко не всегда так.
Ну и в-четвертых, кристалл — далеко не естественное состояние белка. Изучать белки в кристаллах — это как изучать людей, впихнув их вдесятером в малогабаритную прокуренную кухню: можно узнать, что у людей есть руки, ноги и голова, но поведение может быть не совсем таким, как в комфортной обстановке. Тем не менее, рентгеноструктурный анализ — это наиболее распространенный метод определения пространственных структур, и 90% содержимого PDB получено при помощи этого метода.
РСА требует мощных источников рентгеновских лучей — ускорителей электронов или лазеров на свободных электронах (рис. 9). Такие источники стоят дорого — несколько миллиардов долларов США, — но обычно один источник используют сотни или даже тысячи групп по всему миру за достаточно символическую плату. В нашей стране мощных источников нет, поэтому большинство ученых ездит из России в США или Европу для анализа полученных кристаллов. Подробнее об этих романтичных исследованиях можно прочесть в статье «Лаборатория перспективных исследований мембранных белков: от гена к ангстрему» [20].
Устройство лазера на свободных электронах

Как уже было сказано, для рентгеноструктурного анализа необходим мощный источник рентгеновского излучения. Чем мощнее источник, тем меньшим размером кристаллов можно обойтись, и тем меньше мучений придется испытать биологам и генным инженерам, пытающимся получить несчастные кристаллы. Рентгеновское излучение проще всего получить, ускоряя пучок электронов в синхротронах или циклотронах — гигантских кольцевых ускорителях. Когда электрон испытывает ускорение, он излучает электромагнитные волны в нужном диапазоне частот. В последнее время появились новые сверхмощные источники излучения — лазеры на свободных электронах (XFEL) [21].
Принцип работы лазера достаточно простой (рис. 9). Сначала электроны разгоняются до высоких энергий при помощи сверхпроводящих магнитов (длина ускорителя 1–2 км), а потом проходят через так называемые ондуляторы — наборы магнитов разной полярности.
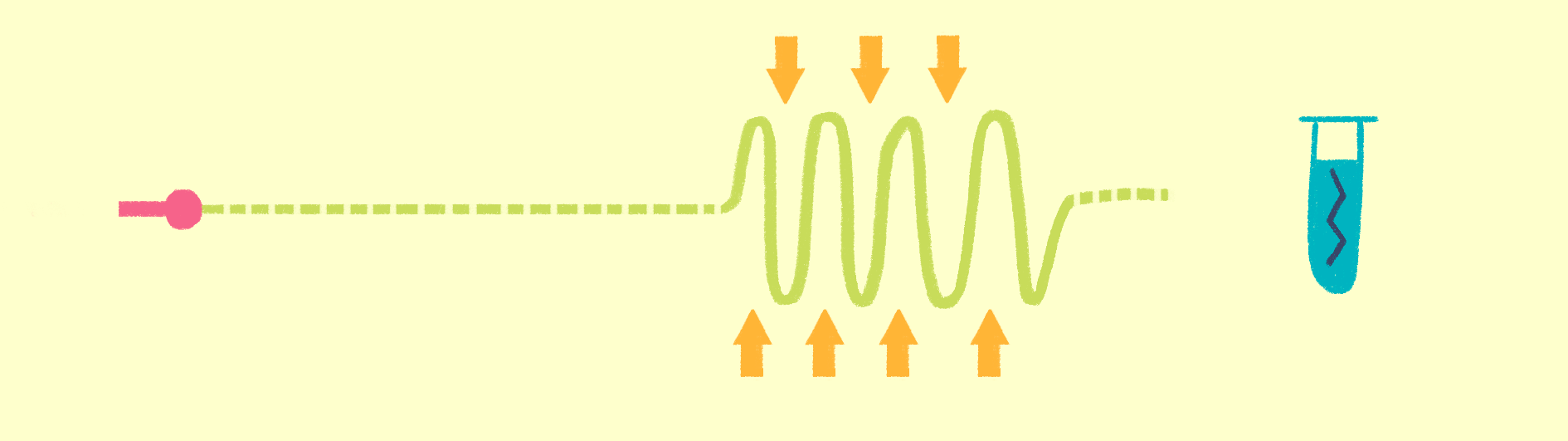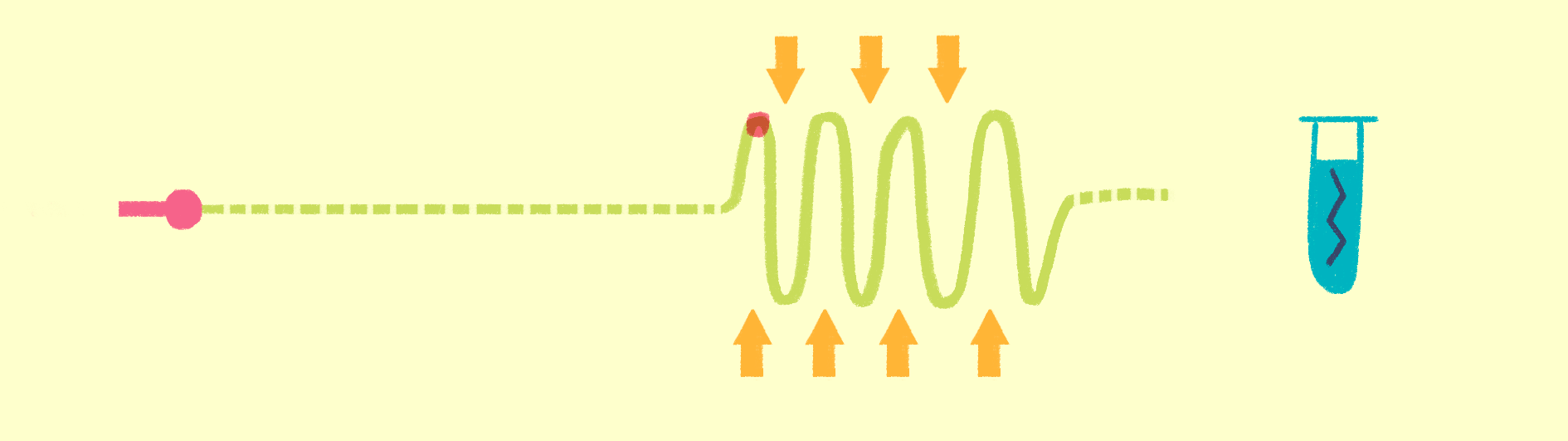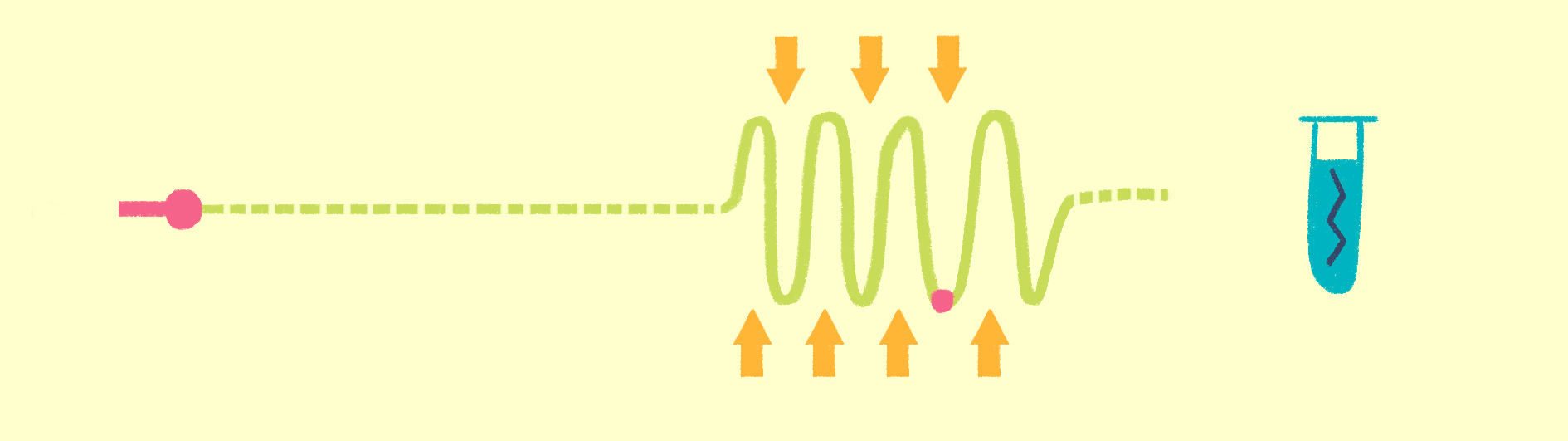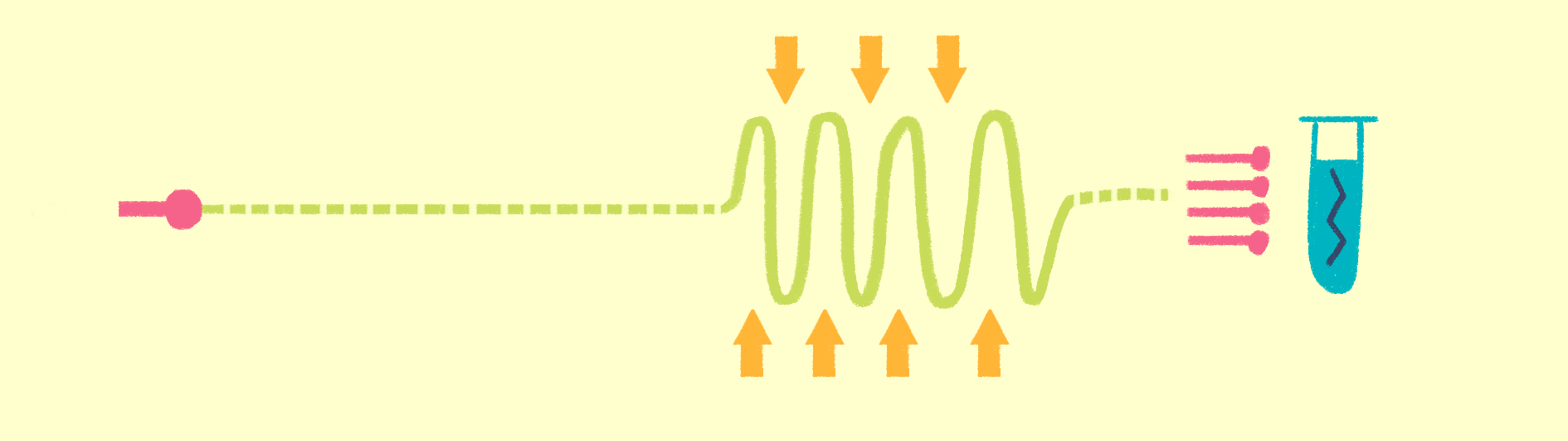
Рисунок 9. Принцип работы лазера на свободных электронах. Пучок электронов ускоряется, проходит через ондулятор и излучает гамма-кванты, которые попадают на биологические образцы.
Рисунок Ольги Пташник.
Проходя через ондулятор, электроны начинают периодически отклоняться от направления пучка, испытывая ускорение и испуская рентгеновское излучение. Поскольку все электроны движутся одинаково, то излучение усиливается за счет того, что другие электроны пучка начинают поглощать и переизлучать рентгеновские волны одной и той же частоты. Все электроны испускают излучение синхронно в виде сверхмощной и очень короткой вспышки (длительностью менее 100 фемтосекунд). Мощность рентгеновского луча настолько высока, что одна короткая вспышка превращает небольшой кристалл в плазму (рис. 10) [22], однако за те несколько фемтосекунд, пока кристалл цел, можно получить изображение высочайшего качества благодаря высокой интенсивности и когерентности луча. Стоимость такого лазера составляет 1,5 миллиарда долларов, а в мире работает всего четыре такие установки (находятся в США (рис. 11), Японии, Корее и Швейцарии). В 2017 году планируется ввод в эксплуатацию пятого — европейского — лазера, в строительстве которого участвовала и Россия.
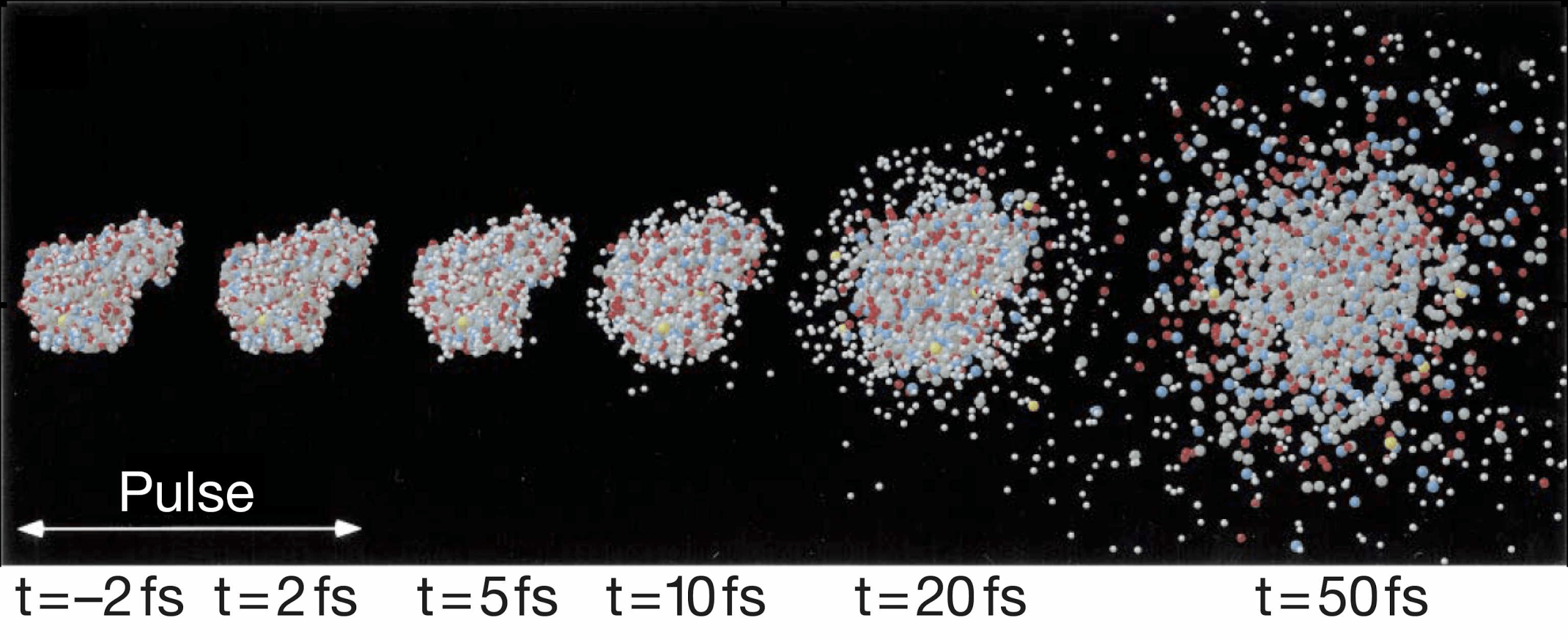
Рисунок 10. Превращение белков в плазму за 50 фс под действием импульса лазера на свободных электронах. Фемтосекунда = 1/1000000000000000 доли секунды.
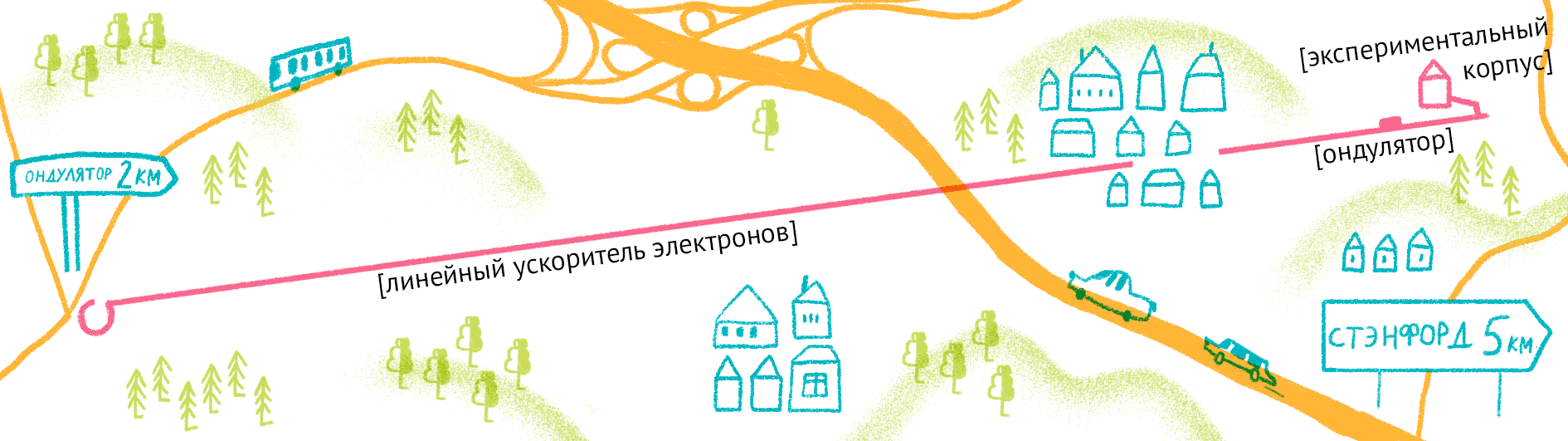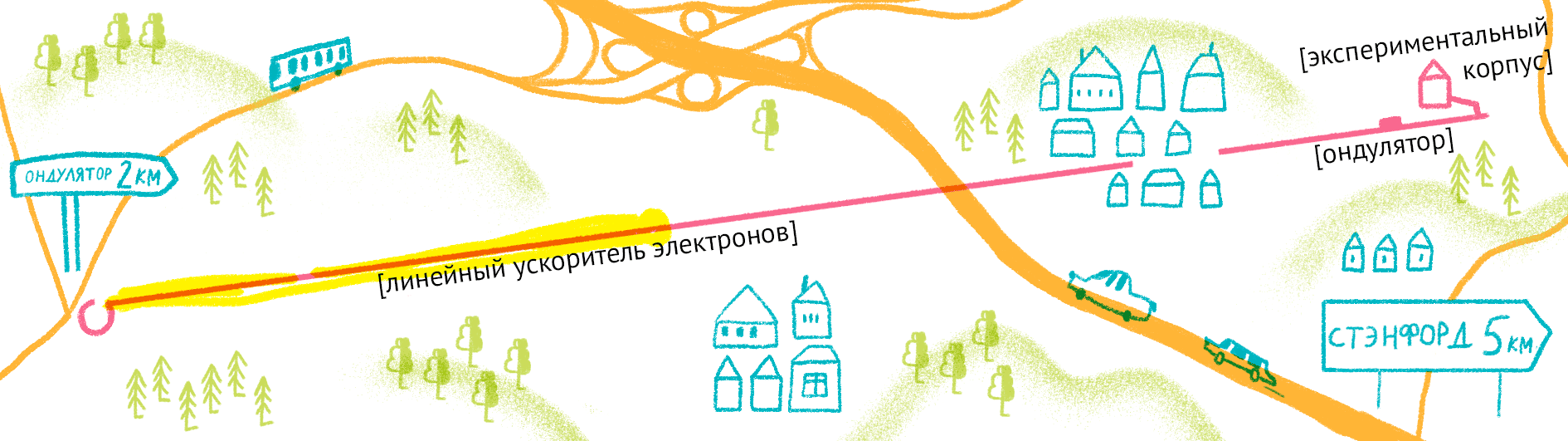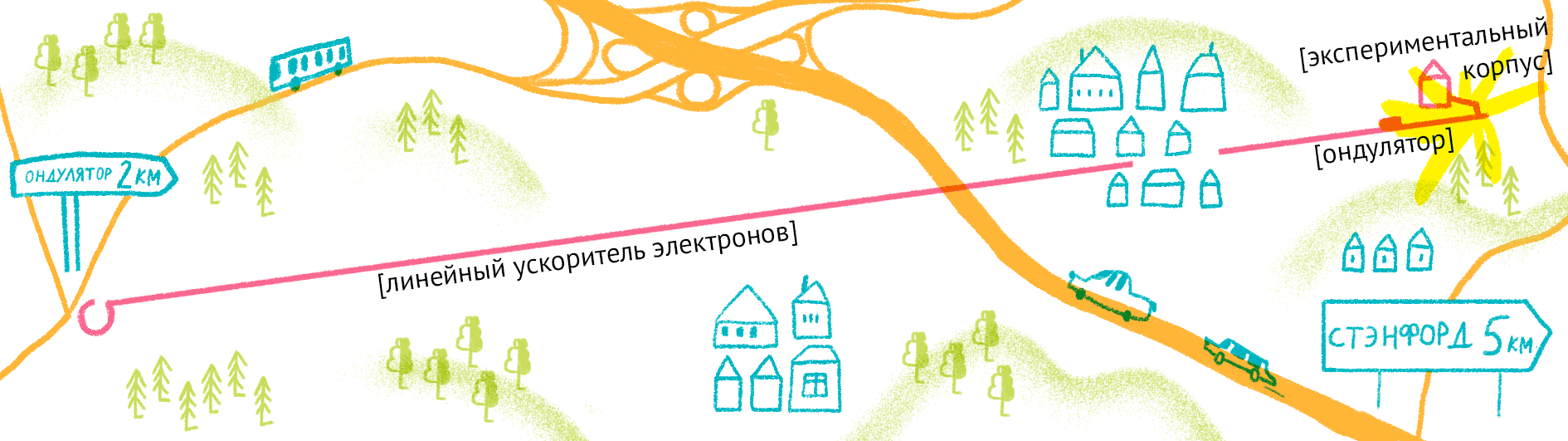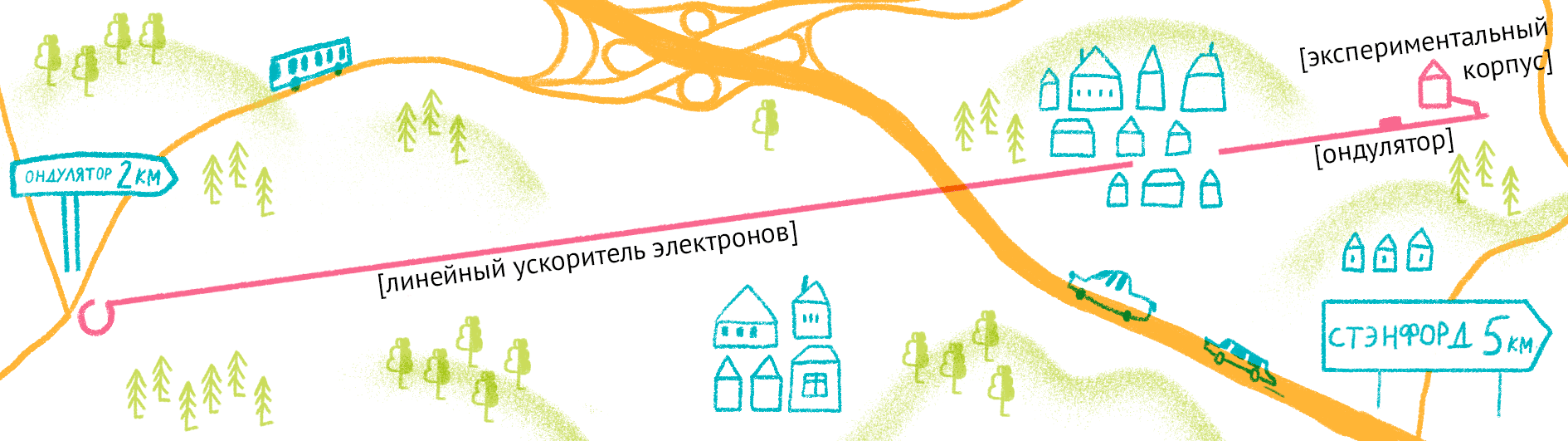
Рисунок 11. Карта лазера на свободных электронах LCLS (США).
Рисунок Ольги Пташник.

Криоэлектронная микроскопия
Электронный микроскоп — это аналог оптического микроскопа [23], использующий пучок быстрых электронов вместо света. Разрешающая способность обычных микроскопов ограничена длиной волны света и составляет около 200 нм; применение быстрых электронов позволяет повысить разрешение до 0,05 нм. Чтобы получить изображение, молекулы нужно зафиксировать, а вокруг образца создать высокий вакуум. Для этого объект замораживают при температуре жидкого азота, что и называется криоэлектронной микроскопией. Если заморозку провести достаточно быстро, то удастся получить любую макромолекулу в естественном окружении — в растворе, в отличие от кристаллографических методов или методов негативного контраста, когда высушенный образец обрабатывается антителами или другими агентами.
При исследовании биомолекул электронным микроскопом получается набор двумерных изображений, соответствующих различным ориентациям молекулы. Если ранее эти двумерные изображения и были финалом исследования, то сейчас появились вычислительные методы анализа двумерных проекций молекулы, которые позволяют получить трехмерные структуры белка или нуклеиновой кислоты (рис. 12) [24]. Благодаря развитию вычислительных методов и разработке новых чувствительных детекторов, криоэлектронная микроскопия набирает сумасшедшую популярность в последние 2–3 года, особенно в применении к мембранным белкам большого размера и целым вирусам (которые тяжело исследовать другими методами) [25], [26]. Наблюдается взрывной рост числа структур белков, полученных на электронном микроскопе: сейчас уже около 1% (1300) структур в базе PDB.

Рисунок 12. Изображения липид-белковых нанодисков размером 10–15 нм, полученные под криоэлектронным микроскопом.
Метод имеет только один существенный недостаток: им нельзя работать с «маленькими» объектами массой менее 60 000 атомных единиц — из-за низкой контрастности изображений и сложности анализа проекций. Интересные данные можно получить при помощи криоэлектронной томографии — трехмерное изображение органелл клеток, конечно, не в атомарном разрешении, но в достаточном для наблюдения структуры некоторых белков (например, АТФаз) в естественном окружении [27–30]. Стоимость криоэлектронного микроскопа находится в «разумных» пределах 5–10 млн долларов США. В России есть несколько криоэлектронных микроскопов, наиболее современный из которых расположен в Курчатовском институте в Москве и используется для получения структур белков самого высокого разрешения.

Ядерный магнитный резонанс
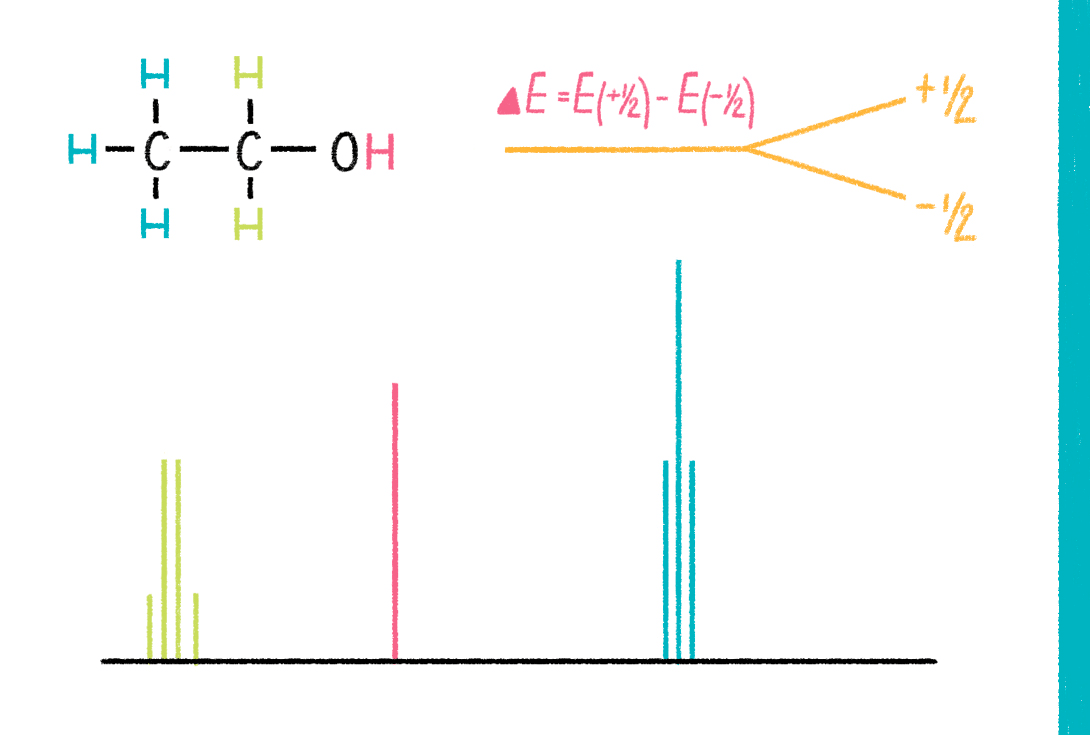
Рисунок 13. Химическая формула и спектр ЯМР этилового спирта. Схематически показана разница энергий двух спиновых состояний ядра водорода.
Рисунок Ольги Пташник.
Метод спектроскопии ядерного магнитного резонанса (ЯМР) имеет мало общего с ядерными реакторами, бомбами и прочими потенциальными причинами всемирного апокалипсиса. Метод изучает электромагнитные взаимодействия между ядрами (спинами ядер) в молекуле (рис. 13). Некоторые типы взаимодействий зависят от расстояния между ядрами или от угла вращения вокруг какой-нибудь химической связи, поэтому, если измерить большое количество таких взаимодействий, то можно восстановить пространственную структуру молекулы [31]. Чтобы получить возможность изучать спины ядер, молекулу нужно сначала поместить в сильное магнитное поле, которое создает магнит из сверхпроводников (поскольку спин, как крохотный магнит, взаимодействует со внешним магнитным полем). Эти магниты имеют гигантские размеры (высота до 5 метров) и стоят несколько миллионов долларов (рис. 14). Магнитное поле внутри ЯМР-спектрометра может достигать 23 тесла. Для сравнения, магнитное поле земли — 32 микротесла (в миллион раз меньше!), а 5 миллитесла (в 10 000 раз меньше) — напряженность магнитного поля, создаваемого магнитиком для холодильника.
Метод ЯМР-спектроскопии не только хорош, но и плох. К его несомненным преимуществам можно отнести возможность изучать молекулы в природном окружении — растворе, — а также потенциальную возможность исследовать не только структуру, но и подвижность биомолекул на различных участках. Среди минусов — длительность и дороговизна экспериментов и ограничения по размеру объектов исследования (масса молекулы не должна превышать 50–100 тысяч а.е.м.). Эксперимент ЯМР может длиться несколько недель из-за низкой чувствительности метода. При этом белки и нуклеиновые кислоты нужно обогащать редкими стабильными изотопами углерода (13С) и азота (15N), поскольку ядра обычных изотопов этих элементов не наблюдаются в ЯМР-спектрах в силу ряда причин, связанных с особенностью их спина.
Существует также модификация ЯМР-спектроскопии для изучения не жидких объектов, таких как белки в мембране или волокнах (фибриллах) — ЯМР твердого тела. Чтобы изучать белки вне раствора, нужно с бешеной скоростью (100 000 оборотов в секунду) вращать образец под «магическим» углом к направлению магнитного поля в 54,73 градуса [32].
При помощи ЯМР-спектроскопии определены около 10% пространственных структур в базе PDB. В России есть несколько сверхмощных чувствительных ЯМР-спектрометров, на которых ведут работы мирового уровня. Самая большая ЯМР-лаборатория не только в России, но и на всем пространстве к востоку от Праги и к западу от Сеула, расположена в Институте биоорганической химии РАН (Москва) [33].
Устройство ЯМР-спектрометра

ЯМР-спектрометр — замечательный пример торжества технологии над разумом. Как мы уже упоминали, для использования метода ЯМР-спектроскопии необходимо мощное магнитное поле, поэтому сердцем прибора является сверхпроводящий магнит — катушка из специального сплава, погруженная в жидкий гелий (−269 °C). Жидкий гелий нужен для достижения сверхпроводимости. Чтобы гелий не испарялся, вокруг него строят огромную цистерну с жидким азотом (−196 °C). Хоть это и электромагнит, он не потребляет электричества: сверхпроводящая катушка не имеет сопротивления. Однако, магнит нужно постоянно «подкармливать» жидким гелием и жидким азотом (рис. 15). Если не уследить, то произойдет «квенч»: катушка нагреется, гелий взрывоподобно испарится, а прибор сломается (см. видео). Также важно, чтобы поле в образце длиной 5 см было крайне однородным, поэтому в приборе присутствуют пара десятков небольших магнитов, нужных для тонкой настройки магнитного поля.

Рисунок 15. Автор текста рядом со своим карманным ЯМР-спектрометром.
Фото Алексея Паевского.
Видео. Запланированный «квенч» 21,14-теслового ЯМР-спектрометра.
Чтобы проводить измерения, нужен датчик — специальная катушка, которая как генерирует электромагнитное излучение, так и регистрирует «обратный» сигнал — осцилляцию магнитного момента образца. Чтобы повысить чувствительность в 2–4 раза, датчик охлаждают до температуры −200 °C, тем самым избавляясь от тепловых шумов. Для этого строят специальную машину — криоплатформу, которая охлаждает гелий до нужной температуры и прокачивает его рядом с детектором.

Методы на основе рассеяния излучения
Есть целая группа методов, которая опирается на явление рассеяния света, рентгеновских лучей или пучка нейтронов. Данные методы по интенсивности рассеяния излучения/частиц на различных углах позволяют определять размер и форму молекул в растворе (рис. 16). При помощи рассеяния невозможно определить структуру молекулы, однако его можно использовать как подспорье при использовании другого метода, например, ЯМР-спектроскопии. Приборы для измерения рассеяния света относительно дешевы и стоят «всего» около 100 000 долларов, в то время как другие методы требуют наличия под рукой ускорителя частиц, который может создать пучок нейтронов или мощный поток рентгеновского излучения.
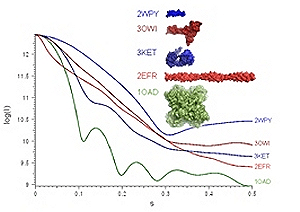
Рисунок 16. Кривые малоуглового рассеяния рентгеновских лучей для нескольких белков различной формы.
Сайт www.rigaku.com

Перенос флуоресценции
Другим методом, при помощи которого нельзя определить структуру, но можно получить кое-какие важные данные, является резонансный перенос энергии флуоресценции (FRET) [34]. Метод использует явление флуоресценции — способность некоторых веществ поглощать свет одной длины волны, испуская свет другой длины волны. Можно подобрать пару соединений, у одного из которых (донора) испускаемый при флуоресценции свет будет соответствовать характерной длине волны поглощения второго (акцептора). Облучать донор лазером нужной длины волны и измерять флуоресценцию акцептора. Эффект FRET зависит от расстояния между молекулами, поэтому, если ввести донор и акцептор флуоресценции в молекулы двух белков или разные домены (структурные единицы) одного белка, можно изучать взаимодействия между белками или взаимное расположение доменов в белке [35]. Регистрация осуществляется при помощи оптического микроскопа, поэтому FRET является дешевым, хотя и малоинформативным методом, использование которого сопряжено со сложностями в интерпретации данных.

Компьютерное моделирование
Рисунок 17. Иногда мощности самого большого компьютера недостаточно, чтобы смоделировать поведение одной-единственной молекулы белка.
Рисунок Ольги Пташник.
Наконец, нельзя не упомянуть о «методе мечты» структурных биологов — компьютерном моделировании (рис. 17). Идея метода состоит в том, чтобы, пользуясь современным знанием о строении и законах поведения молекул, моделировать поведение белка в компьютерной модели. Например, используя метод молекулярной динамики [36], можно в реальном времени отслеживать движения молекулы или процесс «сборки» белка (фолдинг) [37] за одним «но»: максимальное время, которое можно обсчитать, не превышает 1 мс [38], что чрезвычайно мало, но притом требует колоссальных вычислительных ресурсов (рис. 18) [39]. Можно исследовать поведение системы в течение более длительного времени, только достигается это за счет неприемлемой потери точности.

Рисунок 18. Суперкомпьютер ANTON, созданный специально для задач структурной биологии.
Компьютерное моделирование активно используется для анализа пространственных структур белков. При помощи докинга ищут потенциальные лекарства [40], [41], которые имеют высокую склонность к взаимодействию с белком-мишенью. В настоящий момент точность предсказаний все еще невелика, однако докинг позволяет существенно сузить круг потенциально активных веществ, которые необходимо проверить для разработки нового лекарства.
Применение в народном хозяйстве
Основным полем практического применения результатов структурной биологии является разработка лекарств или, как это сейчас модно говорить, драг-дизайн. Существует два способа разработать лекарство на основе структурных данных: можно начинать от лиганда или от белка-мишени. Если уже известно несколько лекарств, действующих на белок-мишень, и получены структуры комплексов белок-лекарство, можно создать модель «идеального лекарства» в соответствии со свойствами «кармана» связывания на поверхности молекулы белка, выделить необходимые черты потенциального лекарства и провести поиск среди всех известных природных и не очень соединений. Можно даже построить зависимости между свойствами структуры лекарства и его активностью. Например, если у молекулы есть сверху бантик, то ее активность выше, чем у молекулы без бантика. И чем больше бантик заряжен, тем лучше работает лекарство. Значит, из всех известных молекул нужно найти соединение с самым большим заряженным бантиком.
Другой способ — используя структуру мишени, на компьютере провести поиск соединений, которые потенциально способны с ней взаимодействовать в нужном месте. При этом обычно используют библиотеку фрагментов — небольших кусочков веществ. Если найти несколько хороших фрагментов, которые взаимодействуют с мишенью в разных местах, но близко друг от друга, можно построить из фрагментов лекарство, «сшив» их между собой. Существует много примеров успешной разработки лекарств при помощи структурной биологии. Первый успешный случай датируется 1995 годом: тогда был одобрен к применению дорзоламид — лекарство от глаукомы.
Общая тенденция в биологических исследованиях все больше склоняется не только к качественному, но и количественному описанию природы. Структурная биология — яркий тому пример. И есть все основания полагать, что она будет и дальше приносить пользу не только фундаментальной науке, но и медицине, и биотехнологиям.
Календарь
На основе статей спецпроекта мы решили сделать календарь «12 методов биологии» на 2019 год. Эта статья представляет март.

Литература
- Биолюминесценция: возрождение;
- Торжество компьютерных методов: предсказание строения белков;
- Heping Zheng, Katarzyna B Handing, Matthew D Zimmerman, Ivan G Shabalin, Steven C Almo, Wladek Minor. (2015). X-ray crystallography over the past decade for novel drug discovery – where are we heading next?. Expert Opinion on Drug Discovery. 10, 975-989;
- Alan R. Fersht. (2008). From the first protein structures to our current knowledge of protein folding: delights and scepticisms. Nat Rev Mol Cell Biol. 9, 650-654;
- На заре молекулярной графики;
- J. C. KENDREW, G. BODO, H. M. DINTZIS, R. G. PARRISH, H. WYCKOFF, D. C. PHILLIPS. (1958). A Three-Dimensional Model of the Myoglobin Molecule Obtained by X-Ray Analysis. Nature. 181, 662-666;
- R. Henderson, P. N. T. Unwin. (1975). Three-dimensional model of purple membrane obtained by electron microscopy. Nature. 257, 28-32;
- Jan Pieter Abrahams, Andrew G. W. Leslie, René Lutter, John E. Walker. (1994). Structure at 2.8 Â resolution of F1-ATPase from bovine heart mitochondria. Nature. 370, 621-628;
- Wimberly B.T., Brodersen D.E., Clemons W.M. Jr., Morgan-Warren R.J., Carter A.P., Vonrhein C. et al. (2000). Structure of the 30S ribosomal subunit. Nature. 407, 327–339;
- Зрительный родопсин — рецептор, реагирующий на свет;
- Новый рубеж: получена пространственная структура β2-адренорецептора;
- Структуры рецепторов GPCR «в копилку»;
- Нобелевская премия по химии (2012): за рецепторы наших первого, третьего и четвертого чувств;
- Иммунологическая Нобелевская премия (2011);
- Толл-подобные рецепторы: от революционной идеи Чарльза Джейнуэя до Нобелевской премии 2011 года;
- Razi A., Britton R.A., Ortega J. (2017). The impact of recent improvements in cryo-electron microscopy technology on the understanding of bacterial ribosome assembly. Nucleic Acids Res. 45, 1027–1040;
- Rafael Fernandez-Leiro, Sjors H. W. Scheres. (2016). Unravelling biological macromolecules with cryo-electron microscopy. Nature. 537, 339-346;
- Ловля бабочек, или Чем структурная геномика поможет биологии;
- Структурная геномика меняет курс;
- Лаборатория перспективных исследований мембранных белков: от гена к ангстрему;
- Фемтосекундные рентгеновские лазеры — кристаллография будущего;
- Neutze R., Wouts R., van der Spoel D., Weckert E., Hajdu J. (2000). Potential for biomolecular imaging with femtosecond X-ray pulses. Nature. 406, 752–757;
- Микроскопия в домашних условиях;
- L. Zhang, J. Song, Y. Newhouse, S. Zhang, K. H. Weisgraber, G. Ren. (2010). An optimized negative-staining protocol of electron microscopy for apoE4*POPC lipoprotein. The Journal of Lipid Research. 51, 1228-1236;
- D. Sirohi, Z. Chen, L. Sun, T. Klose, T. C. Pierson, et. al.. (2016). The 3.8 A resolution cryo-EM structure of Zika virus. Science. 352, 467-470;
- Karl Bertram, Dmitry E. Agafonov, Wen-Ti Liu, Olexandr Dybkov, Cindy L. Will, et. al.. (2017). Cryo-EM structure of a human spliceosome activated for step 2 of splicing. Nature. 542, 318-323;
- Получена первая полная реконструкция эукариотической клетки в трехмерном пространстве;
- Молекулярная модель синаптической везикулы;
- В полном объеме: синаптические везикулы в трехмерной модели синапса;
- L. A. Baker, I. N. Watt, M. J. Runswick, J. E. Walker, J. L. Rubinstein. (2012). Arrangement of subunits in intact mammalian mitochondrial ATP synthase determined by cryo-EM. Proceedings of the National Academy of Sciences. 109, 11675-11680;
- Matthias Huber, Anja Böckmann, Sebastian Hiller, Beat H. Meier. (2012). 4D solid-state NMR for protein structure determination. Phys. Chem. Chem. Phys.. 14, 5239;
- Xavier L. Warnet, Alexandre A. Arnold, Isabelle Marcotte, Dror E. Warschawski. (2015). In-Cell Solid-State NMR: An Emerging Technique for the Study of Biological Membranes. Biophysical Journal. 109, 2461-2466;
- Сверхпроводящие магниты и рецепторы биомембран: Лаборатория биомолекулярной ЯМР-спектроскопии ИБХ РАН;
- Рулетка для спектроскописта;
- Marko Sustarsic, Achillefs N Kapanidis. (2015). Taking the ruler to the jungle: single-molecule FRET for understanding biomolecular structure and dynamics in live cells. Current Opinion in Structural Biology. 34, 52-59;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- Проблема фолдинга белка;
- Миллисекундный барьер взят!;
- Ledford H. (2010). Supercomputer sets protein-folding record. Nature News;
- Драг-дизайн: как в современном мире создаются новые лекарства;
- Виртуальные тропы реальных лекарств.