Аптамеры: назад в будущее. Старая концепция в новой технологической реальности
11 декабря 2025
Аптамеры: назад в будущее. Старая концепция в новой технологической реальности
- 231
- 0
- 6
Компьютерное проектирование аптамера под конкретный участок белка.
Рисунок в полном размере.
иллюстрация Никиты Парфенова
-
Авторы
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: В 1990-х казалось, что аптамеры вот-вот вытеснят антитела. Спустя десятилетия — всего несколько препаратов, а в обзорах все чаще звучит: «высокий потенциал, но мало реализован». В этой статье мы разбираемся, что дело может быть не в самих аптамерах, а в способе их поиска — методе SELEX, который на практике напоминает лотерею. На фоне успехов AlphaFold мы объясняем, почему прямой перенос этого подхода на аптамеры не работает, и как вместо случайного отбора перейти к рациональному дизайну. Наконец, мы рассказываем, как платформа Xelari заменяет месяцы лабораторного перебора полным компьютерным проектированием аптамеров и почему у этих почти забытых молекул снова появился шанс.
Конкурс «Био/Мол/Текст»-2025/2026
Эта работа опубликована в спецноминации «Искусственный интеллект в биологии» конкурса «Био/Мол/Текст»-2025/2026.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Партнер номинации — компания Центральный университет в лице совместной с институтом AIRI Лаборатории ИИ, анализа данных и моделирования.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Как нуклеиновые кислоты получили новую роль
В начале 1990-х в лабораториях США и Европы крутили в пробирках растворы РНК и ДНК, а в головах у людей уже складывалась картинка будущего: вместо капризных, дорогих белковых антител — небольшие, химически синтезируемые, послушные молекулы. Их назвали аптамерами (от лат. aptus — «подходить»). Аптамеры — это одноцепочечные ДНК или РНК (обычно 20–100 нуклеотидов). Одни и те же буквы генетического алфавита, но новая функция: не «носители информации», а «молекулярные ключи» ко всем мыслимым мишеням. Аптамеры обещали стать мечтой инженера. Во-первых, их можно было получить химическим синтезом: не требовались клеточные культуры или животные — это упрощало производство. Во-вторых, в аптамер можно было относительно легко вносить различные химические модификации — например, добавлять фторированные группы или метилирование, чтобы повысить стабильность или задать нужные свойства. В-третьих, аптамеры меньше по размеру и могут быть более стабильными: не агрегируют, легче хранятся, выдерживают температуру и остаются активными. Наконец, благодаря огромному пространству возможных последовательностей нуклеотидов, теоретически можно подобрать аптамер к почти любой мишени — даже там, где трудно получить антитело [1].
Впрочем, антитела не спешат уходить со сцены: первыми на практике показавшие, что такое таргетная терапия [2], сегодня они — рабочие лошадки онкологии и иммунологии, фактически став вторым поколением лекарств [3]: «Краткая история открытия и применения антител» [4], «Антитело: лучший способ распознать чужого» [5] и «Терапевтические моноклональные антитела» [6]. — Ред.
Что такое аптамеры, их преимущества над антителами и этапы отбора — уже визуально обсуждалось в комиксе «Аптамеры: графический гайд» [7].
Прорыв выглядел убедительно. В 1990 году сразу две группы — Крэйг Терк и Ларри Голд [8], с одной стороны; и Эндрю Эллингтон с Джеком Шостаком [9], с другой — показали, что если взять библиотеку из примерно 1015 случайных олигонуклеотидов и прогнать ее через несколько циклов отбора и амплификации, то из хаоса случайных последовательностей найдутся молекулы, которые цепляются к заданной мишени так же избирательно, как антитела. Метод назвали SELEX — Systematic Evolution of Ligands by Exponential Enrichment.
Фантазия сделала остальное. Если можно получить аптамер к белку А, значит, можно сделать то же самое к ферменту B, токсину C, вирусу D... Уже в начале 2000-х в воздухе витал вопрос: «Аптамеры — новые антитела?»
Крутая технология, плохая судьба
К 2010-м годам за спиной аптамеров было уже множество красивых статей и первая одобренная FDA терапия — пегаптаниб (Macugen) против возрастной макулодистрофии [10]; а позже второй аптамер — авацинкаптад пегол (Izervay). Но список одобренных препаратов на этом фактически и заканчивался, тогда как у антител — десятки позиций, миллиарды долларов оборота и целые классы препаратов (см.: «Терапевтические моноклональные антитела» [6]).
В 2014 году один из «отцов» аптамеров — Эндрю Эллингтон — опубликовал блог-пост с говорящим названием On aptamers. В тексте он почти с сухим юмором подвел промежуточные итоги: аптамеры — классная технология с сильным исследовательским трек-рекордом, но с очень слабой коммерческой судьбой; а в прямом сравнении антитела в его лаборатории «работают лучше почти во всех случаях» и часто связывают мишень в 10–100 раз сильнее.
Для внешнего наблюдателя это был просто честный технический разбор. Для части сообщества, которое двадцать лет строило вокруг аптамеров компании и технологии, слова человека, стоявшего у истоков, прозвучали как почти публичное отречение. Если даже Эллингтон говорит, что антитела гораздо практичнее, значит, что-то пошло фундаментально не так.
Часть инвесторов охладела. Несколько компаний были проданы, переформатированы или растворились в крупных фармацевтических структурах. В обзорах все чаще появлялась фраза примерно такого содержания: «аптамеры сохраняют высокий потенциал, но пока мало реализованы на рынке».
И все-таки история на этом не закончилась.
SELEX как лотерея
Чтобы понять, почему потенциал аптамеров остался нереализованным, придется чуть отмотать назад, к самому процессу SELEX (рис. 1) [8].
Классический путь получения аптамеров (SELEX) — это долго и непросто. Исходно исследователь синтезирует совершенно случайную библиотеку олигонуклеотидов из колоссального числа различных последовательностей (обычно порядка 1015, то есть квадриллион разных молекул!). Эту смесь пропускают через колонку с целевым белком и отбирают те несколько молекул, что слегка связались с белком, затем их амплифицируют (через ПЦР ). ПЦР копирует цепочки не идеально: при амплификации возникают редкие ошибки-мутации. В SELEX эту естественную ошибочность ПЦР сознательно усиливают и используют как источник новых вариантов, для чего меняют условия ПЦР так, чтобы полимераза чаще ошибалась (например, добавляют соли марганца вместе с солями магния). После амплификации смесь снова пускают через колонку. И так цикл за циклом, пока не останутся только самые цепкие последовательности. Процесс отдаленно напоминает естественный отбор, только проводится вручную. В итоге через несколько недель и десятки циклов появляется «победитель» — аптамер с наилучшим связыванием. Звучит здорово, но на практике SELEX ненадежен. В реальности в самой технологии заложено несколько системных проблем.
О принципах ПЦР можно быстро вспомнить в статье «12 методов в картинках: полимеразная цепная реакция» [11].
Формально мы работаем с огромной библиотекой — 1015 последовательностей, — но по сравнению с полным пространством возможных цепочек это микроскопический островок. Для цепочки всего из 40 нуклеотидов число вариантов уже 440 (≈1024), то есть существенно больше 1015. SELEX, по сути, берет один случайный островок в этом океане последовательностей и исследует его локально, никогда не зная, лучший он или просто первый попавшийся. При этом процесс легко застревает в локальных максимумах аффинности, так никогда и не дойдя до глобального. Последовательность, которая чуть крепче соседей связалась в первых раундах, получает экспоненциальное преимущество в ПЦР и может вытеснить более перспективные, но пока редкие варианты. В итоге «победитель» часто оказывается таковым не потому, что он глобально оптимален, а потому что ему повезло немного раньше [12], [13].
Вторая системная проблема — жесткая привязка к лабораторным условиям. Определенный буфер с заданными ионами, фиксированный pH, мишень, посаженная на поверхность или колонку, комнатная температура или стандартные 37 oC. В этих условиях и отбирается аптамер, идеально подходящий именно под такую конфигурацию. Но реальная задача редко выглядит так же — могут быть другие концентрации ионов, белок может быть по-другому свернут, присутствуют конкурирующие молекулы. Итог предсказуем — аптамер, который показал хорошие результаты в отборе, при переносе в реальные условия часто проигрывает или вообще перестает связываться [12].
Отдельный пласт проблем связан с амплификацией и артефактами. Последовательности с меньшей длиной, определенным GC-составом, более послушной вторичной структурой (меньше сложных узлов и G-квадруплексов), с более доступными сайтами для праймеров амплифицируются лучше. В итоге отбор идет не только по качеству связывания, но и по амплифицируемости. Редкие, но потенциально очень сильные аптамеры могут просто исчезнуть из пула, потому что их сложная структура мешает полимеразе нормально работать. Также добавьте к этому потери на стадиях промывки, неспецифичную адсорбцию на пластике и т.д. [14].
Каждая новая мишень в классическом SELEX — это отдельный проект на недели или месяцы со своей кухней условий, буферов, температур в каждой лаборатории. В отличие от антител, где сформировались целые платформы — стандартизированные модели, рекомбинантные библиотеки, отлаженная аналитика, — аптамеры так и остались миром, где каждый снова и снова изобретает велосипед.
Итоговая комбинация всех этих факторов приводит к неприятному результату. Можно потратить месяцы на SELEX и в конце получить аптамер, который прекрасно работает в вашем буфере, но не воспроизводится в другой лаборатории. Или, что еще хуже, вообще не получить пригодного кандидата и так и не понять до конца, то ли в библиотеке не было подходящих последовательностей, то ли вы потеряли их по дороге [15]. Отсюда десятки модификаций SELEX (cell-SELEX [16], counter-SELEX [17], in vivo-SELEX [18] и т.д.). Каждая новая вариация улучшала какую-то часть процесса, но не отменяла главный принцип и его ограничения. Это дорогостоящий и трудоемкий путь. Неудивительно, что многие перспективные мишени до сих пор не имеют аптамеров просто потому, что традиционный отбор слишком сложен и случаен.
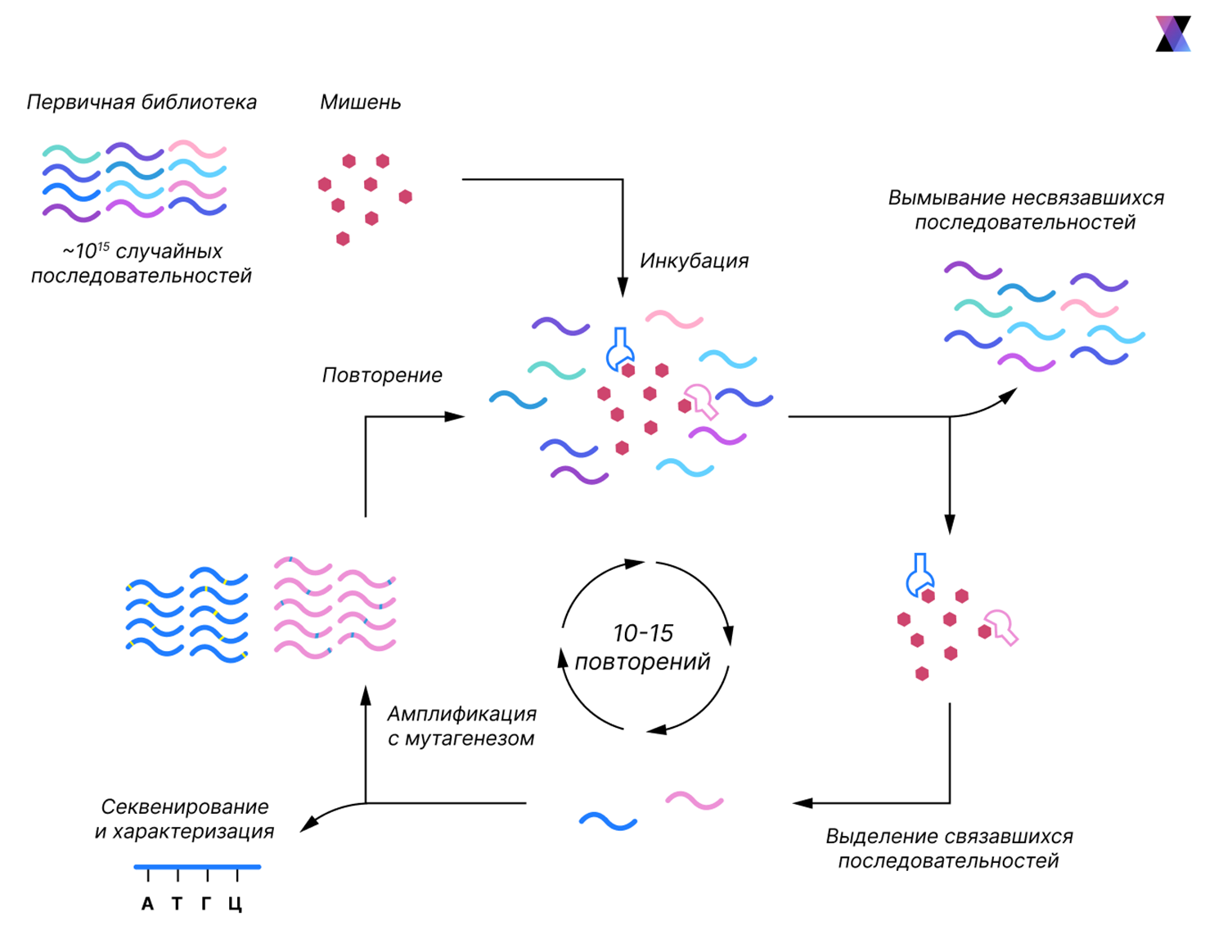
Рисунок 1. Цикл SELEX. На первом этапе синтетическая библиотека одноцепочечных ДНК/РНК с высоким разнообразием случайных участков инкубируется с целевым белком. Связавшиеся с мишенью последовательности отделяются от несвязанных. Далее связавшиеся аптамеры элюируются и подвергаются амплификации с мутагенезом (ПЦР для ДНК или обратная транскрипция с последующей ПЦР для РНК), формируя обогащенную библиотеку следующего цикла. Цикл повторяется несколько раз, отобранные последовательности затем секвенируют.
иллюстрация Никиты Парфенова
И вот здесь возникает неприятный вопрос, которого долго избегали: а что, если мы все это время судили аптамеры по работе одного, далеко не идеального поискового алгоритма?
В таком случае история аптамеров — это уже не рассказ о несостоявшейся технологии, а история о классе молекул, которому банально не дали нормального шанса, ведь их потенциал оценивали по инструменту, который изначально был самым слабым звеном.
AlphaFold показал, как надо работать с биомолекулами
Проблема предсказания структуры белка по его аминокислотной последовательности считалась одним из величайших вызовов биологии последнего полувека [19], [20]. Расшифровка структуры одного белка могла растянуться на годы работы целой команды. Экспериментальные методы вроде рентгеновской кристаллографии, крио-ЭМ или ЯМР-спектроскопии потребовали десятилетий, чтобы определить пространственные укладки примерно 170 тысяч белков (столько их было в PDB к 2020 году). Это много, если смотреть глазами отдельного лабораторного ассистента. И удивительно мало, если вспомнить, что в живой природе миллионы разных белков и их вариантов.
Подробнее о методах структурной биологии можно почитать в обзоре «12 методов в картинках: структурная биология» [21], а детальнее ознакомиться с принципом работы AlphaFold в статьях «AlphaFold: нейросеть для предсказания структуры белков от британских ученых» [22], «Белковые галлюцинации: как справляется AlphaFold?» [23] и, конечно, в материале «Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024)» [24].
Перелом в развитии методов предсказания структуры белка произошел с появлением AlphaFold2 [25]. В рамках соревнования CASP14 эта система по аминокислотной последовательности предсказала трехмерную структуру белка с точностью, сопоставимой с экспериментальными данными. ИИ фактически научился использовать накопленные в PDB данные о сотнях тысяч белков и миллионы последовательностей из генетических данных, угадывая, как цепочка аминокислот сворачивается в характерную трехмерную структуру . Уже к 2022 году AlphaFold2 предоставила научному сообществу структуры практически всех известных науке белков — это свыше 200 миллионов предсказанных форм!
Принципы машинного обучения, произведшие революцию в биоинформатике, «Биомолекула» разбирает в спецпроекте «Искусственный интеллект в биологии» и рекомендует всем читателям ознакомиться с этими важнейшими публикациями: «История развития искусственного интеллекта и его пришествия в биологию» [26] и «Как языковые модели покорили мир белков» [27].
AlphaFold сделал больше, чем просто предсказал структуры массы неизвестных белков. Он показал принцип работы с биомолекулами в эпоху больших данных и доказал эффективность такого подхода. А если мы научились так обращаться с белками, можно ли сделать то же самое для аптамеров?
Почему с аптамерами просто скопировать подход AlphaFold не получится
На первый взгляд напрашивается решение: если AlphaFold научился предсказывать структуру белка, то, может, стоит показать ему структуры комплексов аптамер–белок, чтобы он научился предсказывать и их? Увы, все не так просто: главная проблема — в данных.
AlphaFold опирался на огромный пласт экспериментальных данных: 170 тысяч известных белковых структур в PDB (и, главное, миллионы последовательностей белков, известных из геномных проектов). А сколько же накоплено структурных данных по комплексам аптамеров с белками? Считанные десятки. Первые структуры комплексов аптамер—белок появились в 1990-х, и к 2010-м их число измерялось десятками. Позже, к 2018 г., эта цифра выросла примерно до 45, а в 2025 приближается к сотне с небольшим [28]. Даже если взять все структуры свободных аптамеров (без белка), аптамерных комплексов и нуклеиновых кислот вместе, это ничтожная выборка в сравнении с белковой базой данных. В PDB суммарно уже более 200 000 структур, из них десятки тысяч — белковые комплексы с малыми молекулами или белок—белковые взаимодействия. А вот аптамерных комплексов буквально <1% от этого объема. Попросту не на чем обучать нейросеть (рис. 2).
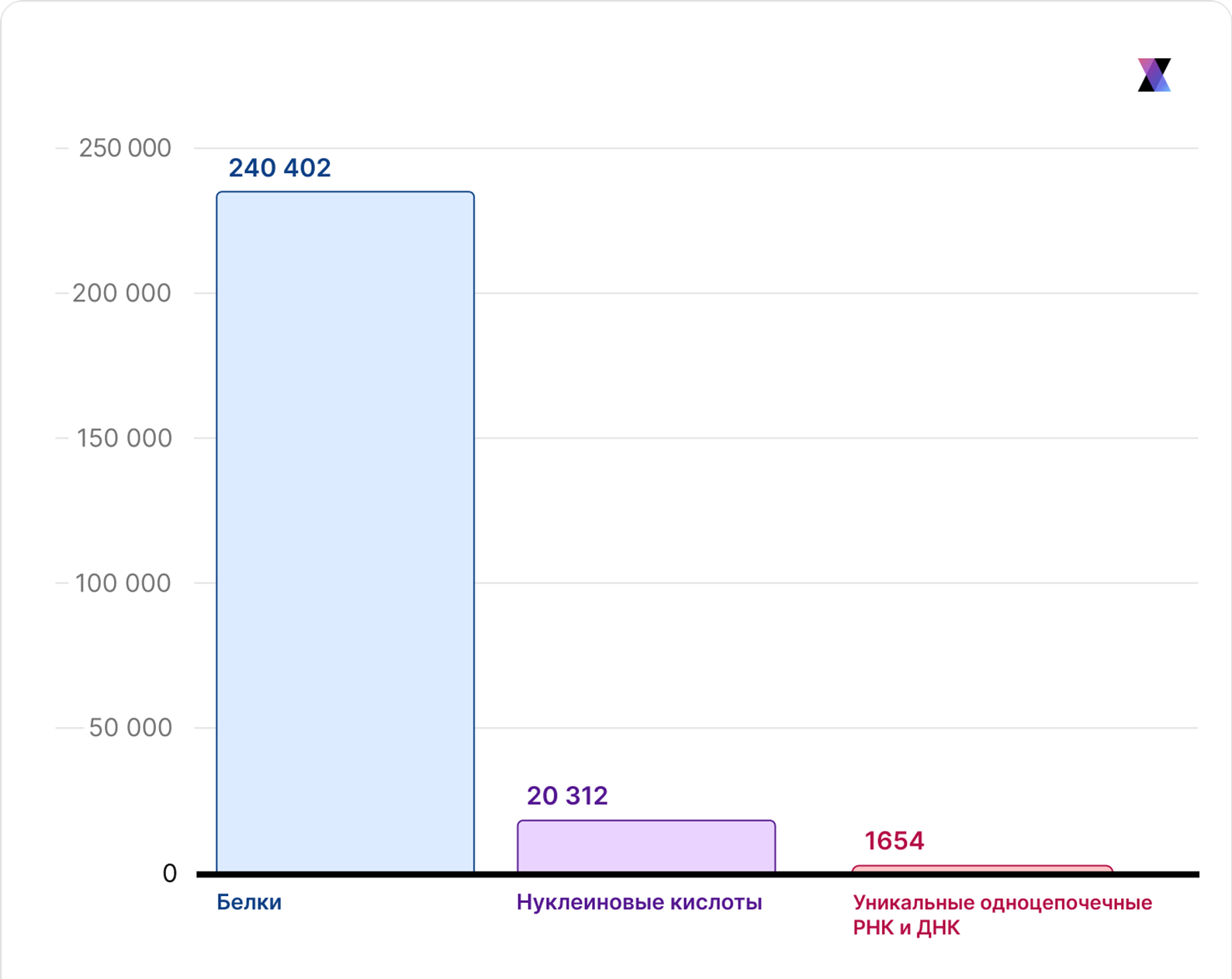
Рисунок 2. Сводная таблица доступных экспериментальных структур в базе данных Protein Data Bank — на 2025 год.
иллюстрация Никиты Парфенова
Шаг 1. Надежная вторичная структура
Ладно, структур комплексов мало, но может, хотя бы есть методы, которые предсказывают структуру самого аптамера? Ведь в задаче проектирования комплекса сначала нужно понять, как аптамерная последовательность свернется, даже без белка. Вторичная структура задает основной каркас этой формы, она образуется по принципу комплементарности (A—T, G—C или A—U в РНК). Могут образовываться различные мотивы (рис. 3): шпилька, внутренняя петля, выпетливание, а иногда и псевдоузлы — варианты укладки, при которых одна и та же одноцепочечная нуклеиновая кислота образует перекрещивающиеся участки и дополнительные контакты между удаленными фрагментами последовательности.
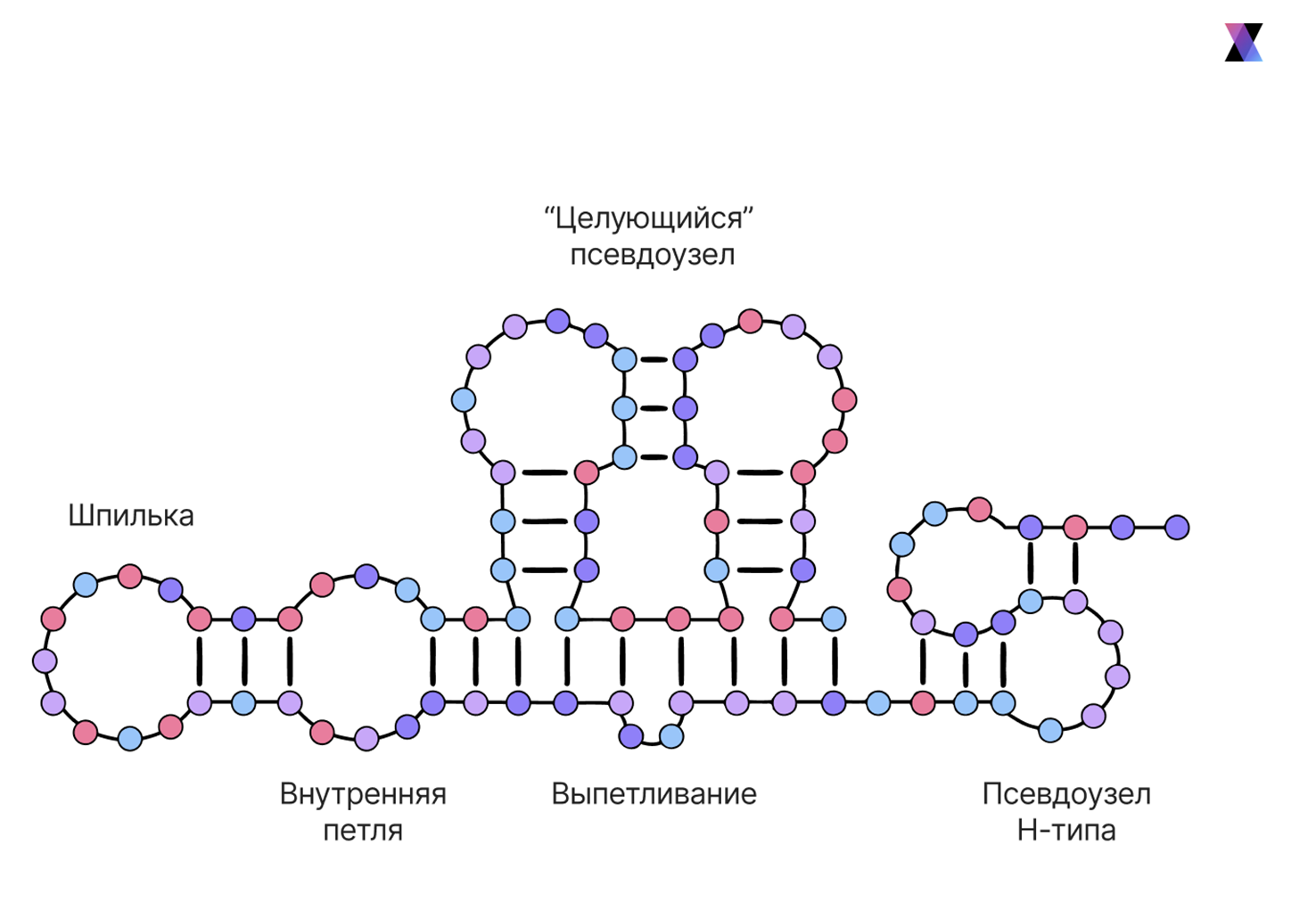
Рисунок 3. Основные типы мотивов вторичной структуры, формируемых одноцепочечными ДНК и РНК. Показана классическая шпилька (hairpin), отдельно выделены внутренние петли и выпетливания, возникающие при неполном комплементарном спаривании, а также псевдоузлы. Функциональные нуклеиновые кислоты часто используют комбинации нескольких типов вторичной структуры.
иллюстрация Никиты Парфенова
Для РНК- и ДНК-аптамеров существуют алгоритмы предсказания вторичной структуры. Классические программы типа RNAfold [29], Mfold [30] и им подобные строят такую структуру, минимизируя свободную энергию. Однако точность этих методов оставляет желать лучшего: по независимым тестам она достигает лишь 50%, и особенно плохо выходит с псевдоузлами [31]. Получается замкнутый круг: чтобы предсказывать третичную структуру и связывание аптамера, неплохо бы уметь находить его вторичную структуру, а стандартные методы дают слишком шумные, ненадежные результаты. Можно было бы обучить нейросеть предсказывать вторичную структуру по последовательности (и такие попытки есть), но для обучения нужны верные структуры, а их опять-таки мало.
Работая над решением этой проблемы в Xelari, мы сначала разработали алгоритм AliNA (ALIgned Nucleic Acids), который демонстрирует высокую точность на негомологичных к обучающей выборке РНК [32]. Модель использует гибридную архитектуру на базе сети U-Net с добавлением механизмов внимания. Изначально в AliNA мы пробовали искусственно расширять обучающую выборку. Для этого брали большие массивы РНК-последовательностей из открытых баз, предсказывали для них вторичную структуру тем же RNAfold и добавляли в обучение как новые примеры. На поздних этапах мы от этой стратегии аугментации отказались и сейчас фокусируемся только на обработке данных структурного секвенирования и коэволюции (рис. 4).
Представьте себе молекулу РНК-аптамера в растворе. Одни ее участки свернулись и образовали структуру, там основания попарно связаны. Другие остаются гибкими, не образуя пар. Структурное секвенирование как раз это и оценивает.
Один из типичных методов — SHAPE-seq [33]: в нем специальный реагент химически метит неспаренные нуклеотиды. Затем всю популяцию таких РНК секвенируют и для каждого нуклеотида считают, как часто он был помечен. Получается профиль реактивности, который хорошо коррелирует с тем, насколько велика вероятность, что нуклеотид одинок и ни с кем не спарен. Такие профили дают подсказку при предсказании структуры — если для некоторого участка реактивность высокая, там вряд ли образуется пара, а если низкая — то наоборот. Профили реактивности по разным экспериментам и условиям кластеризуются, из наиболее крупного кластера строится усредненный профиль для каждой молекулы. Затем он подается на вход набору алгоритмов (RNAshapes [34], SPOT-RNA [35], RNAfold [29] и другие), которые генерируют десятки кандидатных вторичных структур с учетом псевдоузлов и энергетической стабильности комплекса. Хотя метод SHAPE-seq и применим только к РНК, в модели заложен набор ограничений и приемов, которые вынуждают ее учить физические закономерности, благодаря чему этот подход применим также и к ДНК.
Параллельно есть еще один тип информации — коэволюционные данные. Его успешно использовали для белков. Если у вас есть множество родственных последовательностей, вы выравниваете их и ищете согласованные мутации. Когда в рамках одного вида меняется одна позиция, а другая подстраивается до комплементарности, чтобы сохранялось взаимодействие — это намек, что эти позиции пространственно близки и, скорее всего, образуют пару [36–38].
Чтобы не доверять слепо даже хорошо подошедшему кандидату, мы объединяем оба типа данных: профили реактивности и коэволюционные данные. Выбираем тот вариант структуры, который не противоречит сразу обоим источникам. Дальнейшая обработка включает предсказание третичной структуры, симуляция поведения в растворе, кластерный анализ конформационного ландшафта и считывание полученной вторичной структуры, что приводит к избавлению от лишних предсказанных «комплементарных» связей.
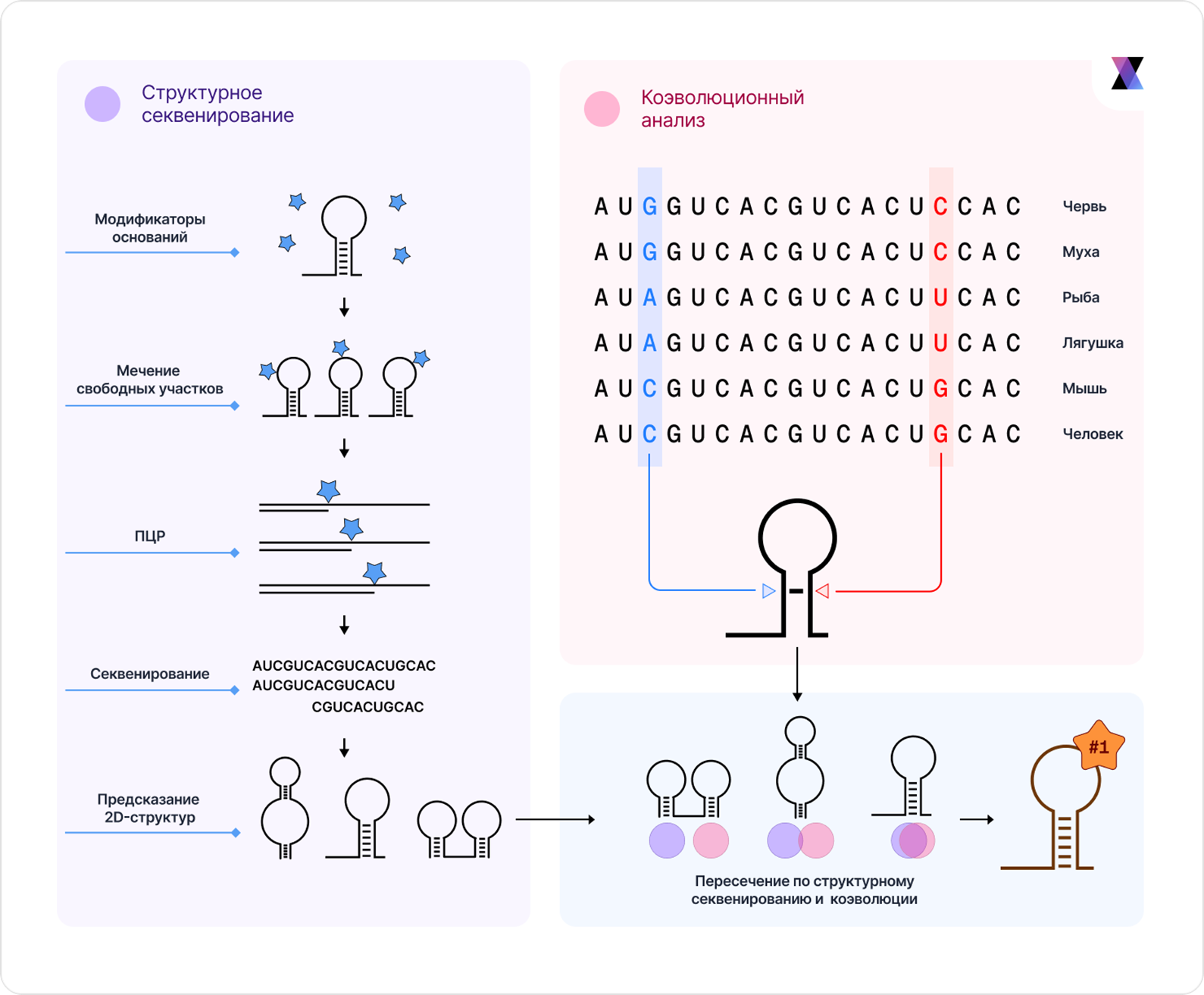
Рисунок 4. Объединение двух независимых источников информации о структуре нуклеиновой кислоты — данных структурного секвенирования и анализа коэволюционных данных в множественном выравнивании родственных последовательностей. Слева — профили реактивности по данным SHAPE-seq. Справа — множественное выравнивание и расчет парных коэволюционных контактов, отражающих совместную изменчивость позиций, предположительно находящихся в пространственной близости. На основе этих данных генерируется набор кандидатных вторичных структур различными алгоритмами, включая методы, учитывающие псевдоузлы и энергетическую стабильность. Далее из этого набора выбирается та структура, которая лучше всего согласуется с обоими источниками данных.
иллюстрация Никиты Парфенова
Этот подход дает AliNA возможность учиться на примерах, которые по качеству ближе к биофизическим экспериментам, чем к сырым предсказаниям старых алгоритмов, и за счет этого достигается точность до 90% для РНК и 81% для ДНК-структур на негомологичных обучающей выборке структурных данных из PDB (метрикой точности является F-значение — среднее гармоническое между долей правильно определенных комплементарных пар и правильно определенных неспаренных оснований).
Однако даже самый точный метод предсказания вторичной структуры сам по себе задачу не решает. Аптамер связывается в конкретной трехмерной структуре и на конкретной поверхности белка. Поэтому после получения разумной гипотезы о вторичной структуре следующий шаг — явное моделирование трехмерной укладки аптамера и его комплекса с белком. Это уже задача структурного моделирования: построить атомарную 3D-модель, перебрать возможные варианты укладки и оценить энергию связывания.
Есть еще принципиальная разница между белками и нуклеиновыми кислотами. Белки — относительно жесткие молекулы, их нативная структура термодинамически выгодна и существенно устойчивее по сравнению с альтернативными укладками. Поэтому задача предсказать структуру по сути совпадает с задачей найти наиболее энергетически выгодную форму укладки. А вот у аптамеров немного иначе: разные укладки цепи РНК или ДНК могут иметь близкую энергию и быть разделены малыми барьерами, и молекула может переходить между несколькими состояниями [39]. Небольшие изменения — и аптамер либо вообще не сложится, либо сворачивается не в ту форму, либо перестанет связываться с белком. То есть для функции аптамера ключевой фактор — энергетический ландшафт: какие состояния для цепи возможны, насколько выгодны они и вероятности переходов между ними. Для надежного дизайна нужно предсказывать не только форму, но и то, насколько легко она образуется и удерживается. Проще говоря, основной вопрос — не как выглядит комплекс аптамера с белком, а насколько устойчиво этот комплекс держится и насколько вероятно, что аптамер примет нужную форму. Таким образом, подход, учитывающий только геометрию, оказывается недостаточным — необходимо явное рассмотрение энергетических и термодинамических характеристик системы.
Шаг 2. От структуры к энергиям — новая база обучающих данных
Если проблемы упираются в недостаток структурных данных, то, возможно, надо поменять постановку задачи. Вместо того, чтобы пытаться переложить подход, успешный для белков, на совершенно другие условия, разумно разработать новую стратегию, учитывающую специфику аптамеров. Эта стратегия, судя по всему, должна в большей степени опираться на энергетические параметры: свободную энергию сворачивания, энергию связывания, константу диссоциации и т.д.
Прежде всего, потребуется собрать данные нового типа для обучения ИИ. Помимо одной лишь геометрии (координат атомов), нужно создать базу энергетических измерений. Однако откуда же их взять?
Во-первых, из экспериментов: в литературе разбросано множество данных о термодинамике нуклеиновых кислот. Например, измерены значения ΔG (свободная энергия Гиббса) для раскручивания различных РНК- и ДНК-мотивов (стеблей, петель, шпилек) при разных условиях. Есть и экспериментальные значения энергий связывания (констант диссоциации, Kd) для некоторых комплексов аптамеров с белками. Все эти разрозненные данные нужно собрать и привести к единому стандарту (нормализовать по условиям: температура, ионная сила и пр.).
Но одних экспериментов мало, их ограниченное количество. Поэтому, во-вторых, задействуем вычислительную химию. Современные программы молекулярной динамики и квантовохимические расчеты позволяют оценивать энергию взаимодействий молекул. Конечно, точный квантовый расчет для большой молекулы выполнить трудно, но можно вычислить энергии для фрагментов, отдельных контактов. Такой подход позволит синтезировать обучающую выборку путем генерации множества примеров «аптамер такой-то формы — такая-то энергия», включая даже запрещенные состояния. Что значит запрещенные? Например, можно смоделировать нуклеотидную последовательность, которая по всем правилам не сможет образовать стабильную структуру или никак не поместится на заданный белок без стерических столкновений. У таких вариантов энергия будет крайне невыгодной (положительной). Добавив их в обучение, мы фактически показываем ИИ «вот так делать нельзя». Большие нейросети очень любят усреднять данные, если не дать негативных примеров, они сгладят экстремумы. Поэтому принципиально важно учить модель не только на хороших (рабочих) структурах, но и на плохих. Таким образом, мы заменяем отсутствующий объем PDB-структур обширным набором энергетических данных, и пусть ИИ учится предсказывать именно значения энергии, а не только взаимное расположение атомов.
Шаг 3. От базы энергий к физической модели системы
Что дает нам эта база энергий? С ее помощью можно обучать нейросети, которые будут очень быстро оценивать то, что обычный физический расчет считает долго, но с сопоставимой точностью.
В качестве базовой модели для оценки энергии сворачивания мы используем нейросеть Edgar. Она по одной лишь последовательности (строка из букв A/C/G/U(T)) и вторичной структуре (записанной в DotBracket-нотации) оценивает, насколько стабильна структура аптамера, которую предсказала AliNA (по предсказанию свободной энергии ΔG). Технически Edgar — графовая нейросеть. Нуклеиновые кислоты представляются в виде графа, где узлы — отдельные нуклеотиды, а два типа ребер — типы связей между ними (ковалентные и нековалентные).
Для каждого нуклеотида строится числовое представление структуры со всей информацией, о которой необходимо знать нейронной сети (т.н. эмбеддинг), с учетом типа основания, положения в цепи и типа молекулы (РНК или ДНК), после чего сверточный слой моделирует ковалентные связи и локальное окружение. Информация о вторичной структуре обрабатывается в отдельном графовом слое. Комплементарные пары из DotBracket-нотации и одиночные нуклеотиды проходят через разные линейные слои, так что модель явно видит нековалентные связи. Затем слой Transformer собирает глобальный контекст по всей последовательности, а общий линейный слой предсказывает для каждого нуклеотида его вклад в энергию сворачивания и неуверенность этой оценки. Усреднив эти значения по цепи, получаем итоговую энергию сворачивания аптамера и доверительный интервал к этому предсказанию.
На тестовом наборе (структуры, не встречавшиеся при обучении) R2≈0,95, то есть модель объясняет ~95% дисперсии энергий. Для ДНК (которых в обучении было на порядок меньше, чем РНК) точность тоже высокая (R2≈0,85). Это говорит о хорошей обобщающей способности, то есть нейросеть уловила общие закономерности.
AliNA и Edgar совместно позволяют предсказать для любой заданной последовательности, как она сложится и насколько прочна будет ее структура. Они работают быстро, что принципиально важно, ведь впереди у нас перебор тысяч и миллионов вариантов. Получается некий цифровой эквивалент эксперимента по отбору стабильных структур.
Также были обучены несколько нейросетей для других задач. Одна предсказывает энергию локальных взаимодействий для аптамера и белка (InNA), другая оценивает изменение свободной энергии образования комплекса с учетом результатов InNA и Edgar (ValentiNA).
Замена мокрого SELEX на цифровой дизайн
За несколько лет мы в Xelari собрали набор алгоритмов: для предсказания стабильной структуры аптамера, для оценки энергии взаимодействия между аптамером и его мишенью и стабильности их комплекса. Объединив все алгоритмы и добавив детерминистические (основанные на физических принципах) методы для уточнения этих предсказаний, мы получаем более надежную модель. Таким образом, в нашем распоряжении оказывается полный набор инструментов: и классические физические методы, и ИИ-модели, которые позволяют провести полный цикл эксперимента от дизайна последовательности до оценки комплекса аптамера с его мишенью с высокой скоростью.
Что, если вместо месяцев лабораторного SELEX вы могли бы нажать кнопку, и система сама бы подбирала оптимальный аптамер под ваш белок-мишень?
Именно это делает платформа Xelari (рис. 5). Она представляет собой набор описанных выше алгоритмов, которые оркестрируются ИИ-агентом для получения стабильного аптамера с высокой специфичностью и сродством. Вместо случайной эволюции платформа рационально конструирует аптамер, опираясь на знания о мишени и поведении нуклеиновых кислот. Архитектурно Xelari — модульная система.
Работа платформы начинается с подготовки белка: она корректируется, устраняются пропуски, проводится релаксация, а затем анализируется поверхность и выбирается участок, наиболее подходящий для связывания. Подходящим является сайт, наименее похожий на другие белки, чтобы аптамер не лип ко всему подряд — так повышается специфичность; — а также содержащий оптимальную геометрию и соотношение полярных аминокислотных радикалов к общему числу радикалов — так повышается аффинность.
Алгоритм DiNA размещает короткие фрагменты олигонуклеотидов на поверхности. Проще говоря, система наращивает на белке кусочки ДНК/РНК там, где им выгодно сесть, притом учитывая физику взаимодействий. Далее система объединяет эти фрагменты в цельную структуру. За этот этап отвечает технология MoNA, использующая модели AliNA и Edgar для проверки стабильности структуры аптамера.
На этом этапе Xelari сгенерированы тысячи кандидатов. За счет комбинаций разных размещений фрагментов и их последовательностей получается большой пул вариантов аптамеров, все — построенные in silico под заданный участок белка. Вместо единичных последовательностей, как в классическом SELEX, мы имеем десятки тысяч смоделированных аптамеров, готовых к оценке и сравнению. Их качество уже заведомо выше случайной библиотеки, ведь они смоделированы под поверхность мишени и проверены.
Полученные комплексы проходят ускоренную, но точную молекулярную динамику (INGA) — это нужно, чтобы аптамер лег в наиболее энергетически выгодную позу на белке. Модель ValentiNA вычисляет изменение свободной энергии связывания комплекса аптамера с белком, что позволяет отобрать набор последовательностей с прогнозируемой константой диссоциации (Kd) в желаемом диапазоне. В последнюю очередь в аптамеры вносятся химические модификации с перерасчетом энергии комплекса и Kd.
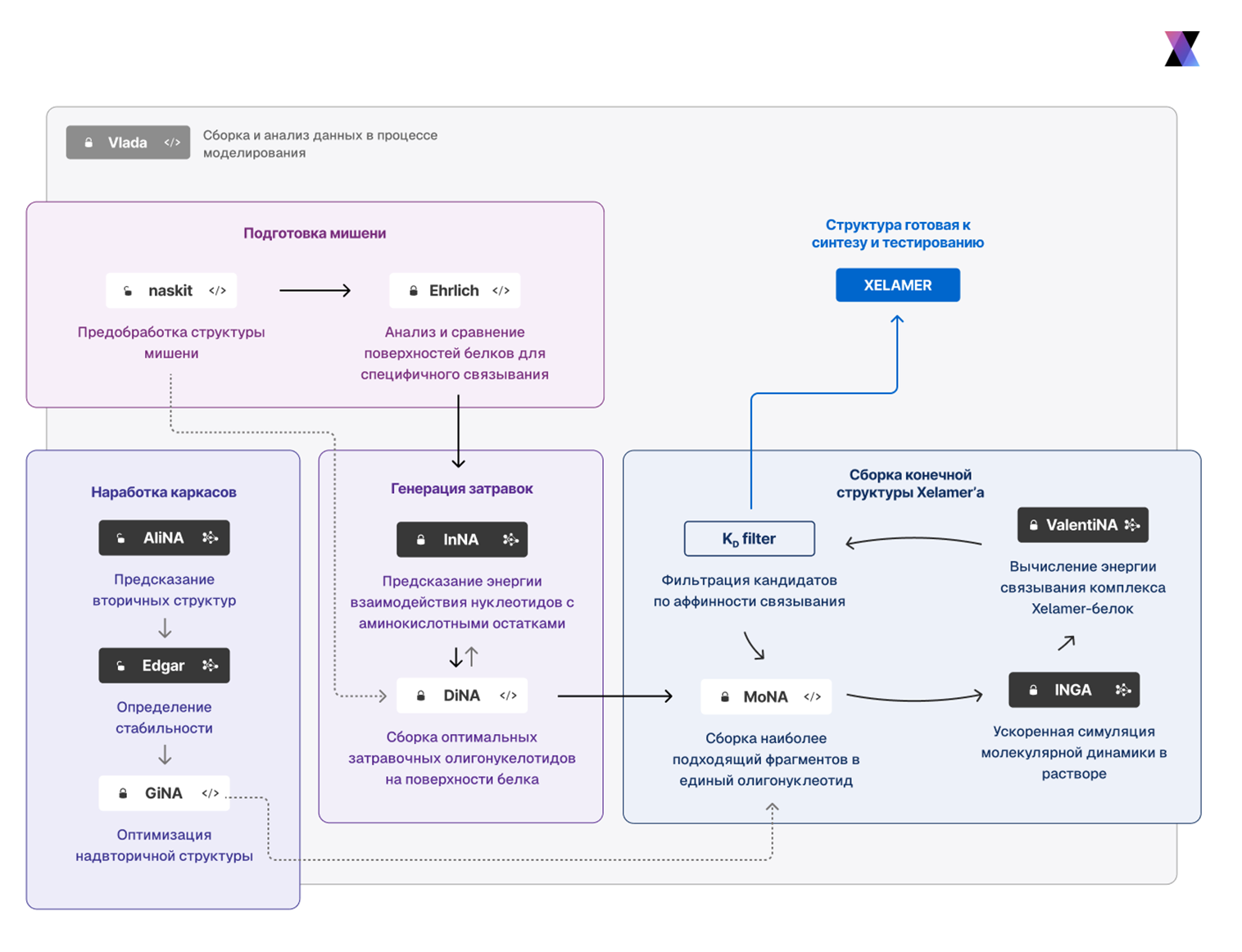
Рисунок 5. Упрощенный пайплайн платформы Xelari. На первом этапе подготавливается структура белка, устраняются пробелы и выбирается участок поверхности, оптимальный по уникальности, составу и геометрии для специфического связывания. Затем алгоритмы размещают на этом участке короткие фрагменты нуклеиновой кислоты, объединяют их в цельный аптамер, предсказывают его вторичную структуру и стабильность, моделируют трехмерный комплекс с белком и рассчитывают энергии сворачивания и связывания (включая прогноз константы диссоциации).
иллюстрация Никиты Парфенова
В финале пользователь получает конкретные последовательности, которые с высокой вероятностью являются аффинными и специфичными аптамерами к заданной мишени. Эти кандидаты уже можно синтезировать и проверять экспериментально. А главное, к этому моменту прошло максимум 24 часа ожидания.
Именно так поступили ученые из компании Nanohmics (США), одни из наших ранних клиентов. Судя по выбору цели, коллеги были уверены в нашем подходе и для проверки выбрали одну из самых капризных мишеней. Treponema pallidum — бактерия, которая вызывает сифилис, с ней очень неудобно работать вне организма хозяина. За 72 часа было спроектировано три аптамера, нацеленных на три разных белка этой бактерии. За предыдущие годы ни аптамеры с помощью SELEX, ни надежные антитела к ней компании разработать не удалось. А цель важная — прямая диагностика сифилиса, который, как выяснилось, набирает обороты в мире [40].
Эти аптамеры синтезировали и сразу проверили на живых бактериях. В экспериментах получили дозозависимое связывание (чем больше бактериальных клеток, тем сильнее сигнал). Перекрестную реактивность тоже посмотрели — аптамеры не связывались с другими грамотрицательными бактериями, что говорит о высокой специфичности. Учитывая, насколько сложно работать с T. pallidum, это оказалось безусловной победой модульного подхода Xelari, о чем исследователи с радостью сообщили нам (и дали разрешение на публичную огласку до научной публикации) [41].
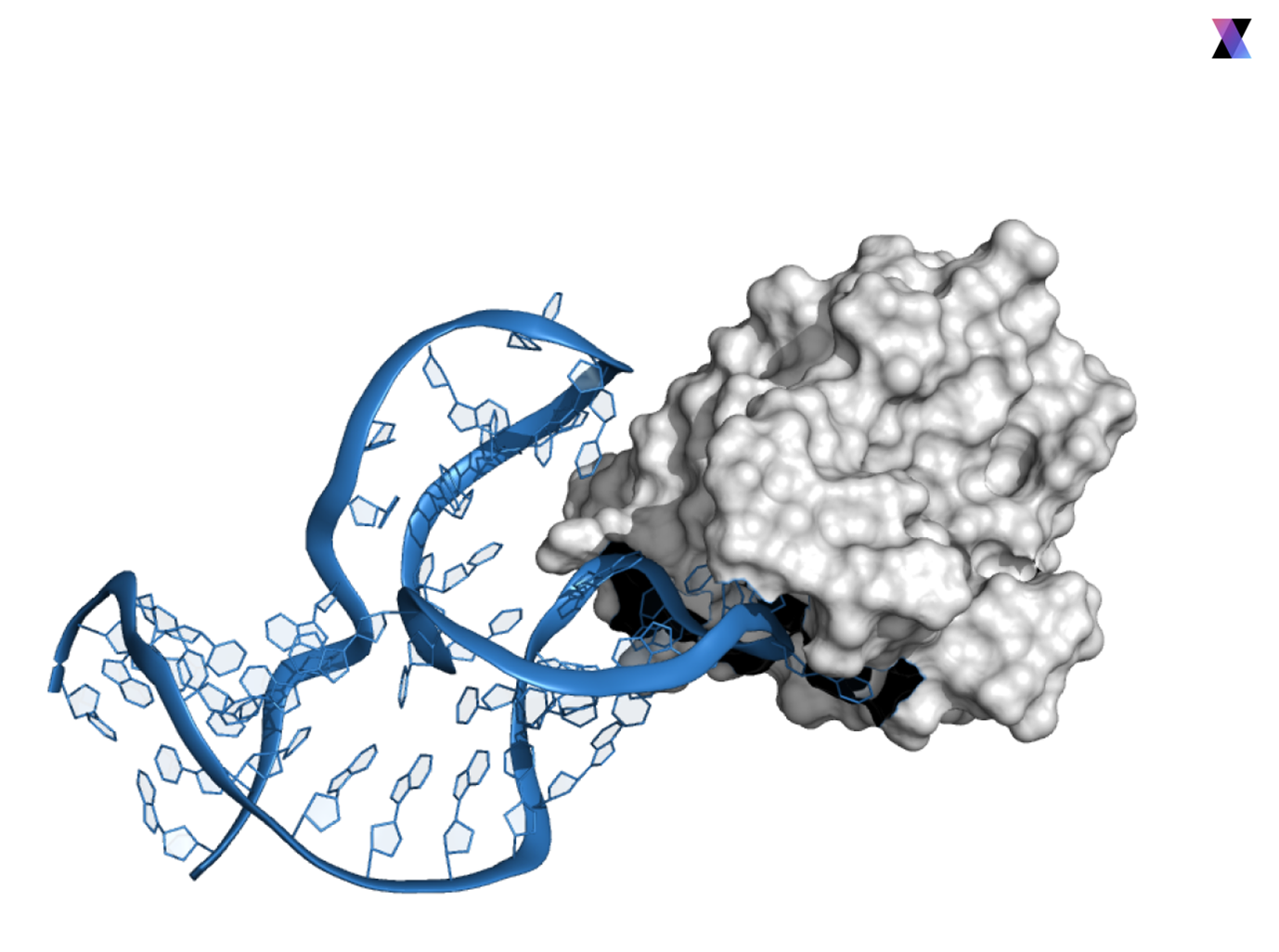
Рисунок 6. Трехмерная структура комплекса аптамера с целевым белком.
иллюстрация Никиты Парфенова
Таким образом, Xelari реализует полностью компьютерный цикл дизайна аптамеров. Конечно, финальная проверка — в лаборатории, ее никто не отменял. Но мы кардинально увеличиваем вероятность успеха и масштабируем процесс, вместо одного аптамера за раз можно параллельно проектировать десятки.
Заключение. Реабилитация аптамеров через отказ от SELEX
Аптамеры почти тридцать лет оставались в статусе несостоявшейся технологии. Их свойства когда-то привлекли много внимания, но главным ограничением оказался способ их получения. Классический SELEX, по сути, представляет собой поиск иголки в стоге сена: случайные библиотеки, долгие циклы отбора и высокий процент неудач. Мы аргументировали, что, помимо прочего, именно SELEX тормозил продвижение аптамеров, а не недостатки самих молекул: провалы за последние десятилетия почти всегда приходились на стадию отбора, тогда как те немногочисленные аптамеры, дошедшие до клиники (Macugen, Izervay), вели себя как обычные биопрепараты и демонстрировали типичные ограничения, а не системные проблемы класса.
Ситуация начала меняться с развитием биоинформатики и методов машинного обучения. Их сочетание создало предпосылки для скачка в аптамерной области. Успех AlphaFold на белках доказал, что такой подход действительно работает, но прямой перенос этого же подхода на аптамеры был бы неэффективным.
Для нас этот проект — способ вернуть аптамерам шанс, который они недополучили из-за ограничений SELEX, и показать, что сочетание ИИ и физики может работать не только на белках, но и на нуклеиновых кислотах. Наша команда реализовала подход к рациональному дизайну аптамеров в виде комплекса технологий Xelari. Вместо того, чтобы вслепую перебирать последовательности, мы сразу конструируем аптамер под заданный белок, опираясь на его структуру и поведение нуклеиновых кислот. Задача разбита на несколько шагов: предсказание структуры аптамера, оценка ее стабильности, моделирование комплекса с мишенью, расчет аффинности и специфичности связывания. В совокупности это превращает поиск оптимального аптамера именно в его проектирование, убирая ключевые ограничения SELEX. Такой подход, подкрепленный постоянно расширяющимися базами данных и совершенствующимися алгоритмами, дает аптамерам новый шанс.
В конце концов, вопрос уже не в том, возможно ли спроектировать in silico идеальный аптамер, а в том, когда первый такой аптамер займет свое место: в диагностике, терапии или доставке лекарств.
Литература
- Liangjie Feng, Yu Sun, Wenshen Jia, Yang Yu, Chang Liu, et. al.. (2025). Advancements in SELEX Technology for Aptamers and Emerging Applications in Therapeutics and Drug Delivery. Biomolecules. 15, 818;
- Таргетная терапия — прицельный удар по болезни;
- Три поколения лекарств;
- Краткая история открытия и применения антител;
- Антитело: лучший способ распознать чужого;
- Терапевтические моноклональные антитела;
- Аптамеры: графический гайд;
- C Tuerk, L Gold. (1990). Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510;
- Andrew D. Ellington, Jack W. Szostak. (1990). In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822;
- Eugene W. M. Ng, David T. Shima, Perry Calias, Emmett T. Cunningham, David R. Guyer, Anthony P. Adamis. (2006). Pegaptanib, a targeted anti-VEGF aptamer for ocular vascular disease. Nat Rev Drug Discov. 5, 123-132;
- 12 методов в картинках: полимеразная цепная реакция;
- Natalia Komarova, Alexander Kuznetsov. (2019). Inside the Black Box: What Makes SELEX Better?. Molecules. 24, 3598;
- Kelsey Pobanz, Andrej Lupták. (2016). Improving the odds: Influence of starting pools on in vitro selection outcomes. Methods. 106, 14-20;
- Michael Kohlberger, Gabriele Gadermaier. (2022). SELEX: Critical factors and optimization strategies for successful aptamer selection. Biotech and App Biochem. 69, 1771-1792;
- Michael Famulok, Günter Mayer. (2014). Aptamers and SELEX in Chemistry & Biology. Chemistry & Biology. 21, 1055-1058;
- Hadi Bakhtiari, Abbas Ali Palizban, Hossein Khanahmad, Mohammad Reza Mofid. (2021). Novel Approach to Overcome Defects of Cell-SELEX in Developing Aptamers against Aspartate β-Hydroxylase. ACS Omega. 6, 11005-11014;
- Nathalie Paniel, Georges Istamboulié, Athar Triki, Clément Lozano, Lise Barthelmebs, Thierry Noguer. (2017). Selection of DNA aptamers against penicillin G using Capture-SELEX for the development of an impedimetric sensor. Talanta. 162, 232-240;
- Congsheng Cheng, Yong Hong Chen, Kim A Lennox, Mark A Behlke, Beverly L Davidson. (2013). In vivo SELEX for Identification of Brain-penetrating Aptamers. Molecular Therapy - Nucleic Acids. 2, e67;
- Проблема фолдинга белка;
- Торжество компьютерных методов: предсказание строения белков;
- 12 методов в картинках: структурная биология;
- AlphaFold: нейросеть для предсказания структуры белков от британских ученых;
- Белковые галлюцинации: как справляется AlphaFold?;
- Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024);
- John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, et. al.. (2021). Highly accurate protein structure prediction with AlphaFold. Nature. 596, 583-589;
- История развития искусственного интеллекта и его пришествия в биологию;
- Как языковые модели покорили мир белков;
- Romualdo Troisi, Nicole Balasco, Ida Autiero, Luigi Vitagliano, Filomena Sica. (2023). Structural Insights into Protein–Aptamer Recognitions Emerged from Experimental and Computational Studies. IJMS. 24, 16318;
- M.A. Kazanskii, L. Uroshlev, F. Zatylkin, I. Pospelova, O. Kantidze, Y. Gankin AutoRNA: RNA tertiary structure prediction using variational autoencoder — Cold Spring Harbor Laboratory;
- M. Zuker. (2003). Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Research. 31, 3406-3415;
- Thomas Binet, Séverine Padiolleau-Lefèvre, Stéphane Octave, Bérangère Avalle, Irene Maffucci. (2023). Comparative Study of Single-stranded Oligonucleotides Secondary Structure Prediction Tools. BMC Bioinformatics. 24;
- Shamsudin S. Nasaev, Artem R. Mukanov, Ivan I. Kuznetsov, Alexander V. Veselovsky. (2023). AliNA – a deep learning program for RNA secondary structure prediction. Molecular Informatics. 42;
- Kevin A Wilkinson, Edward J Merino, Kevin M Weeks. (2006). Selective 2′-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat Protoc. 1, 1610-1616;
- Peter Steffen, Björn Voß, Marc Rehmsmeier, Jens Reeder, Robert Giegerich. (2006). RNAshapes: an integrated RNA analysis package based on abstract shapes. Bioinformatics. 22, 500-503;
- Jaswinder Singh, Jack Hanson, Kuldip Paliwal, Yaoqi Zhou. (2019). RNA secondary structure prediction using an ensemble of two-dimensional deep neural networks and transfer learning. Nat Commun. 10;
- Eleonora De Leonardis, Benjamin Lutz, Sebastian Ratz, Simona Cocco, Rémi Monasson, et. al.. (2015). Direct-Coupling Analysis of nucleotide coevolution facilitates RNA secondary and tertiary structure prediction. Nucleic Acids Res. gkv932;
- Debora S. Marks, Lucy J. Colwell, Robert Sheridan, Thomas A. Hopf, Andrea Pagnani, et. al.. (2011). Protein 3D Structure Computed from Evolutionary Sequence Variation. PLoS ONE. 6, e28766;
- Sergey Ovchinnikov, Lisa Kinch, Hahnbeom Park, Yuxing Liao, Jimin Pei, et. al.. (2015). Large-scale determination of previously unsolved protein structures using evolutionary information. eLife. 4;
- Nicholas P. Schafer, Bobby L. Kim, Weihua Zheng, Peter G. Wolynes. (2014). Learning To Fold Proteins Using Energy Landscape Theory. Israel Journal of Chemistry. 54, 1311-1337;
- Noah Kojima, Jeffrey D Klausner. (2018). An Update on the Global Epidemiology of Syphilis. Curr Epidemiol Rep. 5, 24-38;
- John G. Bruno, Shamsudin Nasaev, Dmitry Ufaev et al. (2025). Evaluation of Artificial Intelligence-Generated DNA Aptamers Against Treponema pallidum Surface Proteins. Preprint (Version 1) available at Research Square.