Клеточная, молекулярная и системная биология: как биологи и биоинформатики вместе рисуют общую картину жизни
16 октября 2020
Клеточная, молекулярная и системная биология: как биологи и биоинформатики вместе рисуют общую картину жизни
- 3025
- 2
- 18
Главная задача биологии как науки — разгадать тайну жизни
иллюстрация Любови Колосовской
-
Автор
-
Редакторы
-
Иллюстратор
Ежегодно, начиная с 2016 года, российские аффилированные лица компании «Филип Моррис Интернэшнл» (ФМИ) вручают на базе Сколковского института науки и технологий стипендию выдающимся молодым ученым, работающим в сфере системной биологии. Эта статья продолжает цикл «Пятерка по системной биологии». В ней мы расскажем, как системная, молекулярная и клеточная биология помогают составить наиболее полное представление о работе живых систем и попросим стипендиатов программы рассказать о своих научных работах — и о том, какой вклад они должны внести в наши представления о жизни.
Пятерка по системной биологии
Победителями Стипендиальной программы по системной, клеточной и молекулярной биологии, организованной российскими аффилированными лицами компании «Филип Моррис Интернэшнл» (ФМИ) на базе Центра наук о жизни Сколковского института науки и технологий, ежегодно становятся пять молодых ученых до 35 лет. Знания по своим темам у стипендиатов, без сомнения, также заслуживают «пятерки» — поэтому спецпроект и получил такое название.
В статьях цикла наши авторы пообщаются со стипендиатами разных лет, профессорами и преподавателями Сколтеха, а также с представителями ФМИ.
Партнер спецпроекта — российские аффилированные лица компании «Филип Моррис Интернэшнл» (ФМИ), которая активно ведет исследования в области биомедицины, системной биологии и биотехнологий.
Разные способы взглянуть на жизнь: «организменная», клеточная, молекулярная и системная биология
Биология — наука о жизни во всех ее проявлениях. Это огромная тема, которая включает самые разные проявления живого: от связей организмов на уровне биосферы до биохимических реакций и молекулярных взаимодействий, которые протекают в клетках и делают возможной саму жизнь (рис. 1).
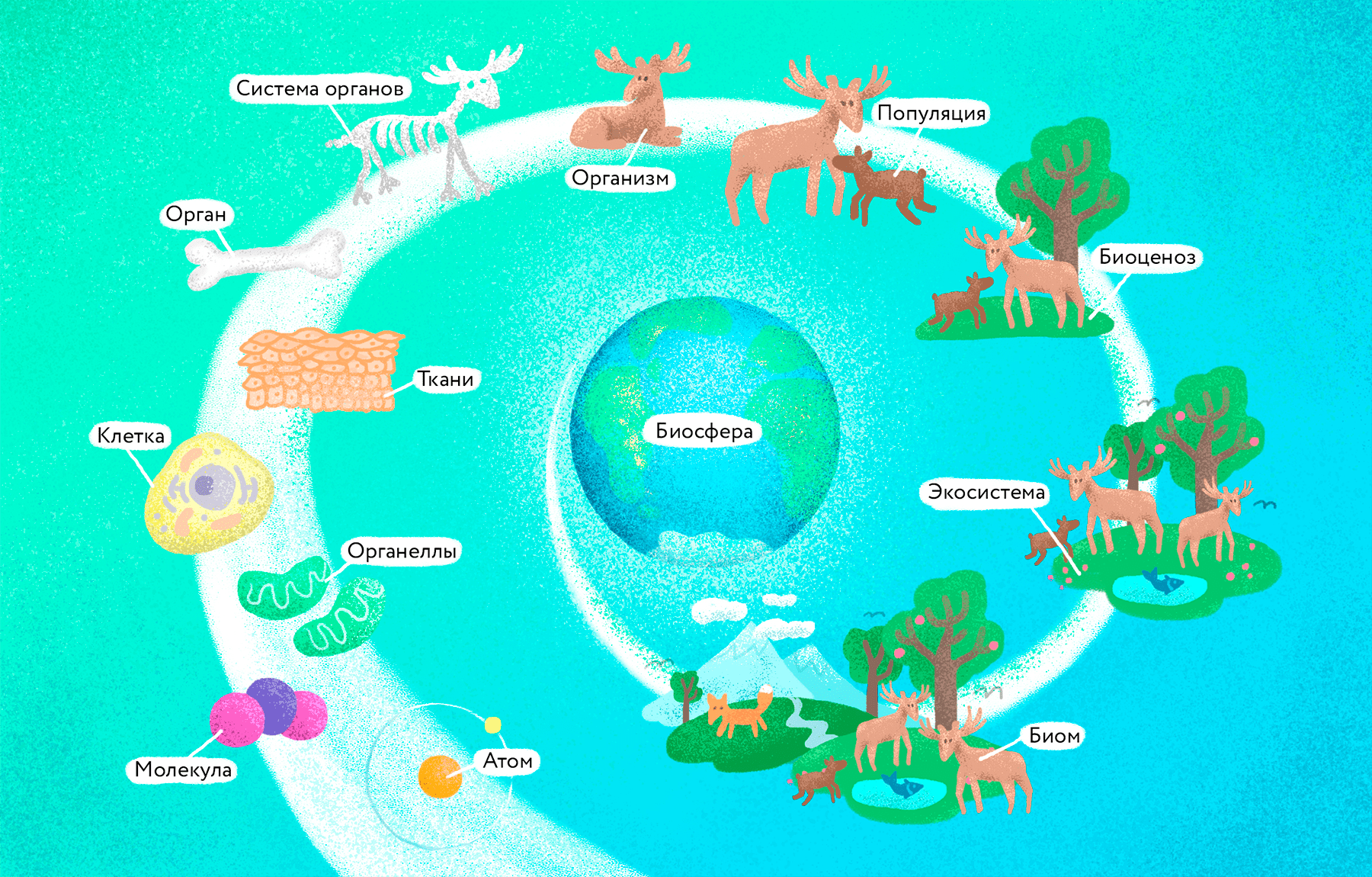
Рисунок 1. Биология изучает всё — от связей организмов с окружающей средой до межмолекулярных взаимодействий
иллюстрация Любови Колосовской по [1]
Способов взглянуть на конкретный организм существует очень много. Живые организмы — сложные системы с несколькими уровнями организации. Специалисты из различных биологических областей проводят границу между уровнями по-своему. Но если немного упростить, то ученые, которые стремятся разгадать тайну жизни, идут несколькими путями [1]:
- Смотрят на организм в целом. Это самое старое направление биологии, которое берет свое начало еще от Аристотеля и Галена, постепенно превращаясь в науку, начиная с XVIII и до конца XIX века.
«Организменной» биологией занимаются зоологи и ботаники. Их интересует, как развиваются, питаются, общаются, размножаются и погибают объекты их изучения — организмы исследуемого вида. «Организменные» биологи изучают, как устроены системы, в которые объединены внутренние органы, и исследуют ткани, из которых эти органы состоят. И тут мы вплотную подбираемся к основе основ всех живых организмов — к клетке. Но их изучают уже другие специалисты. - Изучают работу клеток. Клетки — основные, базовые единицы почти всех живых организмов. Изучение клеток тоже началось очень давно — с конца XVII века, когда Антони ван Левенгук впервые описал свои наблюдения за живыми клетками под микроскопом. О том, как эволюционировал этот замечательный прибор со времен Левенгука, рассказано в статье о микроскопии [2] из спецпроекта «12 биометодов».
Некоторые ученые относят к бесклеточным формам жизни (Acellularia, Acytota, Aphanobionta) вирусы, вироиды и вирусоиды.
В XIX веке Маттиас Шлейден и Теодор Шванн сформулировали первое определение клетки, которое позже дополнил Рудольф Вирхов. Так это определение превратилось в знакомое нам со школы: «Клетка — это единая система, включающая множество закономерно связанных между собой элементов, представляющих целостное образование, состоящее из сопряженных функциональных единиц — органелл. Каждая клетка происходит только путем деления материнской клетки».
Современная клеточная биология (в нашей стране ее часто называют цитологией) изучает внутреннюю структуру и функции клеток, то, как клетки образуются и делятся, как приобретают присущие им свойства и специализацию. Клеточные биологи стремятся выявить как свойства, общие для большинства типов клеток, так и уникальные особенности специализированных клеток, позволяющие им выполнять различные функции.
Исследования в области клеточной биологии тесно связаны с другими областями биологии — генетикой, молекулярной генетикой, биохимией и молекулярной биологией, медицинской микробиологией, иммунологией и цитохимией.
Подробнее о том, как работает клетка, и о самых интересных исследованиях клеточной биологии можно прочитать на нашем сайте в разделе «Цитология». Как именно биологи используют клетки в качестве модельного биологического объекта, рассказывает статья из спецпроекта «12 биометодов»: «Клеточные технологии» [3]. - Исследуют живое на молекулярном уровне. Всеми видами биологической активности на уровне макромолекул занимается молекулярная биология. Это одно из самых молодых направлений биологии — Уоррен Уивер придумал термин «молекулярная биология» совсем недавно, в 1983 году [4].
Молекулярная биология занимается всеми молекулярными механизмами, которые лежат в основе различных биологических процессов: транскрипции, репликации, трансляции и других функций и «задач» макромолекул, не связанных с геномом. Под «не связанными с геномом макромолекулами» обычно подразумевают белки, а также макромолекулярные комплексы — хромосомы, рибосомы, мультиферментные системы, обеспечивающие биосинтез белков и нуклеиновых кислот.
Больше узнать о том, чем занимаются ученые, избравшие для себя это направление, можно из нашей статьи «Молекулярная биология» [5]. О возможностях, которые появились у современных молекулярных биологов, прочитайте в статье из спецпроекта «12 биометодов»: «Генная инженерия» [6].
Исследования в области молекулярной биологии неизбежно пересекаются и смешиваются с работами из других областей науки: от химии и физики до всех разделов биологии. Разумеется, с клеточной биологией они тоже связаны самым тесным образом. - Пытаются взглянуть на жизнь в целом. Как мы видим, все направления биологии контактируют очень плотно — настолько, что иногда даже сложно провести между ними границу. Однако это совсем не означает, что данные, накопленные разными направлениями наук о живом, легко объединяются и встраиваются в общую картину. Чтобы понять, как взаимосвязаны компоненты живого — от макромолекул до организмов, исследователи выделили еще одно исследовательское направление — системную биологию.
Определение системной биологии дать крайне сложно — это признают сами системные биологи. Например, Рон Жермен, руководитель лаборатории системной биологииNIAID (National Institute of Allergy and Infectious Diseases), объясняет, что одни ученые подразумевают под «системной биологией» работу биоинформатиков, которые обрабатывают огромное количество экспериментальной информации, полученной клеточными и молекулярными биологами. Другие исследователи склонны считать, что «системная биология» — это вычислительная биология. Занятые в этом направлении биоинформатики стремятся рассчитать, как работают биологические системы, и построить их компьютерные модели. О том, какие еще задачи решают биоинформатики, можно прочитать на нашем сайте в разделе «“Сухая” биология» — например, в статье «Я б в биоинформатики пошел, пусть меня научат!» [7].
По мнению самого Жермена, системная биология — это и то и другое сразу. Исследователь дал такое определение:
- Системная биология
- научный подход, сочетающий принципы инженерии, математики, физики и информатики с обширными экспериментальными данными для развития количественного и глубокого концептуального понимания биологических явлений, позволяющий предсказывать и точно смоделировать сложное поведение биологических объектов.
Системная биология стремится найти ответы на сложные вопросы, которые стоят перед современными биологами. Как организм отреагирует на новое лекарство или прививку недавно разработанной вакциной? Как может мутировать этот болезнетворный микроорганизм — и насколько опасным для людей может оказаться обновленный штамм? Подобных вопросов сотни, если не тысячи.
А поскольку все уровни организации живого неразрывно связаны друг с другом, стипендиальная программа, организованная российскими аффилированными лицами компании «Филип Моррис Интернэшнл» (PMI affiliates in Russia, ФМИ) на базе Сколковского института науки и технологий, не ограничивается только одним каким-то направлением и поддерживает молодых ученых, которые занимаются и системной, и клеточной, и молекулярной биологией. В этой статье мы расскажем о работах лауреатов стипендии, которые трудятся на стыке всех трех направлений: системной, клеточной и молекулярной биологии.
А если вы сейчас как раз раздумываете о поиске гранта для своего проекта и хотите заниматься наукой, а не бюрократией, то почитайте о стипендии и мнении тех, кто ее уже получил, в первой статье цикла: «Стипендия по системной биологии: инициатива, которая вдохновляет» [8].
Как найти малую молекулу в стоге сена
«“Малые природные соединения” в названии моего проекта — это буквальный перевод английского термина small natural products . В научном дискурсе этот термин (а иногда и просто natural products) употребляется для описания вполне определенного, пусть и очень широкого, класса химических соединений, — рассказывает Алексей Гуревич. — Наиболее близким русским аналогом будут “малые молекулы”. Правда, под этот термин подпадают как синтетические соединения, так и вещества природного происхождения, а в моей работе речь идет только о природных соединениях».
Название проекта, как и вся заявка на стипендию, подается на английском языке.
Разыскивать малые природные соединения (МПС) важно, потому что многие такие соединения обладают полезными биологическими свойствами, в первую очередь с точки зрения фармакологии. Например, МПС могут оказаться потенциальными антибиотиками, противовирусными и противоопухолевыми препаратами [9]. Конкретный пример полезных малых молекул, которые на слуху почти у всех — антибиотики: например пенициллин [10] или тетраценомицин X [11].
Для поиска МПС уже достаточно давно активно используют геномику (данные секвенирования организмов, способных продуцировать МПС) и метаболомику (данные масс-спектрометрического анализа самих МПС). Однако каждый из этих типов данных по отдельности не дает полной картины.
В отличие от «обычных» белков и пептидов, синтез МПС устроен достаточно сложно (рис. 3). Очень часто, чтобы понять, как МПС появляется в том или ином организме, нужно «расшифровать» запутанный многоступенчатый метаболический путь. Даже имея идеально прочитанный геном продуцента МПС (что в реальности далеко не всегда достижимо, ведь пока расшифрованы геномы далеко не всех микроорганизмов), предсказать, как и какие МПС он синтезирует, зачастую либо вообще невозможно, либо возможно — но лишь отчасти, и только для определенных классов МПС.
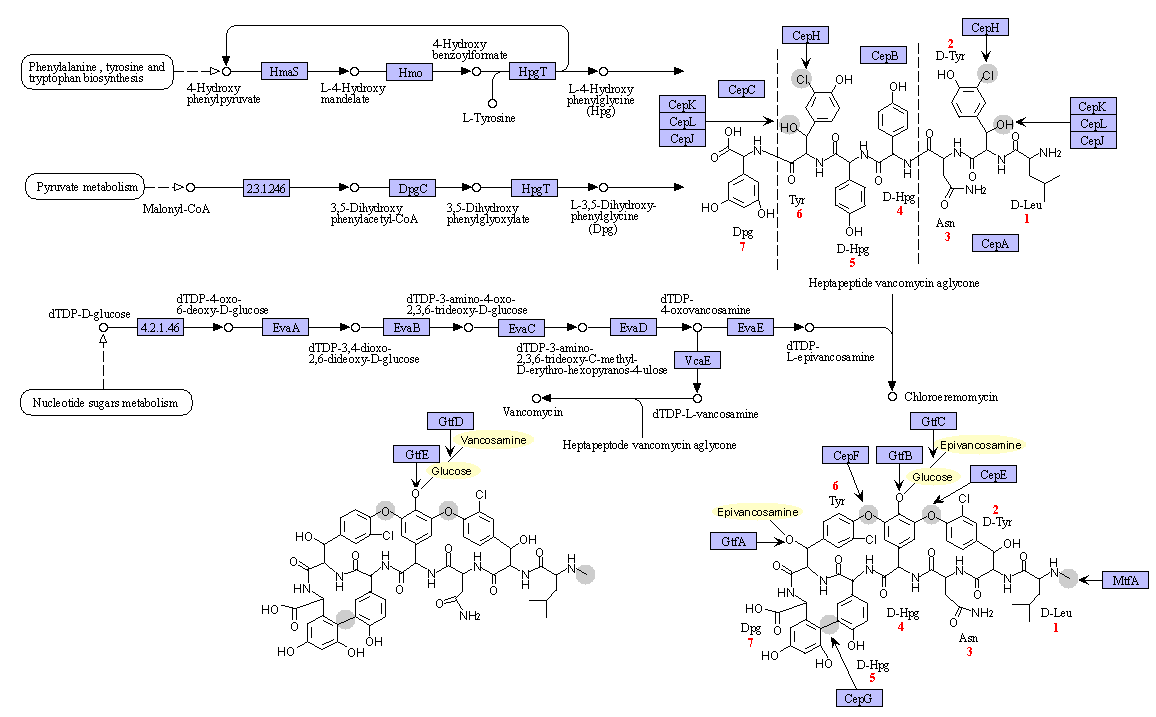
Рисунок 3. Биосинтез антибиотика ванкомицина. На схеме видно, что синтез МПС — очень сложный процесс.
Чаще всего геномные подходы используют, чтобы найти в новом геноме консервативные участки, похожие на уже изученные гены или кластеры генов, ответственные за синтез МПС. На основе этой информации делают предположения о МПС, которые способен производить изучаемый организм, и о том, какая у них может быть химическая структура. Примерно таким же образом биоинформатики из Сколтеха разыскивают новые антибиотики — микроцины [12].
В то же время с помощью масс-спектрометрических подходов исследователи пытаются выяснить структуру произведенного организмом МПС. В отличие от пептидов, для которых фрагментация (то есть разделение молекулы на части, чтобы их проще было изучать) в масс-спектрометрах достаточно хорошо изучена и в целом понятна, в случае МПС возникает много сложностей и потенциальных ошибок в анализе данных. Например, известно, что чаще всего пептиды «расщепляются» по пептидным (амидным) связям. Благодаря этому по масс-спектру можно попытаться «прочитать» исходный пептид «буква за буквой», аминокислота за аминокислотой. Если речь идет о МПС, то пептидных связей в их структуре может не быть в принципе. Да и количество возможных «букв», на которые МПС теоретически способны разбиться в масс-спектрометре, на несколько порядков превышает число стандартных аминокислот, которых всего 20 .
Некоторые исследователи считают, что на самом деле стандартных аминокислот не 20, а 21 или даже 22.
При использовании обоих типов данных о МПС возникает масса сложностей — а информации, которую можно выяснить таким образом, недостаточно. Объединение геномных и метаболомных данных позволяет посмотреть на исследуемые вещества с двух разных сторон одновременно. Это позволяет получить значительно больше информации, а также избежать ошибок, характерных для каждого подхода по отдельности.
«Упрощенно идея состоит в следующем. По геномным данным предсказывают большой набор возможных структур, а по масс-спектрам определяют, какая из них с наибольшей вероятностью синтезируется организмом, — говорит Алексей. — Иногда это еще и помогает определить посттрансляционные модификации в МПС, которые по геномным данным предсказать нельзя или очень сложно. Геномно-предсказанных структур могут быть тысячи или десятки тысяч, но правильные из них лишь единицы — а глядя исключительно на геном, отделить одни от других нельзя. Моделировать поведение в масс-спектрометре для тысяч соединений может быть долго, но это всё равно значительно быстрее, чем моделировать все возможные гипотетические МПС. А еще при таком подходе меньше вероятность сделать ложное предсказание, чем при “свободном” поиске».
Объединение двух типов информации — перспективная идея. При этом Алексей Гуревич признает, что у такого подхода может возникнуть две проблемы.
Во-первых, биоинформатики, работающие с МПС, чаще всего занимаются либо геномикой, либо метаболомикой (либо вообще смотрят на них с какой-то третьей стороны), но не тем и другим вместе. Во многом это два отдельных сообщества, которые ездят на разные конференции и публикуются в разных журналах. Чтобы заниматься интеграцией данных геномики и метаболомики, нужно все-таки хорошо понимать, какие подходы и программы есть в каждой из этих областей, какие конкретные форматы данных там применяются, какие в данных бывают ошибки.
Во-вторых, в проекте придется иметь дело с большими объемами как геномных, так и метаболомных данных. Типичная задача — сопоставить миллионы масс-спектров с тысячами (а иногда — с десятками тысяч) возможных структур МПС, предсказанных по одному генному кластеру. При этом генных кластеров в одном организме вполне может быть несколько. Да и самих организмов может быть тоже много — если предстоит работа с метагеномным образцом или если захочется выполнить сопоставление со всеми бактериями определенного рода или семейства. Ведь зачастую точный продуцент МПС неизвестен или не может быть культивирован в лаборатории.
Чтобы выполнить задачу, потребуется написать высокопроизводительный программный код и придумать эффективные алгоритмические подходы, — так решение конкретных практических задач не будет занимать бесконечное время.
Алексею Гуревичу посчастливилось поработать по несколько лет и в геномике, и в метаболомике. Кроме того, вся его научная деятельность напрямую связана с написанием эффективного научного программного обеспечения, и с придумыванием алгоритмических решений для обработки больших объемов данных. Таким образом, вводные данные для решения основных проблем проекта у него подходящие.
В рамках проекта Алексей планирует работать с тремя конкретными классами МПС:
- Рибосомно синтезированные пептиды с посттрансляционными модификациями (RiPP) —пептиды, синтезированные на рибосомах и модифицированные уже после завершения синтеза [13]. Посттрансляционные модификации позволяют увеличить разнообразие белков в клетке.
- Нерибосомные пептиды (NRP) — пептиды, синтезированные напрямую ферментом синтетазой без участия мРНК. Нерибосомные пептиды синтезируют микроорганизмы: бактерии и грибы.
- Поликетиды (PK) — поликарбонильные соединения, вторичные метаболиты, образующиеся в клетках бактерий, грибов, животных и растений. К этому классу химических соединений относятся, например, антибиотики и токсины.
Эти три класса — одни из наиболее хорошо изученных в плане синтеза. И кроме того к ним относят множество полезных и используемых в индустрии и медицине МПС. Для первых двух классов (RiPP и NRP) у Алексея вместе с коллегами уже есть начальные версии метабологеномных инструментов, которые они и собираются взять за основу стартового этапа проекта.
«На начальном этапе я планирую изучить и протестировать на различных данных все наиболее популярные, а также самые новые программы для предсказания RiPP и NRP по геномным данным. С частью из этих программ я уже успел поработать раньше, но и новые для меня инструменты определенно будут. Наиболее успешные по результатам тестов программы будут использованы для улучшения имеющихся метабологеномных прототипов», — делится Алексей.
После завершения начального этапа Алексей Гуревич хочет перейти к созданию первой версии метабологеномной программы-поисковика для поликетидов. Эта программа будет похожа на уже существующую программу для поиска NRP [14], так как в синтезе этих веществ есть много общего. Однако придется учесть и ряд важных особенностей. В частности, если NRP все-таки состоит из аминокислот (пусть и далеко не всегда только стандартных), то в чистых поликетидах их нет. Так что программу предстоит существенно доработать.
Конечным результатом проекта должен стать ряд самостоятельных программных инструментов для поиска отдельных классов МПС и единый вычислительный конвейер, объединяющий все эти программы. В идеале должно получиться «приложение с одной кнопкой» — то есть очень простая программа, предназначенная для пользователей с разным уровнем компьютерной грамотности.
Я хочу, чтобы исследователи просто загружали свои геномные и метаболомные данные, нажимали ту самую кнопку и через некоторое время получали ответ — какие МНП есть в их данных. В реальности, наверное, всё это будет выглядеть несколько сложнее, но буду стремиться к идеалу.
Если всё получится, разработанную программу можно будет применить к большим объемам как открытых данных, так и данных, полученных от коллабораторов Алексея Гуревича. Он рассчитывает, что при анализе этих материалов получится найти ряд новых МПС и, если всё сложится хорошо, экспериментально подтвердить эти находки и их биологическую активность (рис. 4).
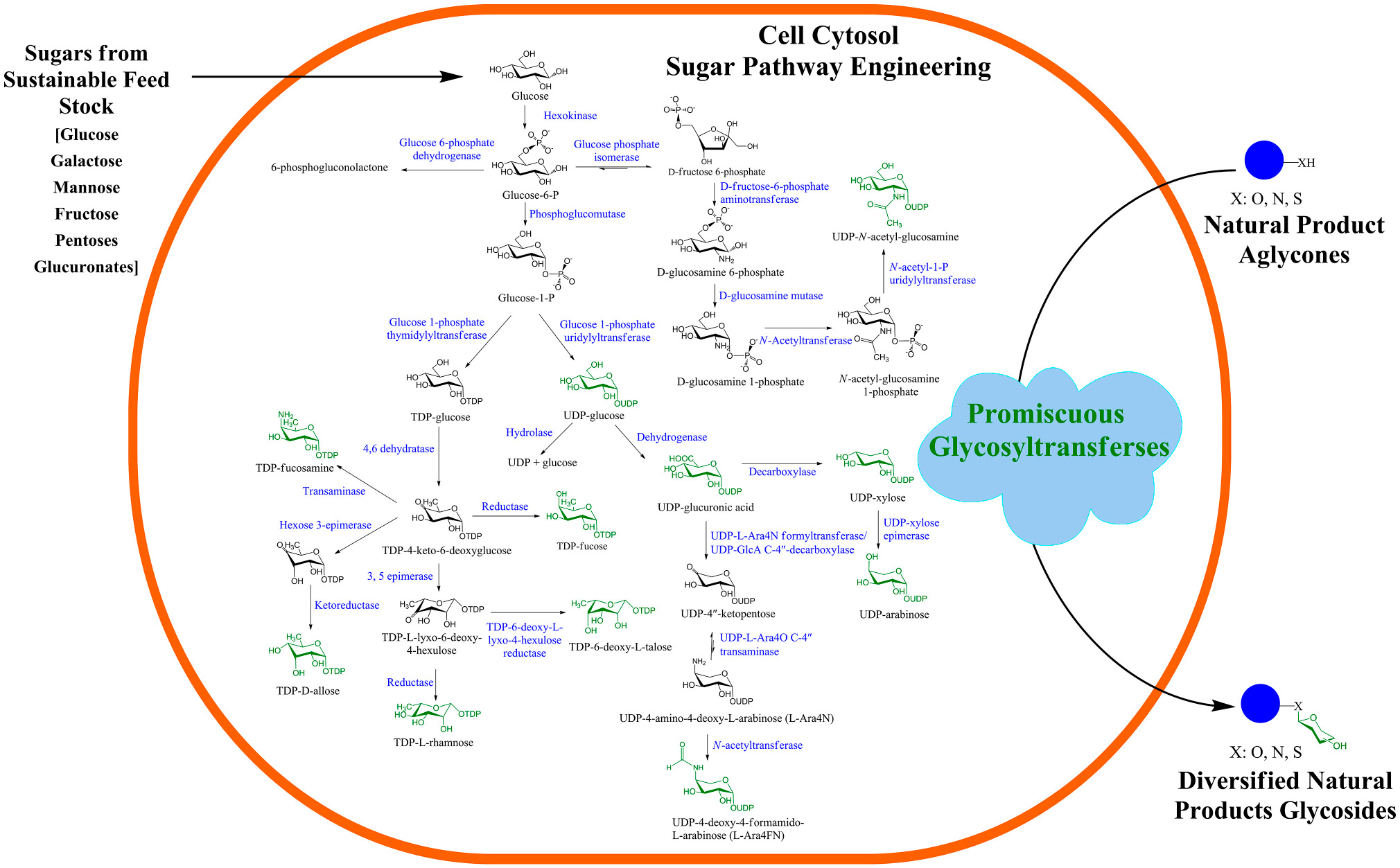
Рисунок 4. Пример того, как можно было бы использовать знания о синтезе МПС. На схеме показано, как должна быть устроена гипотетическая фабрика по созданию «малых природных соединений» в искусственно сконструированной микробной клетке. Такие клетки можно было бы использовать в качестве «заводов» для промышленного производства ценных гликозидов — соединений, на основе которых создают новые лекарства.
В конечном счете, если в наличии у исследователей будут необходимые данные обоих видов, то, применяя созданный в рамках работы программный продукт, они получат вычислительные предсказания возможных МПС в образце. Это существенно упростит и ускорит дальнейшую исследовательскую работу по поиску новых соединений.
Как найти связь между человеческим генотипом и фенотипом
Генотип — это индивидуальный набор наследственной информации организма. Фенотип — наблюдаемые физические или биологические характеристики. На формирование внешних признаков организма влияет и генотип, и окружающая среда. Чтобы связать определенные генетические вариации с конкретными фенотипическими признаками (например, со стигматами, то есть внешними признаками наследственных заболеваний), применяют метод полногеномных исследований ассоциаций (GWAS) между геномными вариантами и фенотипическими признаками.
Метод включает сканирование геномов большого количества разных людей и поиск генетических маркеров, которые подходят для прогнозирования наличия наследственных признаков — скажем, болезни. После того как генетические маркеры идентифицированы, их можно использовать для понимания того, какие гены и как именно вносят вклад в заболевание. Это очень важно для разработки более эффективных стратегий профилактики и лечения.
При этом строить карты, отражающие связь генотипа и фенотипа (genotype-to-phenotype maps, или карты G-P), очень тяжело — ведь для этого нужны генетические данные для всего фенома, то есть для совокупности всех фенотипических черт организма. Такие данные можно получить, анализируя вклад генетических изменений в развитие каждого из всей совокупности признаков организма. Только после того как появился ресурс UK Biobank (Британский биобанк, UKB), который опубликовал набор генетических данных 500 000 человек, ученые по всему миру получили возможность всесторонне изучить генетическую архитектуру тысяч фенотипов человека.
Одно из таких исследований предприняла группа биологов под руководством Юрия Барбитова [15], в котором принимал участие биоинформатик Антон Шиков из Института биоинформатики (с ним мы еще встретимся ниже). Проанализировав открытые данные, они обнаружили, что высоко плейотропные варианты генов — варианты, которые влияют на множество фенотипических признаков, — часто соответствуют широко экспрессируемым генам с универсальными функциями. Например, это могут быть гены, связанные с работой иммунной системы, и гены-регуляторы роста клеток, но больше всего среди них генов, отвечающих за формирование внеклеточного матрикса.
Плейотропия — одно из ключевых свойств карт G-P. Она очень часто встречается у людей и других видов живых существ. Основная цель работы Юрия Барбитова — исследовать ключевые свойства карты G-P человека, причем особое внимание планируется уделить различным аспектам плейотропии.
«Этот проект — продолжение проекта с Антоном Шиковым [15]. Мне интересно взглянуть на то, как генетические факторы влияют на совокупность признаков, то есть феном, в целом. Здесь ключевой момент в том, чтобы найти точки — конкретные генетические замены, которые влияют сразу на многие признаки (это называется плейотропным действием гена), и проанализировать их с функциональной точки зрения. Проект будет основан на анализе данных ресурса UK Biobank, — рассказывает Юрий Барбитов. — Я намерен понять, как связаны между собой генотип и фенотип человека на системном уровне. Для этого я хочу воссоздать сеть G-P взаимосвязей, а также планирую посмотреть и на изменения этих сетей в эволюции между видами и внутри популяции. Так можно выяснить, как что-то меняется в структуре взаимосвязи генотипа и фенотипа — если, конечно, меняется».
Первая цель проекта — проанализировать плейотропные эффекты тканей и типов клеток человека. Для этого потребуется создать эффективный и чувствительный метод анализа набора генов, который нужно будет применить к общему набору данных британского биобанка. Решение этой задачи позволит лучше понимать вклад в формирование фенотипа не только отдельных генов, но и целых тканей и систем организма.
Вторая цель — исследовать модульность сети генотип-фенотип человека. Для этого планируется, основываясь на данных генетической ассоциации, построить карту взаимодействия человеческих генов и исследовать ее с помощью методов сетевого анализа (рис. 5). С такой картой проще разобраться, насколько жестко взаимосвязана функция гена с его влиянием на внешние признаки.
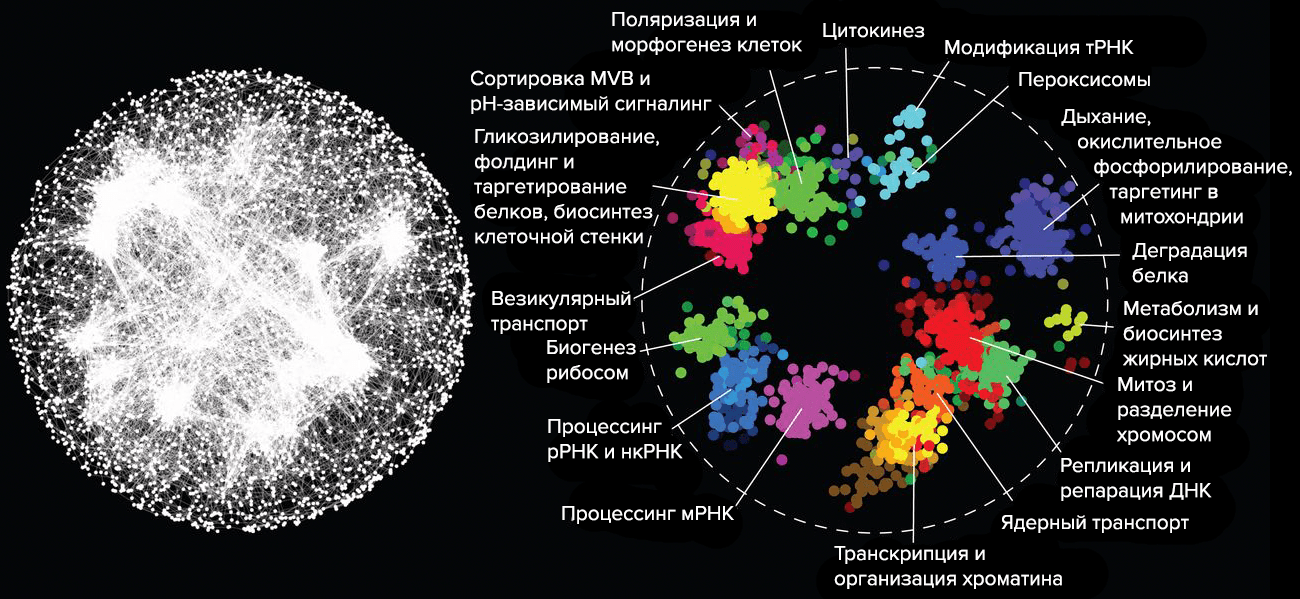
Рисунок 5. Примерно так может выглядеть карта взаимодействия генов. Условные обозначения: MVB — мультивезикулярные тельца; нкРНК — некодирующие РНК.
[28], рисунок адаптирован
Третья цель проекта — оценить эволюционное значение плейотропии и мутационного ландшафта плейотропных локусов в геноме человека. Современные данные свидетельствуют о сильном отрицательном отборе против высоко плейотропных вариантов генов. Эту гипотезу планируется проверить с использованием методов статистической и популяционной генетики.
В целом, в ходе проекта исследователь хочет получить представление о структуре отношений генотип—фенотип у людей. В частности, он стремится разобраться с масштабами и механизмами плейотропных эффектов, их эволюционными последствиями. Это поможет понять, как несколько генетических вариантов с небольшими эффектами формируют сложный фенотип.
Как «вычитать» в генах скрытую болезнь
Бóльшую часть генетических вариантов у людей накапливает некодирующая часть генома. Иногда генетические варианты достаются человеку по наследству, но могут появиться, например, в процессе накопления мутаций в раковой опухоли. Но и в том, и в другом случае они способны сломать регуляторные участки генов.
Благодаря современным технологиям секвенирования исследователи получают миллионы генетических вариантов, отличающих людей друг от друга. Например, если взять геномы больных диабетом, можно посмотреть, чем они отличаются от геномов здоровых людей. Однако мы получим тысячи сцепленных генетических вариантов, из которых только небольшая часть будет действительно влиять на регуляцию. Но это — не единственная проблема.
Хотя все клетки организма содержат примерно одинаковый геном, в разных клетках работают разные наборы генов. Значит, должны быть какие-то механизмы, определяющие активацию одних генов и бездействие других (это называется эпигенетикой). Один из таких механизмов опосредован работой транскрипционных факторов (ТФ) — белков, контролирующих синтез РНК с молекулы ДНК.
Как правило, ТФ «садится» на определенные короткие участки ДНК — сайты связывания. Эти сайты, как правило, располагаются неподалеку от генов. Но точно определить их местоположение бывает непросто, ведь участки генома, кодирующие белки, занимают меньше 2% ДНК, а крохотные регуляторные «островки» раскиданы по 98% «океана» остальной некодирующей части генома. Кроме того, порой различные транскрипционные факторы «узнают» иногда одинаковые, а иногда разные сайты связывания. Да еще и в различающихся условиях нередко работают по-иному.
«Наша задача — понять, какие из вариантов генов наиболее вероятны как причина изменений в экспрессии генов, — рассказывает Илья Воронцов. — Если мы научимся это определять, мы сможем подсказать экспериментаторам, на какие десять вариантов из тысячи возможных стоит обратить особое внимание при исследовании причин развития болезней. И, разумеется, лучше поймем, как вообще работают регуляторные механизмы генома».
Базовая гипотеза большинства методов приоритезации вариантов состоит в том, что если генетический вариант влияет на способность ТФ связываться с ДНК, то он может влиять и на транскрипцию. По этой причине необходимо изучать паттерны связывания ДНК с транскрипционными факторами (рис. 6). Пополнение коллекции таких паттернов HOCOMOCO (HOmo sapiens COmprehensive MOdel COllection — коллекция факторов транскрипции человека) — одна из задач, которой достигли исследователи.
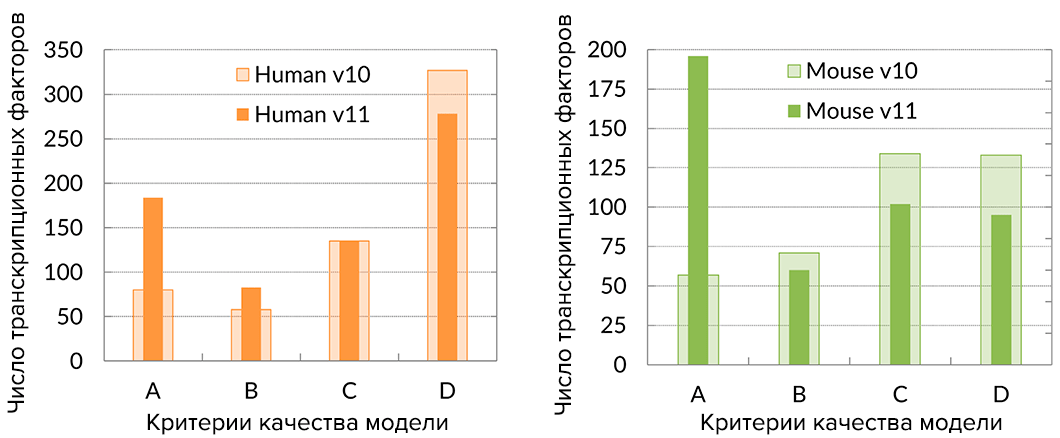
Рисунок 6. Две версии коллекции HOCOMOCO: старая (v10) и обновленная в результате исследований группы ученых, в которую входил Илья Воронцов (v11). Видно, что удалось построить модели для 131 ранее не описанных паттернов связывания ТФ человека и 105 ТФ мыши.
[29], рисунок адаптирован
Это позволяет разложить задачу на две части: сначала провести приоритезацию транскрипционных факторов, а уже затем — соответствующих вариантов гена. Благодаря этому удается повысить статистическую мощность анализа.
«Чтобы улучшить предсказания сайтов связывания ТФ, мы также разработали метод переноса экспериментальных данных с одного типа клеток на другой, учитывающий такую информацию, как, например, доступность хроматина. Проблема, которую мы решали, следующая.
В организме есть несколько сотен типов клеток и больше тысячи транскрипционных факторов. Провести по паре экспериментов для каждого фактора и по паре экспериментов для каждого типа клеток — тяжело, но возможно. А вот выполнить сотни тысяч экспериментов, чтобы изучить, как работает каждый фактор в каждой ткани, — малореальная задача.
Чтобы понять, как тип клеток влияет на связывание, нам пришлось применять методы машинного обучения. Метод решения этой задачи мы разработали и применили в рамках международного соревнования ».
Подробнее про соревнование можно почитать в статье на сайте «Биомолекулы»: «Мечту вызывали?» [20].
По итогам работы группа ученых, в которую входил Илья Воронцов, серьезно продвинулась в понимании того, как можно и, что важнее, как нельзя использовать машинное обучение для предсказания эффекта некодирующих вариантов на экспрессию [21]. Но, конечно, остается еще огромный простор для исследований.
Исследователи создали коллекцию участков связывания [22], воспроизводимых между различными экспериментами, и разработали подходы к тому, чтобы делать более точные предсказания для различных типов клеток. Затем ученые опробовали методы приоритезации, основанные на изменении предсказанной аффинности транскрипционных факторов к ДНК, чтобы отобрать регуляторные варианты для нескольких генов для экспериментальной проверки [23–26].
Заключение
Стипендиальная программа по системной, клеточной и молекулярной биологии помогает в развитии таких разных, но вместе с тем взаимно дополняющих друг друга работ, цель которых — проникнуть в тайны жизни. И если в этой статье мы в основном касались тем, которые имеют отношение к клеточной, молекулярной и системной биологии, то в следующей статье рассмотрим стипендию по еще одной захватывающей и, безусловно, весьма актуальной в посткоронавирусную эру теме — иммунологии.
Литература
- Bagley M. (2017).What is biology? Live Science;
- 12 методов в картинках: микроскопия;
- 12 методов в картинках: клеточные технологии;
- W. Weaver. (1970). Molecular Biology: Origin of the Term. Science. 170, 581-582;
- Молекулярная биология;
- 12 методов в картинках: генная инженерия. Часть II: инструменты и техники;
- Я б в биоинформатики пошёл, пусть меня научат!;
- Стипендия по системной биологии: инициатива, которая вдохновляет;
- David J. Newman, Gordon M. Cragg. (2012). Natural Products As Sources of New Drugs over the 30 Years from 1981 to 2010. J. Nat. Prod.. 75, 311-335;
- Победитель бактерий;
- В каждой бочке затычка: как тетраценомицин X затыкает бактериальные и эукариотические рибосомы;
- Антибиотики, структура генома и CRISPR: чем занимается в Сколтехе лаборатория Северинова;
- Paul G. Arnison, Mervyn J. Bibb, Gabriele Bierbaum, Albert A. Bowers, Tim S. Bugni, et. al.. (2013). Ribosomally synthesized and post-translationally modified peptide natural products: overview and recommendations for a universal nomenclature. Nat. Prod. Rep.. 30, 108-160;
- Hosein Mohimani, Wei-Ting Liu, Roland D. Kersten, Bradley S. Moore, Pieter C. Dorrestein, Pavel A. Pevzner. (2014). NRPquest: Coupling Mass Spectrometry and Genome Mining for Nonribosomal Peptide Discovery. J. Nat. Prod.. 77, 1902-1909;
- Anton E. Shikov, Rostislav K. Skitchenko, Alexander V. Predeus, Yury A. Barbitoff. (2020). Phenome-wide functional dissection of pleiotropic effects highlights key molecular pathways for human complex traits. Sci Rep. 10;
- Как перестать бояться и полюбить системную биологию;
- Yury A. Barbitoff, Dmitrii E. Polev, Andrey S. Glotov, Elena A. Serebryakova, Irina V. Shcherbakova, et. al.. (2020). Systematic dissection of biases in whole-exome and whole-genome sequencing reveals major determinants of coding sequence coverage. Sci Rep. 10;
- Сome to the bioinformatics side: Институт биоинформатики в Санкт-Петербурге;
- Летняя школа по биоинформатике — как это было;
- Мечту вызывали?;
- Dmitry D. Penzar, Arsenii O. Zinkevich, Ilya E. Vorontsov, Vasily V. Sitnik, Alexander V. Favorov, et. al.. (2019). What Do Neighbors Tell About You: The Local Context of Cis-Regulatory Modules Complicates Prediction of Regulatory Variants. Front. Genet.. 10;
- Ilya E. Vorontsov, Alla D. Fedorova, Ivan S. Yevshin, Ruslan N. Sharipov, Fedor A. Kolpakov, et. al.. (2018). Genome-wide map of human and mouse transcription factor binding sites aggregated from ChIP-Seq data. BMC Res Notes. 11;
- Anton M. Schwartz, Lidia V. Putlyaeva, Milica Covich, Anna V. Klepikova, Kseniya A. Akulich, et. al.. (2016). Early B-cell factor 1 (EBF1) is critical for transcriptional control of SLAMF1 gene in human B cells. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1859, 1259-1268;
- Putlyaeva L.V., Schwartz A.M., Klepikova A.V., Vorontsov I.E., Kulakovskiy I.V., Kuprash D.V. (2017). The minor variant of the singlenucleotide polymorphism rs3753381 affects the activity of a SLAMF1 enhancer. Acta Naturae. 9, 94–102;
- Anton M. Schwartz, Denis E. Demin, Ilya E. Vorontsov, Artem S. Kasyanov, Lidia V. Putlyaeva, et. al.. (2017). Multiple single nucleotide polymorphisms in the first intron of the IL2RA gene affect transcription factor binding and enhancer activity. Gene. 602, 50-56;
- Marina A. Afanasyeva, Lidia V. Putlyaeva, Denis E. Demin, Ivan V. Kulakovskiy, Ilya E. Vorontsov, et. al.. (2017). The single nucleotide variant rs12722489 determines differential estrogen receptor binding and enhancer properties of an IL2RA intronic region. PLoS ONE. 12, e0172681;
- Ramesh P. Pandey. (2017). Diversifying Natural Products with Promiscuous Glycosyltransferase Enzymes via a Sustainable Microbial Fermentation Approach. Front. Chem.. 5;
- M. Costanzo, B. VanderSluis, E. N. Koch, A. Baryshnikova, C. Pons, et. al.. (2016). A global genetic interaction network maps a wiring diagram of cellular function. Science. 353, aaf1420-aaf1420;
- Ivan V Kulakovskiy, Ilya E Vorontsov, Ivan S Yevshin, Ruslan N Sharipov, Alla D Fedorova, et. al.. (2018). HOCOMOCO: towards a complete collection of transcription factor binding models for human and mouse via large-scale ChIP-Seq analysis. Nucleic Acids Research. 46, D252-D259.