Цифровая ПЦР: измеряя истину
22 июля 2022
Цифровая ПЦР: измеряя истину
- 8606
- 0
- 14
Цифровая ПЦР позволяет детектировать даже единичные события
иллюстрация Елены Беловой
-
Авторы
-
Редакторы
-
Иллюстратор
Метод полимеразной цепной реакции (ПЦР) изменил биологию и медицину 80-х годов XX века, позволив быстро и точно диагностировать наследственные заболевания и инфекции, определять патогенов и свободно манипулировать генами. Интереснейшая его модификация — цифровая ПЦР — быстро завоевывает пространство лабораторной диагностики XXI века, когда требуется абсолютное количественное определение ДНК. В каком случае это нужно и каким образом может быть реализовано на современном оборудовании — читайте в нашей статье спецпроекта «Ультрасовременные методы».
Ультрасовременные методы

Партнер публикации — компания QIAGEN — один из мировых лидеров в технологиях анализа биологических образцов в области наук о жизни, молекулярной диагностики, геномики и онкологии, криминалистики и женского здоровья. Компания предлагает диагностическим и научным лабораториям наборы реагентов, оборудование и программное обеспечение, позволяющее проводить все этапы исследований «от образца к результату» — от гомогенизации, стабилизации и экстракции образцов до их молекулярно-генетического анализа, биоинформатической обработки и интерпретации данных. QIAGEN осуществляет не только поставку своей продукции в лаборатории, но и оказывает сервисное обслуживание, техническую поддержку и обучение пользователей, а также имеет собственный тренинговый центр.
Методы, которыми оперирует современная наука, постоянно совершенствуются. Некоторые области развиваются настолько стремительно, что порой даже специалистам сложно уследить за новейшими приборами и модификациями методик. Так происходит, например, в геномике. Другие же области (такие как гистология и связанные с ней световая и флуоресцентная микроскопия) развиваются куда медленнее, но всё равно расширяются и обзаводятся более высокопроизводительными методами. В статьях нового спецпроекта (он продолжает наш хит — «12 биологических методов в картинках») мы хотим рассказать, какие методы помогают ученым совершать новые открытия сегодня, и чего нам ждать в будущем.
Цифровую ПЦР (цПЦР) для количественного определения мишени разрабатывали независимо несколько раз: в 1990 и 1991 годах она была упомянута в работе по изучению ВИЧ как «ПЦР с предельным разведением» (англ. limiting dilution PCR) [1], в 1992 году детально описана в публикации Алекса Морли [2], а в 1999 году названа цифровой ПЦР (англ. digital PCR, dPCR) в статье о мутациях в гене ras , [3], [4]. Однако внедрения в рутинную лабораторную практику не случилось ни тогда, ни потом: в 1996 году описали количественную ПЦР в реальном времени (кПЦР), требующую намного меньших трудозатрат, и цПЦР отошла на второй план. В настоящее время цПЦР переживает ренессанс. Благодаря появлению на рынке новых инструментов и химических реактивов, метод стал намного более простым, практичным и привлекательным.
О гене ras, белках Ras и их ингибиторах мы писали в статье «Ингибиторы Ras: в поисках Грааля таргетной терапии» спецпроекта «Современные лекарства» [5].
До 2007 года количество публикаций о цПЦР оставалось небольшим (всего несколько штук в год), но пять лет назад всё изменилось, и сейчас наблюдается экспоненциальный рост числа статей об исследованиях с использованием метода цПЦР. Причем если сначала эти статьи публиковались преимущественно в изданиях по инженерным системам и микрофлюидике, то теперь они появляются в биологических и медицинских журналах.
Классическая ПЦР [6] может использоваться либо для изучения свойств молекулы-мишени, либо для определения ее относительного количества; цПЦР используется для этих же задач, с той лишь разницей, что в цПЦР образец сначала разделяется до отдельных молекул методом предельного разведения , и лишь после этого проводится ПЦР-амплификация. Получается цифровой сигнал «всё или ничего» (по принципу двоичного кода). Но об этом давайте поподробнее.
Предельное разведение — это метод, основанный на использовании конечной точки по принципу «всё или ничего» и на предпосылке, что одна или несколько ДНК-мишеней в реакционной смеси приводят к положительной конечной точке.
ПЦР, количественная ПЦР, цифровая ПЦР: кто кого?
ПЦР, или полимеразная цепная реакция
ПЦР — это метод, при котором происходит амплификация ДНК-матрицы, то есть синтез нескольких миллионов копий определенного фрагмента ДНК из малого количества исходного материала , [7].
Подробнейшим образом мы писали об этом в одноименной статье «Полимеразная цепная реакция» [6] классического спецпроекта «12 биологических методов в картинках», куда и отправляем первым делом читателей, которые хотят разобраться во всём досконально.
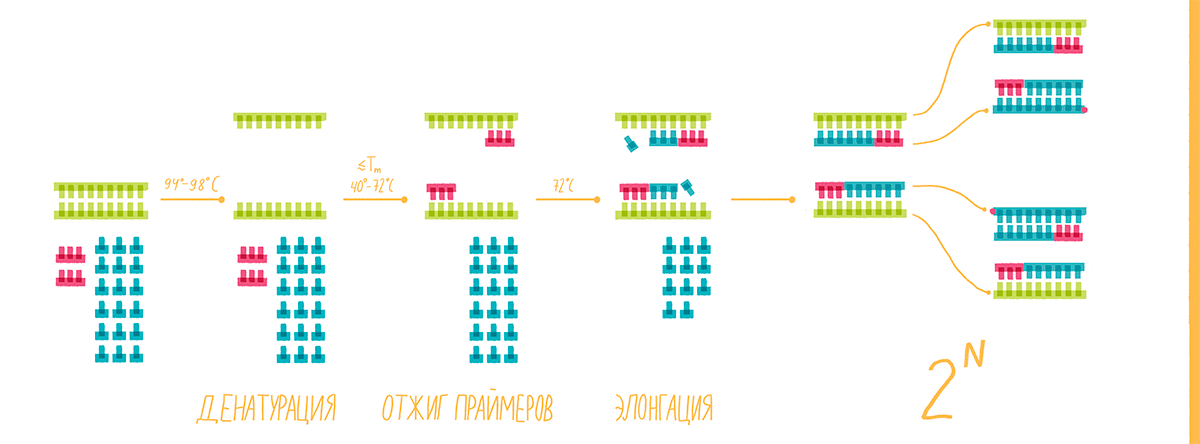
Специфичность ПЦР зависит от гибридизации с матрицей коротких последовательностей — праймеров, — которые комплементарны участкам, фланкирующим (ограничивающим) последовательность-мишень, а чувствительность — от природы фермента ДНК-полимеразы. ПЦР обычно состоит из серии температурных циклов, повторяющихся 20–40 раз. Каждый цикл — это последовательные денатурация двухцепочечной ДНК, гибридизация праймеров с ДНК-матрицей и элонгация (удлинение этих праймеров ДНК-полимеразой) (рис. 1а). Таким образом в каждом цикле количество молекул ДНК-мишени удваивается (так называемая экспоненциальная амплификация), и теоретически после n циклов может быть получено 2n копий. На практике амплификация насыщается и выходит на плато по мере того, как реагенты ПЦР истощаются, а накопленные продукты ПЦР «отжигаются» сами на себя, предотвращая дальнейшую амплификацию. Наличие и примерное количество («много—мало») ПЦР-продукта можно оценить при помощи гель-электрофореза.
Эффективность ПЦР зависит от многих факторов. Это и производительность анализа, зависящая от последовательностей и структур праймеров и матриц; и чистота матрицы (например, отсутствие примесей, ингибирующих ПЦР); и концентрация используемых реактивов. В целом, любой из реагентов ПЦР может ограничивать скорость и производительность анализа [8].
Количественная ПЦР
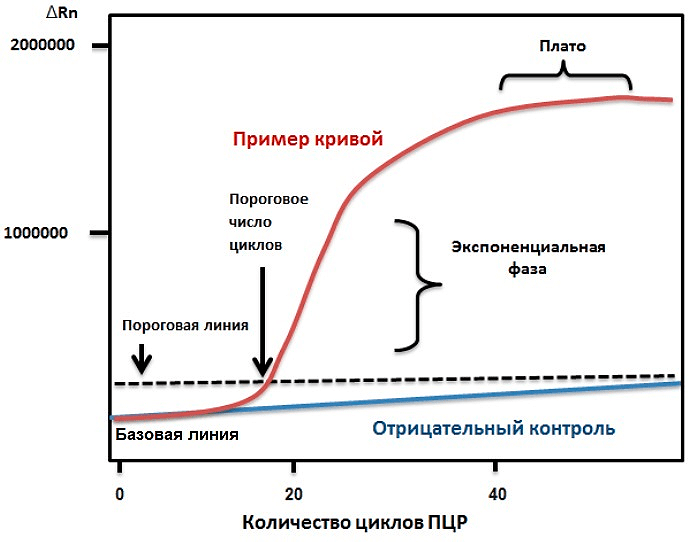
Количественная ПЦР (кПЦР, также называемая ПЦР в реальном времени) может использоваться для измерения количества продукта после каждого раунда амплификации с помощью считывания флуоресценции [9]. ПЦР в реальном времени позволяет проводить относительную количественную оценку мишени по отношению к калибратору (образец с точно известной концентрацией, относительно которого рассчитывается количество ДНК-мишени в опытном образце). Соответственно, при наличии стандартной калибровочной кривой (и, что важно, эквивалентной эффективности амплификации образца и стандарта!) метод является количественным (рис. 2) [10].
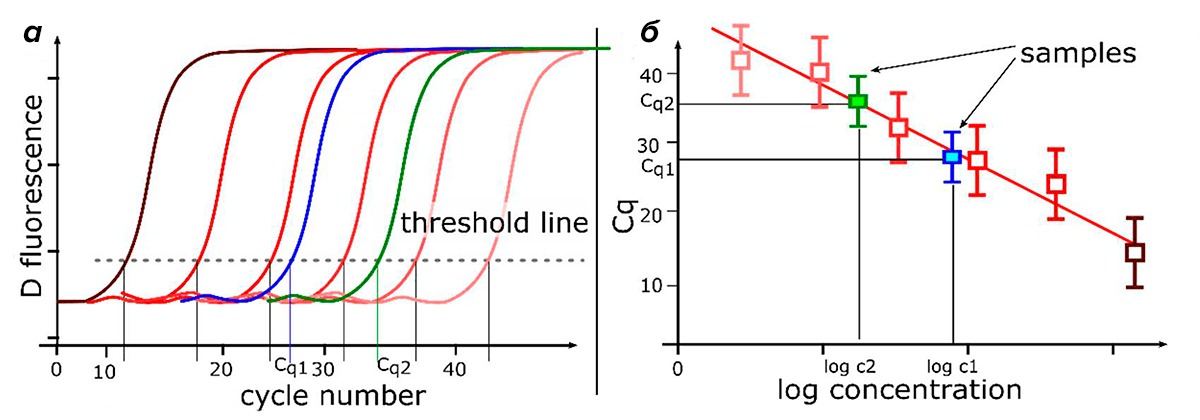
Рисунок 2. Анализ количественной ПЦР в реальном времени с использованием стандартной кривой. а — Кривые амплификации для шеститочечной серии десятикратных разведений матрицы с известными концентрациями (стандарт — образец с известной концентрацией, используемый для построения стандартной кривой) более пяти порядков (например, геномная ДНК, ампликон ПЦР, линеаризованная плазмида). Определяют значение Cq каждого серийно разбавленного стандарта. Кривые от коричневого до розового цветов — стандартные кривые различных разведений. Синяя и зеленые кривые — кривые амплификации образцов. б — Стандартная кривая создается путем нанесения значений Cq, полученных из кривых амплификации серии разведений, в зависимости от логарифма стандартного количества. По ней можно определить количество исследуемого образца, нанеся его значение Cq на график. Наклон стандартной кривой измеряет эффективность амплификации количественной ПЦР. Наклон −3,32 (для стандартной кривой, полученной из серии десятикратных разведений) указывает на 100%-эффективность амплификации (количество продукта ПЦР удваивается во время каждого цикла).
Главным недостатком кПЦР является относительность информации об исследуемом образце. Скажем, если образец, обработанный лекарственным веществом, содержит в два раза больше целевой мРНК, чем контрольный образец, то, чтобы определить абсолютное количество ее молекул, исследователи должны сравнить значения Ct (cycle threshold) образца со стандартной кривой [2], [4], [11].
Ct (cycle threshold) — это количество циклов создания дополнительных копий РНК, начиная с которого результат конкретного теста считается положительным. — Ред.
Цифровая ПЦР
Цифровая ПЦР (цПЦР) позволяет проводить определение абсолютного количества целевых нуклеиновых кислот, присутствующих в образце. В цПЦР образец сначала разводят и разделяют на множество независимых реакций ПЦР, так что каждая ячейка реакционной смеси содержит либо одну ДНК-мишень (или всего несколько), либо не содержит их вовсе (рис. 3). Далее проводят ПЦР с флуоресцентным красителем или гидролизными зондами и считывают флуоресцентный сигнал. Доля положительных по амплификации ячеек используют для количественного определения концентрации целевой последовательности [12], [13]. Интересно, что разделение образцов эффективно концентрирует ДНК-мишени в ячейках. Этот эффект концентрации снижает конкуренцию матриц и, таким образом, позволяет обнаруживать редкие мутации на фоне последовательностей дикого типа. Кроме того, это также обеспечивает более высокую толерантность к ингибиторам, присутствующим в образце, так как их количество также многократно разводится.
Короткая одноцепочечная последовательность ДНК, имеющая флуорофор и «гаситель» на 3′- и 5′-концах, комплементарна внутреннему участку ДНК-мишени. При гибридизации с ДНК-мишенью зонд не флуоресцирует, при прохождении полимеразы во время элонгации зонд гидролизуется за счет 5′–3′ экзонуклеазной активности Taq-полимеразы, метки оказываются в растворе сильно разнесенными, и флуорофор начинает работать.
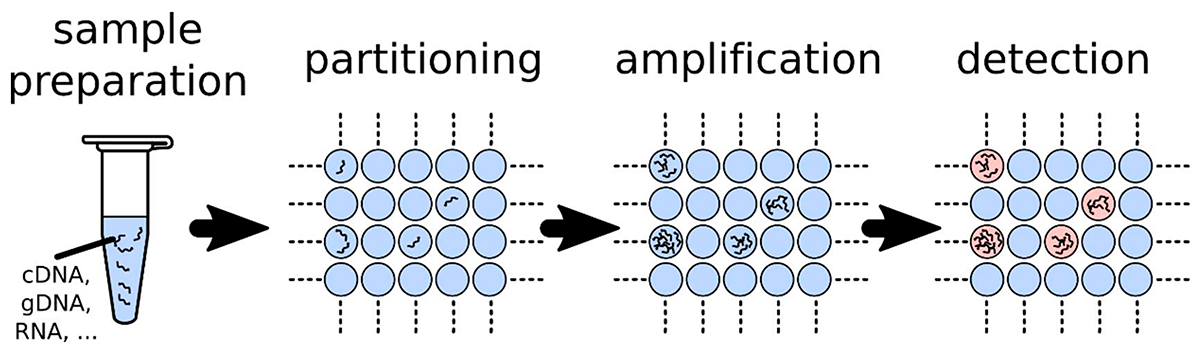
Рисунок 3. Принципы цифровой ПЦР. Образец разведен и наносится во множество независимых ячеек (или капель). Каждая ячейка действует как отдельный микрореактор ПЦР, и ячейки, содержащие амплифицированные ДНК-мишени, обнаруживаются по флуоресценции. Распределение ДНК-мишеней в ячейках можно аппроксимировать распределением Пуассона. Отношение положительных сигналов (наличие флуоресценции) к общему количеству позволяет определить концентрацию мишени в образце.
Ключевое различие между цПЦР и кПЦР заключается в их стратегии измерения количества ДНК-мишени (табл. 1). При кПЦР накопление продукта отслеживается на протяжении всего процесса амплификации, а количественная оценка основана на анализе флуоресцентного сигнала в экспоненциальной фазе ПЦР. Напротив, при цПЦР анализируются сигналы флуоресценции конечного продукта по окончании ПЦР и оценивается количество положительных ячеек из общего количества для обратного расчета исходной концентрации ДНК-мишени (рис. 4). цПЦР сводит количественную оценку к положительным или отрицательным результатам, таким образом, преобразуя аналоговый сигнал в серию двоичных (или цифровых) сигналов. В отличие от кПЦР, цПЦР не использует калибровочные кривые для количественного определения образца. Следовательно, она позволяет избежать искажения результатов, связанных с изменением эффективности реакции [10]. Количественная оценка с помощью цПЦР основана на биномиальной статистике, которая математически определяет присущую ей точность и показатели производительности.
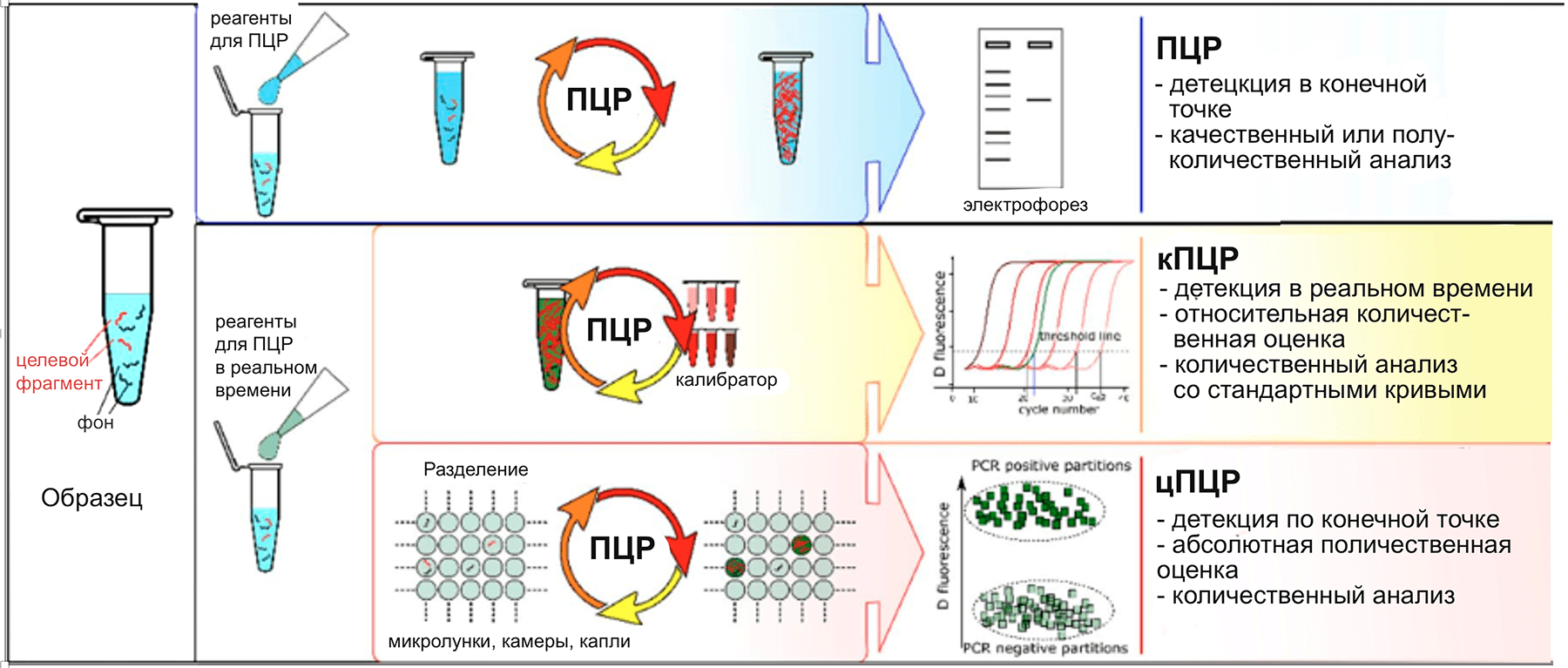
Рисунок 4. Сравнение методов на основе ПЦР
| кПЦР | цПЦР |
|---|---|
| Различия | |
| Требуется стандартная кривая | Стандартная кривая не требуется |
| Нет разделения образца | Образец разделен на множество ячеек |
| Данные собираются в режиме реального времени | Сбор данных в конечной точке после окончания реакции |
| Относительная количественная оценка | Абсолютная количественная оценка |
| Сходства | |
| Совместимость методов пробоподготовки | |
| Компоненты реакции амплификации и их концентрации одинаковые — PCR Master Mix, флуоресцентные зонды, праймеры, ДНК-матрица | |
| Служат для определения количеств мишени, присутствующей в образце | |
| Одинаковые начальные объемы образца | |
| Оба работают либо с гидролизными зондами (TaqMan Probes, Molecular Beacons, Hybridization Probes, Scorpions Probes etc.), либо с ДНК-связывающим красителем (SYBR Green I, YO-PRO-1, SYBR® Gold, SYTO, BEBO, BOXTO и EvaGreen) | |
| Широкий динамический диапазон | |
| Возможность мультиплексирования | |
Анализ результатов цПЦР: вспоминаем статистику
Огромное преимущество цПЦР — обсчет результатов с помощью статистических методов, которые позволяют сделать вывод как об исходной концентрации матрицы, так и о точности количественного определения.
Интуитивно понятно, что достоверность оценки исходной концентрации ДНК-мишени зависит от количества пустых ячеек. В крайних случаях, то есть когда большинство ячеек либо пусты, либо заполнены, достоверность расчетной концентрации очень низкая, поскольку паттерн «пустой/полный» не очень информативен.
Можно предположить, что существует ожидаемое значение, для которого начальную концентрацию образца можно оценить с наибольшей достоверностью. В случаях 10 000 и более ячеек максимальная достоверность достигается при ожидаемом значении λ около 1,6, что соответствует 20% ячеек (рис. 6). Точность оценки λ увеличивается с количеством ячеек, а оптимальная точность (при λ = 1,6) масштабируется как обратный квадратный корень из числа ячеек.
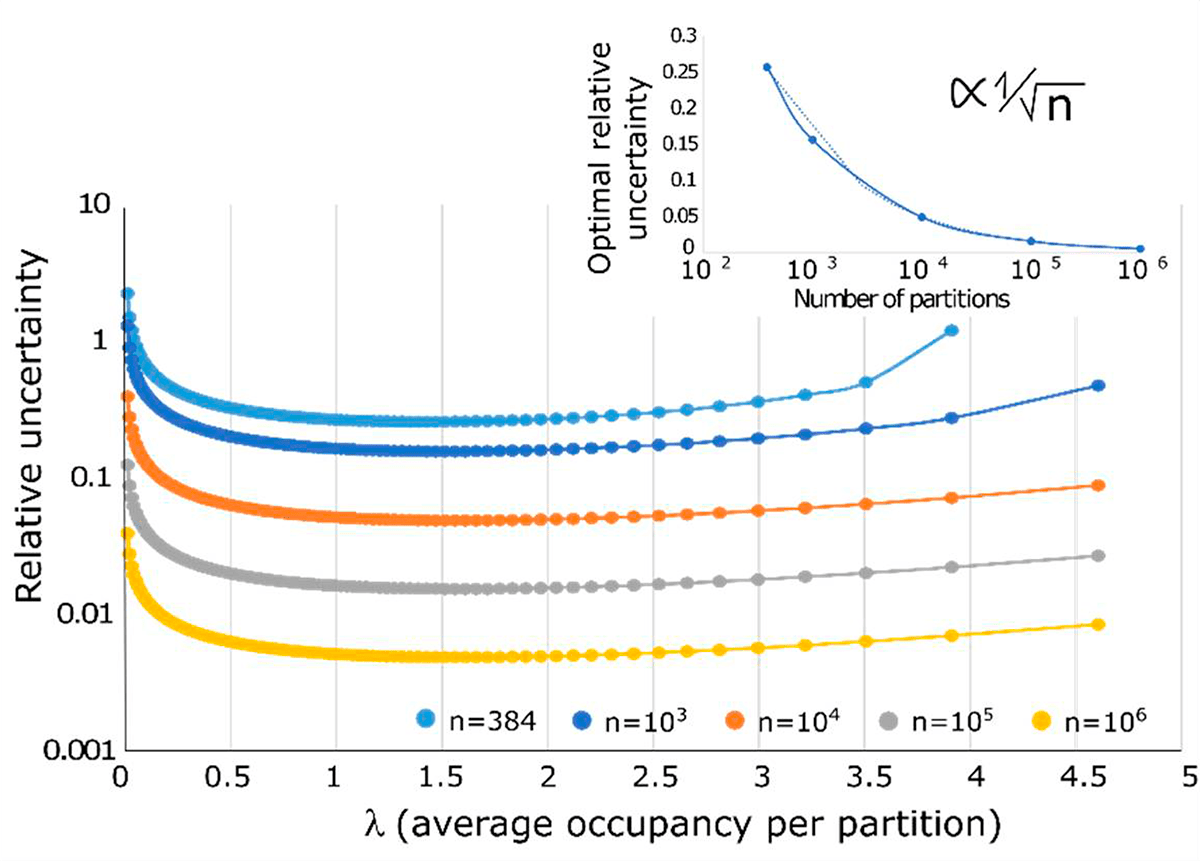
Рисунок 6. Точность количественной оценки цПЦР. Точность цПЦР неравномерна и зависит от средней занятости ДНК-мишени на ячейку, а также от количества ячеек. На рисунке показано, что эволюция относительной неопределенности (взятая при λ ≈ 1,6) уменьшается как обратный квадратный корень из числа ячеек.
У цПЦР есть и присущие методу статистические ограничения. Так, точность цПЦР ограничена неопределенностью измерения из-за:
- технической погрешности отбора образцов, эффект которой особенно заметен при низких концентрациях ДНК-мишени и может быть сведен к минимуму только при использовании технических повторов;
- ее статистического характера — в этом случае неопределенность может быть уменьшена за счет увеличения числа разбиений [14].
Собственная точность цПЦР не является постоянной во всем динамическом диапазоне и может быть довольно низкой в крайних точках (тот случай, когда большинство разбиений либо положительные, либо отрицательные). Еще одно техническое ограничение цПЦР связано с изменением объема ячейки, что может давать негативный эффект при высокой средней занятости λ и доминировать в неопределенности количественной оценки при очень большом количестве ячеек. Соответственно, очень важно использовать оптимальное количество ячеек одинакового объема для анализа [15].
Эффективность цПЦР
Чувствительность или нижний предел обнаружения ДНК-мишени для цПЦР — это обнаружение одной молекулы в одной ячейке. Следовательно, минимальная концентрация, которую можно детектировать, зависит от общего объема реакции (или от суммарного количества ячеек и их объема). Объем каждой ячейки в цПЦР лежит в диапазоне пл–нл (пиколитры–нанолитры). Количество ячеек обычно на практике ограничено, и именно это определяет предел чувствительности обнаружения цПЦР. Напротив, объем реакции кПЦР обычно намного больше, и его также можно легко отрегулировать для достижения более высокой чувствительности.
Наибольшая концентрация молекул напрямую зависит от объема ячейки, т.е. ячейки с меньшим объемом соответствуют более высоким концентрациям молекул при заданном λ. Интересно, что наибольшее количество обнаруженных последовательностей-мишеней может быть намного больше, чем количество ячеек. Это значение оценивается путем решения λ для заданной точности и количества ячеек в ситуации высокой занятости ячейки. Например, при точности 12,6% наибольшее количество обнаруженных последовательностей-мишеней может быть в 5 раз больше, чем 20 000 сгенерированных капель [14], или в 11 раз больше, чем 106 созданных ячеек [16].
Исходя из этих соображений, большой динамический диапазон обнаружения создает противоположные ограничения на объем ячеек; при этом большие объемы ячеек улучшают нижний предел обнаружения, а меньшие объемы ячеек улучшают верхний предел обнаружения.
Эту загадку можно решить, используя конструкции цПЦР с многообъемными ячейками, где ряд ячеек большого объема обеспечивает высокую чувствительность, в то время как ряд ячеек малого объема обеспечивает высокий предел обнаружения, а несколько рядов ячеек среднего объема обеспечивают высокую точность [17], [18]. С практической точки зрения этот подход уменьшает общее количество ячеек, необходимых для достижения заданного динамического диапазона, и, следовательно, общую площадь устройств.
Надежность цПЦР: ложноотрицательные и ложноположительные сигналы
Чувствительность метода сильно зависит от частоты ложноположительных и ложноотрицательных событий. Хотя цПЦР основана на цифровом анализе, обнаруженный сигнал изначально аналоговый, и при интерпретации результатов необходимо вводить порог, чтобы отделить истинный сигнал от фонового (о методе статистической пороговой обработки можно прочитать в [19]). Ложноположительные результаты могут возникать из-за плохого дизайна эксперимента или из-за обнаружения ложной амплификации при большом количестве циклов ПЦР, а также в результате контаминаций во время постановки эксперимента [14].
Ложноотрицательные результаты (или выбросы) менее вероятны, чем ложноположительные. Однако они появляются из-за того, что:
- Взаимодействие реагентов с поверхностью повышает вероятность ингибирования ПЦР, так как цПЦР сопровождается увеличением отношения суммарной поверхности ячеек к ее суммарному объему [20].
- Амплификация одной молекулы часто менее эффективна, чем амплификация большего их числа [21].
- Эффективность амплификации сильно зависит от источника ДНК (т.е. геномной или плазмидной, фрагментированной или длинной ДНК) [13], [21], [22] и может снижаться при нагревании молекул ДНК [23].
На результаты цПЦР, конечно, влияют и предыдущие стадии эксперимента (например, выделение ДНК и предварительная амплификация [24]). Скажем, у исследователя может возникнуть соблазн провести предварительную амплификацию образца с низкой исходной концентрацией, чтобы достичь оптимального значения λ, равного 1,6. Однако дисперсия, связанная с реакцией предварительного усиления, не является систематической и не может быть исправлена. Поэтому прямой количественный анализ даже мишени с низкой концентрацией всё же предпочтительнее [13], [25].
Приборы для цПЦР
За свою более чем 30-летнюю историю цПЦР давно ушла от использования традиционных 384-луночных планшетов, стала более высокотехнологичной и удобной в использовании. Сейчас на рынке представлено несколько платформ от разных производителей для проведения цПЦР, при этом общая концепция цПЦР остается неизменной: исходная ДНК разбавляется для создания нано- или пикомасштабных (количественных) реакций, содержащих 1 или 0 копий матрицы.
Standard BioTools Inc. (Fluidigm Corporation)
Одним из первых коммерчески доступных инструментов для цПЦР стала система BioMark™ HD dPCR (Standard BioTools Inc.), основанная на технологии микрофлюидных чипов. Чипы можно приобрести в различных форматах, включая 12-камерные или 48-камерные массивы. В массиве из 12 камер образцы разделены на 765 нанолитровых реакций, что дает 9180 реакций на чип (в 48-камерном массиве — 36 960 реакций на чип). Образцы загружаются во входное отверстие каждой камеры, и нанолитровые реакционные смеси распределяются по ней при помощи клапанов и насосов с регулируемым давлением. Разделение и смешивание образцов, а также реакции термоциклирования выполняются на чипе. После амплификации флуоресценция детектируется с помощью системы BioMark [26]. Встроенная обработка данных позволяет выполнять меньше ручных манипуляций, тем самым снижая вероятность внесения ошибок и сохраняя при этом более простую и удобную для пользователя систему.
Система цПЦР компании Standard BioTools позволяет мультиплексировать реакции и детектировать четыре разных ДНК-мишени на образец. Флуоресцентный сигнал чипа снимается до и после каждого цикла термоциклирования. Это позволяет вычесть любой фон до термоциклирования из окончательного флуоресцентного изображения, облегчая точный подсчет положительных компартментов. Еще одной особенностью системы является возможность количественного определения ДНК-мишени, которая разделена на каждую камеру, с использованием конкретных для каждой камеры графиков амплификации в реальном времени. Соответственно, BioMark™ HD можно использовать и как инструмент для проведения кПЦР [25], [27].
Thermo Fisher Scientific (Life Technologies™)
В основе работы систем OpenArray® и QuantStudio® 12K Flex dPCR (Thermo Fisher Scientific) лежит микрофлюидная технология. Приборы различаются по производительности: OpenArray вмещает до трех планшетов цПЦР, а QuantStudio 12K Flex dPCR — до четырех. Каждый 384-луночный планшет имеет 48 массивов с 64 «сквозными отверстиями». В результате получаются 3072 разделенные реакции на планшет. Нанолитровые реакции распределяются по «сквозным отверстиям» с помощью автоматизированной системы дозирования и стабилизируются там за счет гидрофобных и гидрофильных взаимодействий между каплей и покрытием планшета. Как и в BioMark™ HD, образцы можно мультиплексировать: правда, в данном случае одновременно детектируются лишь две ДНК-мишени. Данный прибор можно использовать и в формате количественной ПЦР [28].
Bio-Rad (RainDance Technologies)
Прибор RainDrop™ (изначально созданный компанией RainDance Technologies) — еще одна высокочувствительная платформа для цПЦР. Увеличение чувствительности и количественной мощности в нем связано с более мелкими реакциями объемом в пиколитры и увеличенным количеством разделенных капель: около 10 миллионов на образец. RainDrop основан на микрофлюидной технологии эмульгирования капель. Чипы предназначены для нанесения восьми образцов, при этом каждый из них разделяется на 10 миллионов реакций. В результате на чип приходится 80 миллионов разделенных реакций. Благодаря увеличенному количеству реакционных ячеек на такой чип может быть нанесен образец с большим количеством ДНК, что делает эту платформу идеальной для выявления чрезвычайно редких мутаций. Результаты экспериментов показывают, что система RainDrop™ может использоваться для количественного определения 1 из 200 000 мутантов и имеет нижний предел обнаружения 1 из 1 000 000 [29].
В дополнение к повышенной чувствительности, на платформе RainDrop™ можно мультиплексировать пять ДНК-мишеней на образец, просто используя флуоресцентные зонды с красной и зеленой маркировкой. Изменение количества флуоресцентного зонда для каждой мишени создает уникальную интенсивность цвета, которая соответствует мутационно-специфичному зонду с двойной меткой, а интенсивность сигнала напрямую связана с концентрацией зонда, используемого в анализе. Метод разбавления флуоресцентных зондов для генерации оптического кода может не ограничиваться системой пятикратного мультиплексирования и может использоваться для создания системы десятикратного мультиплексирования, что делает эту систему одной из самых мощных доступных систем мультиплексирования цПЦР [30], [31].
Хотя RainDrop™ является одной из самых экономичных платформ с точки зрения цены анализа одного образца, но создана она так, что ее нельзя использовать для проведения количественной ПЦР. Кроме того, настройка этого прибора более трудоемка и, следовательно, сопровождается бóльшим количеством ошибок [31].
В 2017 году компания Bio-Rad Laboratories купила RainDance Technologies, сейчас прибор не выпускается.
Bio-Rad
Bio-Rad QX100™ Droplet Digital™ — единственная платформа, в которой не используются микрожидкостные чипы ни на одном этапе. В основе разделения образцов в данном случае лежит технология эмульгирования масла в стандартном формате 96-луночного планшета. В картридж можно одновременно загрузить восемь образцов. Каждый образец помещается рядом с лункой, содержащей масло, и вместе они подвергаются капельному эмульгированию с использованием генератора капель на основе вакуума. Таким образом, образец разбивается на 20 000 реакций и переносится в лунку стандартного 96-луночного планшета. Полностью загруженный образцами планшет содержит 1 920 000 капель. Далее образцы подвергают стандартной ПЦР, а конечные флуоресцентные сигналы считывают при помощи устройства для считывания капель на основе метода проточной цитометрии [25], [32].
Большое количество реакций, созданных с помощью этой платформы, повышает точность цПЦР, а также дает возможность загружать большее количество матричной ДНК. Прибор не совместим с приложениями количественной ПЦР и может использоваться для мультиплексирования только двух мишеней на образец.
В таблице 2 приведена сравнительная характеристика упоминавшихся платформ для проведения цПЦР.
| Метод | кПЦР | цПЦР | |||||
|---|---|---|---|---|---|---|---|
| Прибор | — | QuantStudio 3D (Thermo Fisher Scientific, Life Technologies™) | BioMark™ HD dPCR (Standard BioTools Inc., Fluidigm Corporation) | Bio-Rad QX200 (Bio-Rad) | Naica® system 6-color dPCR (Stilla Technologies) | RainDrop (Bio-Rad, RainDance Technologies) | QIAcuity One/Four/Eight (QIAGEN) |
| Метод разделения | — | микрочипы | микрочипы | микрочипы | капли | капли | нанопланшеты |
| Ячеек на образец; образцов на чип | — | 20 000 на 1 образец | 770 на 48 образцов | 15 000 — 20 000 на 8 образцов | 30 000 на 4 образца | До 10 миллионов на 1 образец | 8500 или 26 000 (в зависимости от выбранного нанопланшета) |
| Объем ячейки | — | 0,72 нл | 0,85 нл | 0,837 нл | 0,43 нл | 4,39 пл | 0,34 нл |
| Неопределенность объема | — | N/A | 0,7% | 0,8% | 8% | 2,90% | N/A |
| Динамический диапазон | >9 log | 5 log | 2 log | 5 log | 5 log | 6 log | 5 log |
| Объем образца | 0,03–200 мкл | 14,5 мкл | 4 мкл | 20 мкл | 20 мкл | 20 мкл | 40 или 12 мкл |
| Точность числа копий | 1–2 копии, в 1,5–2 раза | 7–8 копий, 1,2-кратный | 4–5 копий, 1,2-кратный | 5–6 копий, 1,2-кратный | 5–6 копий, 1,2-кратный | >6, возможно, в 1,1 раза | 1–2 копии |
| Точность (95% доверительный интервал) | Варьирует | ±10% | N/A | ±10% | ±10% | ±10% | ±10% |
| Мультиплексирование | ≤5 каналов | 2 канала | ≤5 каналов | 2 канала | 3 канала | 2 канала | 2 или 5; 5; 5 |
| Обнаружение редких мутаций | Варьирует | 1/1000 | N/A | 1 / 200 000 | 1 / 200 000 | 1 / 200 000 | N/A |
| Обнаружение редких последовательностей | Варьирует | N/A | N/A | 1 / 1 000 000 | 1 / 1 000 000 | 1/1,000,000 | N/A |
| Пропускная способность | Тысячи образцов в день | 24 образца за ~4 часа | 48 образцов за ~4 часа | 96 образцов за ~5 часов | 12 образцов за ~2 часа | 8 образцов за ~4 часа | 96 луночный планшет — 384, 672, 1248 образцов за 2 часа
24 луночный планшет — 96, 168, 312 образцов за 2 часа |
| Стоимость инструмента + стоимость реакции | $25–50K + $2/образец | $45K + $10/образец | $200–250K + $400/чип | $80–100K + $5/образец | $80–100K + $7/образец | ~$120K + $10/образец | $80–100K + $3–5/образец |
Постановка реакции цПЦР на примере QIAcuity Digital PCR System
А теперь давайте представим, что мы проводим реакцию цПЦР для интересующего нас образца. Что же нам потребуется?
Во-первых, инструменты. Как было рассказано в предыдущей главе, машины для цифровой ПЦР с каплями производят несколько компаний; базовый экспериментальный план почти одинаков для любой из них. Цифровая ПЦР-система на основе капель состоит из двух машин: одна генерирует капли, а другая считает количество положительных сигналов после амплификации. Также понадобится термоциклер для проведения самой ПЦР — в принципе, это может быть любой прибор подобного рода, в идеале, количественный. Таким образом, можно отслеживать реакцию в режиме реального времени, и если она по каким-то причинам не проходит, остановить ее и избежать дальнейшей ненужной работы по подсчету сигналов.
Во-вторых, реагенты. За исключением масла, образующего капли, все реагенты в основном такие же, как и в количественной ПЦР. То есть можно выбрать анализ на основе зонда или с использованием флуоресцентного красителя. Если нужно мультиплексировать образцы, стоит использовать зонды с разными красителями. Можно прибегнуть к хитрости и мультиплексировать образцы без зонда, сыграв на длине амплифицируемого фрагмента — более длинные ампликоны будут связывать больше флуоресцентного красителя и сильнее флуоресцировать.
При использовании больших матриц ДНК (геномной ДНК из свежих тканей/клеток) сначала стóит расщепить ДНК эндонуклеазами, так как геномная ДНК очень вязкая, и это может поставить под угрозу образование капель воды в масле (только нужно убедиться, что ферменты не разрезают область амплификации!). Геномная ДНК, выделенная из образцов парафиновых блоков (FFPE), не нуждается в этом этапе предварительной обработки, поскольку она уже фрагментирована из-за фиксации ткани.
Чтобы анализ был информативен, необходимо правильно разбавить образец. Для этого стоит заранее проверить динамический диапазон машины (минимальное и максимальное количество детектируемых копий шаблона в образце). Для справки: в 400 нг ДНК человека содержится примерно 120 000 копий гаплоидного генома. Таким образом, для оценки количества копий на образец можно использовать формулу:
m(g) = n × 1,096×10–21 г/пн,
где n — размер генома в парах оснований, г/пн — масса одной пары оснований.
Три, два, один — начали!
Первым шагом в эксперименте с капельной цифровой ПЦР является смешивание реакционной смеси с маслом, загрузка ее в генератор капель и ожидание их появления под действием давления. В некоторых системах после этого может потребоваться перенос капель в планшет для ПЦР. Осторожнее с пипетированием: капли на этом этапе нестабильны.
Второй этап — термоциклирование, проводимое в условиях, указанных для интересующего гена. Поскольку капли неподвижны, скорость нормальной термической диффузии в воде снижается, а это означает, что капли могут нагреваться не одновременно. Чтобы преодолеть это, нужно установить скорость линейного изменения на 2,5 °С/с, чтобы позволить всем каплям достичь правильной температуры.
Финальный аккорд — детекция капель в считывателе. В нем капли сначала проходят контроль качества: те, что не соответствуют критериям размера и формы, исключаются из измерения. Каплям, содержащим ДНК-мишень и поэтому имеющим флуоресценцию, превышающую порог, присваивается значение 1; без матрицы (а значит, имеющим флуоресценцию ниже порогового значения) — 0. Программное обеспечение подсчитывает количество положительных (1) и отрицательных (0) капель и вычисляет точное количество копий ДНК-мишени. Общее количество капель, включенное в итоговое измерение, в конечном счете не важно, потому что результат рассчитывается по доле отрицательных капель.
Детекция результата может занять несколько часов. Полезно знать, что капли очень стабильны при ПЦР. Вы можете хранить их несколько дней в холодильнике, если у вас недостаточно времени, чтобы прочитать их в тот же день.
И самое главное: прежде чем начать, нужно четко определить, что искать.
Как бы великолепно ни звучала технология цифровой ПЦР, она не подходит для каждого ПЦР-эксперимента. Лучшее применение технологии — задачи из серии «найти иголку в стоге сена»: поиск редких аллельных мутаций, небольших кратных изменений в экспрессии генов (например, двукратных, которые не может обнаружить кПЦР) или небольшой разницы в количестве копий (скажем, между 10× и 11×). Эксперименты с высокой пропускной способностью и анализ больших изменений в экспрессии успешно проводятся с помощью традиционной ПЦР или ПЦР в реальном времени. В случаях же, когда точность и чувствительность имеют первостепенное значение, использование цифровой ПЦР оправдано. Надо помнить, что чувствительность повышается с количеством капель, поэтому при выборе системы важно соотнести параметр количества капель с тем, насколько чувствительным должно быть обнаружение.
Задачи для цПЦР
За последние годы цПЦР проникла чуть ли не во все сферы, где необходимо провести генетический анализ образца: она используется в науке, прикладной медицине, сельском и пищевом хозяйствах. Дальше мы расскажем о нескольких типичных задачах, которые можно элегантно решить при помощи цПЦР.
Применение цПЦР в научных исследованиях
Анализ экспрессии генов, единичных клеток и обнаружение редких последовательностей
цПЦР не требует создания стандартных кривых или нормализации по генам домашнего хозяйства, что делает ее идеальной для обнаружения и количественного определения слабо экспрессируемых генов, поиска редких последовательностей и анализа материала, взятого из отдельных клеток. Кроме того, цПЦР достаточно чувствительна, чтобы надежно обнаруживать едва уловимые изменения в экспрессии генов (до 2-кратных различий).
Если добавить предварительный этап обратной транскрипции (ОТ), цПЦР может количественно определять уровни не только ДНК, но и РНК практически со всеми преимуществами цифрового разделения. Особенно это интересно при обнаружении циркулирующих микроРНК (мкРНК), которые, как известно, могут быть биомаркерами рака, эндокринной дисфункции и других заболеваний. Показана возможность такого варианта цПЦР, если «вручную» выполнить обратную транскрипцию перед цПЦР в системе Bio-Rad QX100 [33]. ОТ-цПЦР имела на 37–86% более низкую вариабельность и в 7 раз лучшую воспроизводимость, чем ее аналог кПЦР, и такой же предел обнаружения. ОТ-цПЦР могла лучше различать образцы сыворотки крови с наличием или отсутствием miR-141 — маркера рака простаты. Аналогичными исследованиями удалось подтвердить микроРНК-биомаркеры плазмы крови для рака легких [34] и рака молочной железы [34].
С использованием системы цПЦР от Fluidigm (сейчас Standard BioTools) было проведено профилирование внутриклеточной РНК отдельных клеток [35]. Собранные клетки окрашивали и сортировали с помощью флуоресцентно-активируемой сортировки клеток (FACS) , а затем индивидуально лизировали и переносили на чип. В исследовании изучали экспрессию генов в пяти типах гемопоэтических предшественников, и в результате была обнаружена гетерогенная экспрессия PU.1 и GADPH — генов домашнего хозяйства, активность которых ранее считалась постоянной.
Этому методу посвящены статьи «Проточная цитометрия» [36] спецпроекта «12 биологических методов в картинках» и «Проточная цитометрия: где вы были эти пять лет?» [37] цикла «Ультрасовременные методы».
Исследования экспрессии генов с помощью ОТ-цПЦР предлагают абсолютное количественное определение мРНК даже на уровне одной клетки. Альтернативы ОТ-цПЦР с более высокой пропускной способностью — новейшие методы секвенирования транскриптома одной клетки [38], [39].
Наконец, ОТ-цПЦР также применялась для вирусного мониторинга Pegivirus A — скрытого РНК-вируса, который может замедлять прогрессирование ВИЧ [40], [41]. После выделения тотальной клеточной РНК и обратной транскрипции применяли чип Fluidigm 48.770 для цПЦР, что существенно уточнило нижней предел обнаружения (до 3–10 копий/мкл) и повысило оценку точности вариабельности образца (11% по сравнению с 23% для кПЦР).
Количественная оценка библиотек для глубокого полногеномного секвенирования
NGS — так называемое секвенирование нового поколения — начинается с приготовления высококачественной библиотеки ДНК в точном количестве. Создание библиотеки NGS (например, для дальнейшего секвенирования на оборудовании Illumina) включает несколько этапов: фрагментацию образца, выбор размера фрагмента, восстановление концов, лигирование адаптера, обогащение методом ПЦР и количественный анализ. Из этих этапов цПЦР оказалась полезной для оценки целостности выбора размера, обогащения и количественного определения. Используя систему RainDance, исследователи количественно определяли распределение размеров и целостность фрагментов ДНК в своих библиотеках [42]. Они выполнили мультиплексную цПЦР с четырьмя зондами, каждый из которых гибридизовался с ДНК-фрагментами разной длины. При этом приблизительное распределение длин нуклеотидных последовательностей можно количественно определить в цифровом виде, подсчитав количества ампликонов, полученных из конкретного образца. Когда то же самое распределение анализируется с помощью количественной ПЦР, оно имеет коэффициент вариации примерно 10–50%.
Что такое секвенирование вообще и почему так много новых технологий называют просто «новым поколением», читайте в наших материалах «12 методов в картинках: секвенирование нуклеиновых кислот» [43], «Нанопоровое секвенирование: на пороге третьей геномной революции» [44] и «Одноклеточное секвенирование: разделяй, изучай и властвуй» [39].
Полногеномное секвенирование часто требует амплификации предварительно обозначенных локусов, обычно с помощью ПЦР с выбранной библиотекой праймеров. Разделение фрагментированной ДНК на небольшие ячейки эффективно обогащает ДНК-мишени по сравнению с фоном, тем самым улучшая амплификацию редких или трудно амплифицируемых последовательностей. Используя вариант системы RainDance, ученые амплифицировали почти 4000 мишеней одновременно, используя библиотеку праймеров с высоким мультиплексированием [45]. Сначала было создано и объединено несколько библиотек капель, содержащих каждую из пар праймеров. Затем капли библиотеки праймеров последовательно сливали с каплями, содержащими геномный образец. После ПЦР эмульсию капель разбивали, а амплифицированную ДНК обогащали и секвенировали с высокой специфичностью и чувствительностью.
Важный вариант применения цПЦР — количественный анализ библиотек для NGS. Для достижения высокопроизводительного и высококачественного полногеномного секвенирования требуется относительно узкий диапазон емкости загрузки ДНК (например, Illumina предлагает 60 гигабаз на дорожку секвенатора HiSeq 2500. Слишком малое количество ДНК приведет к плохому покрытию, низкой глубине считывания и невозможности обнаружить SNP или редкие последовательности. Слишком большое — к чрезмерной кластеризации, когда не все последовательности генома будут разрешены. Количественный анализ ДНК с помощью спектрофотометрии или флуоресценции является качественным и имеет ограниченную чувствительность, требуя более чем в 1000 раз больше образца, чем сам секвенатор. Получается, что драгоценный образец тратится фактически впустую. Используя чип Fluidigm dPCR с универсальными адаптерными праймерами, биологи провели абсолютный количественный анализ всего лишь 100 молекул матрицы и далее секвенировали библиотеки нанограммового, а не микрограммового масштаба [46].
В другой работе исследователи секвенировали 11 образцов с использованием платформы Illumina HiSeq 2500, а перед загрузкой определили количество ДНК с помощью обоих методов и после проведения эксперимента сравнили корреляцию начальных данных и результатов секвенирования, показав, что количественный анализ с помощью системы Raindance цПЦР более тесно коррелирует с выходом секвенирования по сравнению со рутинным исследованием на приборе Agilent Bioanalyzer, в котором для измерения концентрации используются капиллярный электрофорез и детекция флуоресценции (рис. 7). Похоже, благодаря отличным возможностям количественного анализа и амплификации, цПЦР быстро становится стандартным инструментом в NGS.
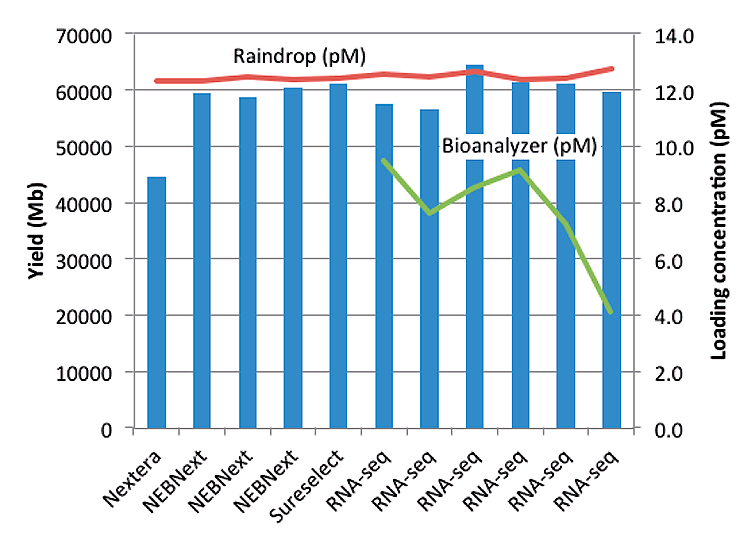
Рисунок 7. Количественный анализ библиотек для NGS: сравнение цПЦР с количественным определением методом капиллярного электрофореза. Образцы секвенировали на HiSeq 2500 с реагентами для секвенирования v4 SBS. Загрузка проточной кюветы основывалась на измерениях концентрации RainDrop. Синие столбцы представляют выход секвенирования в мегабазах, красная линия представляет загрузочную концентрацию, измеренную с помощью RainDrop, зеленая — загрузочную концентрацию, определенную Bioanalyzer (доступно только для 6 библиотек).
Применение цПЦР в лабораторной диагностике
Анализ числа копий (CNV)
Существуют генетические заболевания, обусловленные вариацией числа копий некоторых участков генома, в первую очередь, некоторых генов (CNV). Например, большое количество копий гена ERBB связано с агрессивными формами рака молочной железы [9]. Для некоторых вариантов CNV могут быть различия в сотне копий между людьми, однако в большинстве случаев различий намного меньше: около 10 [47].
По оценкам Международного проекта HapMap, CNV составляют примерно 12% генома человека и включают большую долю нуклеотидов, чем однонуклеотидные полиморфизмы (SNP). Как и в случае с SNP, консорциумы (например, проект CNV, координируемый Институтом Wellcome Trust Sanger) изучают CNV, чтобы исследовать ряд вопросов, включая влияние CNV на экспрессию генов, их роль в заболеваниях и эволюции человека, а также их полезность в картографировании родословных.
Различия в количестве копий конкретного сегмента ДНК могут быть как связаны, так и не связаны с обнаруживаемым изменением фенотипа: CNV чаще встречаются в некодирующих областях. Наличие CNV способствует генетической изменчивости среди особей внутри вида.
Например, меньшее количество копий гена хемокина CCL3L1 может сделать человека более восприимчивым к ВИЧ [48], а дополнительные копии гена EGFR могут привести к повышенному риску рака молочной железы [49]. Анализ CNV требует количественного определения соотношения ДНК-мишени по сравнению с эталоном. В первых отчетах об анализе CNV человека в 2004 г. использовалась сравнительная геномная гибридизация и микрочипы [50], [51], позже для этого начали использовать глубокое секвенирование и кПЦР.
Метод цПЦР обеспечивает бóльшую точность и чувствительность, чем другие методы ПЦР, и идеально подходит для количественного определения небольших различий в количестве копий генов. Таким образом, цПЦР можно использовать для количественной оценки и мониторинга мельчайших различий в CNV, которые могут объяснить разницу между здоровой и опухолевой тканями, а также служить маркерами агрессивности опухоли.
Преимущество цПЦР перед кПЦР заключается в ее способности разрешать небольшие различия в количестве копий. Там, где кПЦР может статистически разрешать двукратные различия (от нуля до двух копий [52]), а с повторностями в 1,25 раза (до четырех копий), один цикл цПЦР может разрешать до восьми копий. Увеличение количества ячеек до 1 миллиона и более улучшает разрешение до 1,1-кратных различий даже при низких концентрациях (λ < 0,1) [53]. Улучшенное разрешение позволяет исследователю идентифицировать не только отдельные делеции и дупликации, но и более сложные изменения, которые играют роль в фенотипах заболеваний.
Как в кПЦР, так и в цПЦР определение CNV требует совместной амплификации мишени с фиксированным эталонным локусом. Затем CNV рассчитывается по соотношению положительных событий для экспериментального и эталонного образцов. В гетерогенных образцах, где сам эталонный ген может иметь CNV, рекомендуют брать два эталонных локуса.
Система BioMark от Standard BioTools была первой коммерческой системой цПЦР, которая в 2008 г. сообщила об исследованиях CNV, продемонстрировав 15% разрешение RPP30, добавленного в два гаплоидных генома [26]. Чтобы избежать тандемных копий (две прикрепленные копии, которые находятся в одной ячейке), авторы выбрали специфическую целевую амплификацию (STA) вместо использования рестриктаз.
Перед цПЦР мишень предварительно амплифицировали вместе с эталонным геном (RNAseP) в течение пяти циклов, производя отдельные ампликоны в том же соотношении, что и в исходном образце. В 2011 году, используя систему Bio-Rad, провели точный анализ CNV до шести копий гена MRGPRX1 в образцах HapMap [32]. Та же система использовалась для измерения амплификации числа копий HER2 в образцах рака молочной железы [54], [55]. Более поздняя система Quantstudio 3D продемонстрировала разрешение до восьми копий в образцах с известным числом копий гена CCL3L1 (коэффициент вариации <2,6%) и в клинических образцах рака молочной железы для гена HER2 (коэффициент вариации 7%).
Используя комбинацию секвенирования и цПЦР, одна из групп исследователей предоставила ранние доказательства того, что CNVs в циркулирующих опухолевых клетках специфичны для типов рака и воспроизводимы среди пациентов [56].
Неинвазивный пренатальный тест (НИПТ)
Анализ числа копий также используется в пренатальных скринингах на анеуполоидию плода [57]. Трисомия обычно диагностируется с помощью цитогенетического кариотипирования хромосом, полученных при амниоцентезе, или в образцах ворсин хориона; однако тест может занять до 1–2 недель. Используя цПЦР, можно точно идентифицировали все случаи трисомии в образцах в течение нескольких часов [57].
Жидкостная биопсия
цПЦР можно использовать для жидкостной биопсии — методе диагностики рака, в котором измеряется выброс цоДНК (циркулирующей опухолевой ДНК) в кровоток пациента [58], [59]. Менее инвазивная, чем прямая биопсия, жидкостная биопсия особенно привлекательна, когда опухоль неоперабельна. Несколько соматических мутаций в цоДНК (например, мутации в генах KRAS, BRAF, NRAS, TP53, PIK3CA и др.) представляют новое поколение биомаркеров рака, которые могут предсказывать, например, прогрессирование метастатических опухолей [60], ответ на терапию [61] или устойчивость к химиотерапевтическим препаратам [60], [62]. Например, широкомасштабное исследование продемонстрировало клиническую полезность цоДНК для мониторинга прогрессирования и даже потенциальной заболеваемости различными видами рака [60]. В нем обнаружили, что доля пациентов, у которых обнаруживалась цоДНК, увеличивалась в зависимости от стадии рака. Концентрация цоДНК также увеличивалась со стадией, и люди с более низким уровнем цоДНК в крови жили значительно дольше, чем люди с более высоким уровнем.
Проблема жидкостной биопсии заключается в том, что цоДНК обычно составляет <1% от фрагментированной ДНК в кровотоке. Более того, только часть цоДНК (<10%) содержит интересующие мутации [63]. Отличительной чертой цПЦР является ее способность обнаруживать редкие мутантные формы путем разделения, которое обогащает их по сравнению с нормальным вариантом. Системы цПЦР от RainDance и Bio-Rad могут детектировать редких мутантов с частотой до 0,0005% [29] по сравнению с 1% для количественной ПЦР и 2% для NGS [58]. Кроме того, NGS может определить только наличие мутации, цПЦР же может дать ее количественную оценку, что позволяет отслеживать прогрессирование заболевания.
Несколько исследований показали многообещающие результаты по обнаружению цоДНК в крови пациентов с опухолями молочной железы с использованием системы BioMark от Standard BioTools: было показано, что распространенность локусов PIK3CA и TP53 является лучшим предиктором опухолевой нагрузки, чем подсчет циркулирующих опухолевых клеток или проведение иммунологического анализа на раковый антиген CA15-3 [64].
Используя вариант системы RainDance, исследователи разработали мультиплексную цПЦР для выявления мутаций в онкогене KRAS у больных колоректальным раком [65]. Они смогли обнаружить до пяти мутаций в одном эксперименте и достигли разрешающей чувствительности к редким мутантам 0,0005% (1 из 200 000) [30].
Мутации, активирующие эндоплазматический фактор роста (EGFR), делают опухоль более восприимчивой к терапии, основанной на ингибиторах тирозинкиназы, например, эрлотиниба. Количественное определение EGFR T790M с чувствительностью 0,005% позволило предсказать, будет ли опухоль чувствительна к этому лекарству или разовьет к нему устойчивость. Мутации были обнаружены за 16 недель до того, как рост опухоли можно было наблюдать с помощью радиометрии [66]. Эти исследования показывают перспективность цПЦР в ранней диагностике и в руководстве терапевтическим лечением — такой подход можно отнести к фармакогеномике [67].
Исследование вирусной нагрузки
Некоторые патогены присутствуют в исследуемом образце в небольшом количестве, что затрудняет использование для их обнаружения традиционной количественной ПЦР. цПЦР же обеспечивает беспрецедентную точность без необходимости внедрения внутреннего стандартного контроля, что позволяет сэкономить время и деньги в долгосрочной перспективе.
Точная количественная оценка циркулирующей вирусной или бактериальной ДНК имеет решающее значение не только для ранней диагностики, но и для мониторинга прогрессирования инфекции и ответа на лечение [40]. Пациенты с ВИЧ, получающие ретровирусную терапию, регулярно проходят контроль на наличие остаточной инфекции, а пациенты с трансплантацией органов с подавленным иммунитетом — скрининг на цитомегаловирус, вирус Эпштейна—Барр [68] и другие опасные в условиях иммуносупрессии вирусы. Ранние диагностические подходы для выявления патогенов раньше основывались на световой микроскопии, однако этот подход был трудоемким и не позволял визуализировать вирусы.
С 1990-х годов золотым стандартом вирусологического тестирования стала количественная ПЦР, которая количественно определяет вирусную или бактериальную нагрузку путем измерения циркулирующей ДНК. Однако в количественной ПЦР построение калибровочных кривых и эффективность амплификации могут сильно различаться от лаборатории к лаборатории, что приводит к коэффициенту вариации 20–30% при низких концентрациях матрицы [40]. цПЦР оказалась удобной альтернативой другим вариантам ПЦР, обеспечивая абсолютную количественную оценку ДНК-мишеней [69], [70].
В 2008 году ранний вариант платформы RainDance использовался для амплификации аденовирусов с пределом детекции всего 1 матрица на 167 капель [71]. В 2013 г. исследователи использовали систему Bio-Rad QX100 для количественного определения HIV-pol и 2-LTR в более чем 150 клинических образцах крови [72]. Для HIV-pol они определили, что цПЦР была в пять раз более точной, чем кПЦР, для частот ДНК-мишени менее 300 копий на миллион клеток. Предел, определенный по ошибке подвыборки, составил 14 копий на миллион клеток (доверительный интервал 95%).
Отслеживать вирусы важно не только в организме. Наблюдение за сточными водами для выявления вспышек и распространения инфекционных заболеваний становится всё более востребованным направлением исследований. Во время пандемии интерес к тестированию сточных вод многократно возрос. Применение цПЦР в таком анализе может помочь количественно определять COVID-19 в популяциях и отслеживать динамику мутаций.
Применение цПЦР в пищевом и сельскохозяйственном тестировании
цПЦР нашла применение при анализе образцов окружающей среды (семян, растительного материала, почвы и сточных вод), где ингибиторы, присутствующие в образце, могут негативно влиять на процесс выделения ДНК и снизить эффективность и воспроизводимость кПЦР [73].
Исследователи продемонстрировали пользу цПЦР при количественном определении генетически модифицированных организмов (ГМО) в образцах кукурузного корма [74], [75]. В анализе с помощью цПЦР сравнивалось количество копий трансгена MON810 с количеством копий эталонного гена HMG. В результате удалось обнаружить пять копий ДНК-мишени, а также достичь широкого динамического диапазона, аналогичного кПЦР. В ходе работы было показано, что цПЦР дает лучшую повторяемость результатов и лучшую устойчивость к ингибиторам порошка семян, чем кПЦР.
цПЦР также использовалась для количественного определения патогенных бактерий в растениях [76] и для обнаружения вируса легкой крапчатости перца [73]. В последнем исследовании было показано, что цПЦР более устойчив к сложным матрицам и ингибиторам, чем кПЦР [73].
Вместо заключения
Простая на первый взгляд идея разделения образца на тысячи лунок и капель с анализом амплификации по принципу «всё или ничего» и последующая статистическая обработка результатов на основе распределения Пуассона сделала цПЦР удобным методом для изучения минимальных различий между образцами и детекции редких событий (например, мутаций во внеклеточной ДНК или определения количества копий локусов в геноме). Под потребности такого анализа подстраивается и рынок приборов: с каждым годом становится всё больше систем для проведения цПЦР, различающихся производительностью, количеством одновременно анализируемых образцов, возможностью точной оценки концентрации ДНК-мишени. Судя по всему, совсем скоро цПЦР станет стандартом не только для проведения высокотехнологичных тестов в лабораторной диагностике, но и для решения рутинных научных задач, например, оценки качества библиотек для секвенирования.
Литература
- P Simmonds, P Balfe, J F Peutherer, C A Ludlam, J O Bishop, A J Brown. (1990). Human immunodeficiency virus-infected individuals contain provirus in small numbers of peripheral mononuclear cells and at low copy numbers. J Virol. 64, 864-872;
- Sykes P.J., Neoh S.H., Brisco M.J., Hughes E., Condon J., Morley A.A. (1992). Quantitation of targets for PCR by use of limiting dilution. Biotechniques. 13, 444–449;
- Alexander A. Morley. (2014). Digital PCR: A brief history. Biomolecular Detection and Quantification. 1, 1-2;
- Bert Vogelstein, Kenneth W. Kinzler. (1999). Digital PCR. Proc. Natl. Acad. Sci. U.S.A.. 96, 9236-9241;
- Ингибиторы Ras: в поисках Грааля таргетной терапии;
- 12 методов в картинках: полимеразная цепная реакция;
- Manit Arya, Iqbal S Shergill, Magali Williamson, Lyndon Gommersall, Neehar Arya, Hitendra RH Patel. (2005). Basic principles of real-time quantitative PCR. Expert Review of Molecular Diagnostics. 5, 209-219;
- Hua J. Lou, Xiaohong Fang, Lisa Hilliard, Xiaojing Liu, Jianwei J. Li, et. al.. (2001). Ultrasensitive DNA and protein analysis using molecular beacon probes. SPIE Proceedings;
- Stephen A Bustin, Vladimir Benes, Jeremy A Garson, Jan Hellemans, Jim Huggett, et. al.. (2009). The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chemistry. 55, 611-622;
- David Svec, Ales Tichopad, Vendula Novosadova, Michael W. Pfaffl, Mikael Kubista. (2015). How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16;
- O Kalinina. (1997). Nanoliter scale PCR with TaqMan detection. Nucleic Acids Research. 25, 1999-2004;
- Simant Dube, Jian Qin, Ramesh Ramakrishnan. (2008). Mathematical Analysis of Copy Number Variation in a DNA Sample Using Digital PCR on a Nanofluidic Device. PLoS ONE. 3, e2876;
- Alexandra S. Whale, Simon Cowen, Carole A. Foy, Jim F. Huggett. (2013). Methods for Applying Accurate Digital PCR Analysis on Low Copy DNA Samples. PLoS ONE. 8, e58177;
- Leonardo B. Pinheiro, Victoria A. Coleman, Christopher M. Hindson, Jan Herrmann, Benjamin J. Hindson, et. al.. (2012). Evaluation of a Droplet Digital Polymerase Chain Reaction Format for DNA Copy Number Quantification. Anal. Chem.. 84, 1003-1011;
- Jim F Huggett, Simon Cowen, Carole A Foy. (2015). Considerations for Digital PCR as an Accurate Molecular Diagnostic Tool. Clinical Chemistry. 61, 79-88;
- Kevin A Heyries, Carolina Tropini, Michael VanInsberghe, Callum Doolin, Oleh I Petriv, et. al.. (2011). Megapixel digital PCR. Nat Methods. 8, 649-651;
- Jason E. Kreutz, Todd Munson, Toan Huynh, Feng Shen, Wenbin Du, Rustem F. Ismagilov. (2011). Theoretical Design and Analysis of Multivolume Digital Assays with Wide Dynamic Range Validated Experimentally with Microfluidic Digital PCR. Anal. Chem.. 83, 8158-8168;
- Feng Shen, Bing Sun, Jason E. Kreutz, Elena K. Davydova, Wenbin Du, et. al.. (2011). Multiplexed Quantification of Nucleic Acids with Large Dynamic Range Using Multivolume Digital RT-PCR on a Rotational SlipChip Tested with HIV and Hepatitis C Viral Load. J. Am. Chem. Soc.. 133, 17705-17712;
- Andrew C. Hatch, Jeffrey S. Fisher, Armando R. Tovar, Albert T. Hsieh, Robert Lin, et. al.. (2011). 1-Million droplet array with wide-field fluorescence imaging for digital PCR. Lab Chip. 11, 3838;
- Zhi Zhu, Gareth Jenkins, Wenhua Zhang, Mingxia Zhang, Zhichao Guan, Chaoyong James Yang. (2012). Single-molecule emulsion PCR in microfluidic droplets. Anal Bioanal Chem. 403, 2127-2143;
- Somanath Bhat, Jan Herrmann, Paul Armishaw, Philippe Corbisier, Kerry R. Emslie. (2009). Single molecule detection in nanofluidic digital array enables accurate measurement of DNA copy number. Anal Bioanal Chem. 394, 457-467;
- Lianhua Dong, Ying Meng, Zhiwei Sui, Jing Wang, Liqing Wu, Boqiang Fu. (2015). Comparison of four digital PCR platforms for accurate quantification of DNA copy number of a certified plasmid DNA reference material. Sci Rep. 5;
- Somanath Bhat, Jacob L. H. McLaughlin, Kerry R. Emslie. (2011). Effect of sustained elevated temperature prior to amplification on template copy number estimation using digital polymerase chain reaction. Analyst. 136, 724-732;
- Kate R. Griffiths, Daniel G. Burke, Kerry R. Emslie. (2011). Quantitative polymerase chain reaction: a framework for improving the quality of results and estimating uncertainty of measurement. Anal. Methods. 3, 2201;
- Rebecca Sanders, Jim F. Huggett, Claire A. Bushell, Simon Cowen, Daniel J. Scott, Carole A. Foy. (2011). Evaluation of Digital PCR for Absolute DNA Quantification. Anal. Chem.. 83, 6474-6484;
- Jian Qin, Robert C. Jones, Ramesh Ramakrishnan. (2008). Studying copy number variations using a nanofluidic platform. Nucleic Acids Research. 36, e116-e116;
- Sandra L. Spurgeon, Robert C. Jones, Ramesh Ramakrishnan. (2008). High Throughput Gene Expression Measurement with Real Time PCR in a Microfluidic Dynamic Array. PLoS ONE. 3, e1662;
- Colin Brenan, Tom Morrison. (2005). High throughput, nanoliter quantitative PCR. Drug Discovery Today: Technologies. 2, 247-253;
- Deniz Pekin, Yousr Skhiri, Jean-Christophe Baret, Delphine Le Corre, Linas Mazutis, et. al.. (2011). Quantitative and sensitive detection of rare mutations using droplet-based microfluidics. Lab Chip. 11, 2156;
- Qun Zhong, Smiti Bhattacharya, Steven Kotsopoulos, Jeff Olson, Valérie Taly, et. al.. (2011). Multiplex digital PCR: breaking the one target per color barrier of quantitative PCR. Lab Chip. 11, 2167;
- Eric Brouzes, Martina Medkova, Neal Savenelli, Dave Marran, Mariusz Twardowski, et. al.. (2009). Droplet microfluidic technology for single-cell high-throughput screening. Proc. Natl. Acad. Sci. U.S.A.. 106, 14195-14200;
- Benjamin J. Hindson, Kevin D. Ness, Donald A. Masquelier, Phillip Belgrader, Nicholas J. Heredia, et. al.. (2011). High-Throughput Droplet Digital PCR System for Absolute Quantitation of DNA Copy Number. Anal. Chem.. 83, 8604-8610;
- Christopher M Hindson, John R Chevillet, Hilary A Briggs, Emily N Gallichotte, Ingrid K Ruf, et. al.. (2013). Absolute quantification by droplet digital PCR versus analog real-time PCR. Nat Methods. 10, 1003-1005;
- Jie Ma, Ning Li, Maria Guarnera, Feng Jiang. (2013). Quantification of Plasma miRNAs by Digital PCR for Cancer Diagnosis. Biomark Insights. 8, BMI.S13154;
- Luigi Warren, David Bryder, Irving L. Weissman, Stephen R. Quake. (2006). Transcription factor profiling in individual hematopoietic progenitors by digital RT-PCR. Proc. Natl. Acad. Sci. U.S.A.. 103, 17807-17812;
- 12 методов в картинках: проточная цитофлуориметрия;
- Проточная цитометрия: где вы были эти пять лет?;
- Evan Z. Macosko, Anindita Basu, Rahul Satija, James Nemesh, Karthik Shekhar, et. al.. (2015). Highly Parallel Genome-wide Expression Profiling of Individual Cells Using Nanoliter Droplets. Cell. 161, 1202-1214;
- Одноклеточное секвенирование: разделяй, изучай и властвуй;
- Ruth Hall Sedlak, Keith R. Jerome. (2013). Viral diagnostics in the era of digital polymerase chain reaction. Diagnostic Microbiology and Infectious Disease. 75, 1-4;
- Richard Allen White, Stephen R. Quake, Kenneth Curr. (2012). Digital PCR provides absolute quantitation of viral load for an occult RNA virus. Journal of Virological Methods. 179, 45-50;
- Audrey Didelot, Steve K Kotsopoulos, Audrey Lupo, Deniz Pekin, Xinyu Li, et. al.. (2013). Multiplex Picoliter-Droplet Digital PCR for Quantitative Assessment of DNA Integrity in Clinical Samples. Clinical Chemistry. 59, 815-823;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Нанопоровое секвенирование: на пороге третьей геномной революции;
- Ryan Tewhey, Jason B Warner, Masakazu Nakano, Brian Libby, Martina Medkova, et. al.. (2009). Microdroplet-based PCR enrichment for large-scale targeted sequencing. Nat Biotechnol. 27, 1025-1031;
- Richard A White, Paul C Blainey, H Christina Fan, Stephen R Quake. (2009). Digital PCR provides sensitive and absolute calibration for high throughput sequencing. BMC Genomics. 10;
- Andy Itsara, Gregory M. Cooper, Carl Baker, Santhosh Girirajan, Jun Li, et. al.. (2009). Population Analysis of Large Copy Number Variants and Hotspots of Human Genetic Disease. The American Journal of Human Genetics. 84, 148-161;
- Enrique Gonzalez, Hemant Kulkarni, Hector Bolivar, Andrea Mangano, Racquel Sanchez, et. al.. (2005). The Influence of CCL3L1 Gene-Containing Segmental Duplications on HIV-1/AIDS Susceptibility. Science. 307, 1434-1440;
- G Peiró, D Mayr, P Hillemanns, U Löhrs, J Diebold. (2004). Analysis of HER-2/neu amplification in endometrial carcinoma by chromogenic in situ hybridization. Correlation with fluorescence in situ hybridization, HER-2/neu, p53 and Ki-67 protein expression, and outcome. Mod Pathol. 17, 277-287;
- A John Iafrate, Lars Feuk, Miguel N Rivera, Marc L Listewnik, Patricia K Donahoe, et. al.. (2004). Detection of large-scale variation in the human genome. Nat Genet. 36, 949-951;
- Jonathan Sebat, B. Lakshmi, Jennifer Troge, Joan Alexander, Janet Young, et. al.. (2004). Large-Scale Copy Number Polymorphism in the Human Genome. Science. 305, 525-528;
- Suzanne Weaver, Simant Dube, Alain Mir, Jian Qin, Gang Sun, et. al.. (2010). Taking qPCR to a higher level: Analysis of CNV reveals the power of high throughput qPCR to enhance quantitative resolution. Methods. 50, 271-276;
- Alexandra S. Whale, Jim F. Huggett, Simon Cowen, Valerie Speirs, Jacqui Shaw, et. al.. (2012). Comparison of microfluidic digital PCR and conventional quantitative PCR for measuring copy number variation. Nucleic Acids Research. 40, e82-e82;
- Heidrun Gevensleben, Isaac Garcia-Murillas, Monika K. Graeser, Gaia Schiavon, Peter Osin, et. al.. (2013). Noninvasive Detection of HER2 Amplification with Plasma DNA Digital PCR. Clinical Cancer Research. 19, 3276-3284;
- Nicholas J. Heredia, Phillip Belgrader, Shenglong Wang, Ryan Koehler, Jack Regan, et. al.. (2013). Droplet Digital™ PCR quantitation of HER2 expression in FFPE breast cancer samples. Methods. 59, S20-S23;
- Xiaohui Ni, Minglei Zhuo, Zhe Su, Jianchun Duan, Yan Gao, et. al.. (2013). Reproducible copy number variation patterns among single circulating tumor cells of lung cancer patients. Proc. Natl. Acad. Sci. U.S.A.. 110, 21083-21088;
- H. Christina Fan, Yair J. Blumenfeld, Yasser Y. El-Sayed, Jane Chueh, Stephen R. Quake. (2009). Microfluidic digital PCR enables rapid prenatal diagnosis of fetal aneuploidy. American Journal of Obstetrics and Gynecology. 200, 543.e1-543.e7;
- Luis A. Diaz, Alberto Bardelli. (2014). Liquid Biopsies: Genotyping Circulating Tumor DNA. JCO. 32, 579-586;
- Jonathan C. M. Wan, Charles Massie, Javier Garcia-Corbacho, Florent Mouliere, James D. Brenton, et. al.. (2017). Liquid biopsies come of age: towards implementation of circulating tumour DNA. Nat Rev Cancer. 17, 223-238;
- Chetan Bettegowda, Mark Sausen, Rebecca J. Leary, Isaac Kinde, Yuxuan Wang, et. al.. (2014). Detection of Circulating Tumor DNA in Early- and Late-Stage Human Malignancies. Sci. Transl. Med.. 6;
- Simon Chang-Hao Tsao, Jonathan Weiss, Christopher Hudson, Christopher Christophi, Jonathan Cebon, et. al.. (2015). Monitoring response to therapy in melanoma by quantifying circulating tumour DNA with droplet digital PCR for BRAF and NRAS mutations. Sci Rep. 5;
- Astrid Lièvre, Jean-Baptiste Bachet, Valérie Boige, Anne Cayre, Delphine Le Corre, et. al.. (2008). KRAS Mutations As an Independent Prognostic Factor in Patients With Advanced Colorectal Cancer Treated With Cetuximab. JCO. 26, 374-379;
- Frank Diehl, Meng Li, Devin Dressman, Yiping He, Dong Shen, et. al.. (2005). Detection and quantification of mutations in the plasma of patients with colorectal tumors. Proc. Natl. Acad. Sci. U.S.A.. 102, 16368-16373;
- Sarah-Jane Dawson, Dana W.Y. Tsui, Muhammed Murtaza, Heather Biggs, Oscar M. Rueda, et. al.. (2013). Analysis of Circulating Tumor DNA to Monitor Metastatic Breast Cancer. N Engl J Med. 368, 1199-1209;
- Valerie Taly, Deniz Pekin, Leonor Benhaim, Steve K Kotsopoulos, Delphine Le Corre, et. al.. (2013). Multiplex Picodroplet Digital PCR to Detect KRAS Mutations in Circulating DNA from the Plasma of Colorectal Cancer Patients. Clinical Chemistry. 59, 1722-1731;
- Geoffrey R. Oxnard, Cloud P. Paweletz, Yanan Kuang, Stacy L. Mach, Allison O'Connell, et. al.. (2014). Noninvasive Detection of Response and Resistance in EGFR-Mutant Lung Cancer Using Quantitative Next-Generation Genotyping of Cell-Free Plasma DNA. Clinical Cancer Research. 20, 1698-1705;
- Фармакогеномика: изучение генов на службе персонализированной медицины;
- От поцелуя до лимфомы один вирус;
- Tanis C Dingle, Ruth Hall Sedlak, Linda Cook, Keith R Jerome. (2013). Tolerance of Droplet-Digital PCR vs Real-Time Quantitative PCR to Inhibitory Substances. Clinical Chemistry. 59, 1670-1672;
- Ruth Hall Sedlak, Keith R Jerome. (2014). The potential advantages of digital PCR for clinical virology diagnostics. Expert Review of Molecular Diagnostics. 14, 501-507;
- Margaret Macris Kiss, Lori Ortoleva-Donnelly, N. Reginald Beer, Jason Warner, Christopher G. Bailey, et. al.. (2008). High-Throughput Quantitative Polymerase Chain Reaction in Picoliter Droplets. Anal. Chem.. 80, 8975-8981;
- Matthew C. Strain, Steven M. Lada, Tiffany Luong, Steffney E. Rought, Sara Gianella, et. al.. (2013). Highly Precise Measurement of HIV DNA by Droplet Digital PCR. PLoS ONE. 8, e55943;
- Nejc Rački, Tanja Dreo, Ion Gutierrez-Aguirre, Andrej Blejec, Maja Ravnikar. (2014). Reverse transcriptase droplet digital PCR shows high resilience to PCR inhibitors from plant, soil and water samples. Plant Methods. 10;
- Mojca Milavec, David Dobnik, Litao Yang, Dabing Zhang, Kristina Gruden, Jana Žel. (2014). GMO quantification: valuable experience and insights for the future. Anal Bioanal Chem. 406, 6485-6497;
- Dany Morisset, Dejan Štebih, Mojca Milavec, Kristina Gruden, Jana Žel. (2013). Quantitative Analysis of Food and Feed Samples with Droplet Digital PCR. PLoS ONE. 8, e62583;
- Amar S. Basu. (2017). Digital Assays Part I: Partitioning Statistics and Digital PCR. SLAS Technology. 22, 369-386;
- Phenix-Lan Quan, Martin Sauzade, Eric Brouzes. (2018). dPCR: A Technology Review. Sensors. 18, 1271.