
Дмитрий Пензар: «В задачах регуляторной геномики мы видим, что всегда побеждают нейронные сети»
31 декабря 2025
Дмитрий Пензар: «В задачах регуляторной геномики мы видим, что всегда побеждают нейронные сети»
- 638
- 0
- 1
Супергерой этого интервью — Дмитрий Пензар — неслучайно изображен в киберпанковской стилистике в струях цифрового (квази)нуклеотидного дождя. Ведь, раз он лауреат премии за искусственный интеллект, и эту картинку нарисовать должен был ИИ.
Рисунок в полном размере.
картинка сгенерирована в Flux 1.1 (но это неточно)
-
Автор
-
Редакторы
9 декабря 2025 года Дмитрий Пензар, кандидат биологических наук, научный сотрудник Лаборатории системной биологии и вычислительной генетики Института общей генетики (ИОГен) РАН получил Научную премию Сбера в номинации «AI в науках о жизни» «за разработку нейросетей для моделирования, прогнозирования свойств и рационального конструирования нуклеотидных последовательностей ДНК, контролирующих работу генов, для решения задач биотехнологий и медицинской генетики». Дмитрий Пензар — специалист по нейронным сетям в регуляторной геномике. Надежда Маркина поговорила с лауреатом о проектах, в которых он участвовал, и в целом о том, какую роль модный ныне искусственный интеллект играет в этой области биологии.
Зачем биологам нейросети
— Дмитрий, вы можете объяснить, почему нейросети так востребованы в геномике? Что они принципиально дают такого, чего без них невозможно узнать?
Во многом тут та же история, что в принципе с применением нейронных сетей, где бы то ни было. У вас накапливаются данные, и на каком-то этапе вам хватает эксперта, чтобы он посмотрел данные десяти экспериментов и сказал, что, наверное, это работает таким-то образом, поэтому мы можем воздействовать на это и получить вот это. А когда данных становится всё больше, то эксперт уже не отсмотрит десять тысяч экспериментов. Тогда начинается применение биоинформатики, то есть биоалгоритмы, точные правила и эвристики выдают человеку какой-то сжатый репорт, с которым можно поработать и получить какие-то выводы. Но когда у вас этих экспериментов становится еще больше или у них масштаб становится другим — когда вы активность не десяти последовательностей посмотрели, а миллиона или десятков миллионов, эксперт не сможет создать такой детерминированный бионформатический алгоритм, который будет это всё обрабатывать и выдавать какую-то сжатую информацию. Вот на этом этапе как раз и начинается машинное обучение. Нейронные сети работают лучше, чем эвристика, и лучше, чем простые статистические модели. Вы можете предоставить им данные, задать то, что они должны по каждой последовательности предсказать, обучить их, и они смогут вычислить некие закономерности, которые и эксперт пропустит, и традиционной программе трудно обнаружить.
— Могу я задать несколько вопросов для общего понимания, «для чайников»? Есть просто машинное обучение, есть глубокое. В чем разница?
Классическое машинное обучение — оно ближе к статистике. Скажем, есть линейная регрессия, и это уже, в принципе, машинное обучение. Конечно, ею классическое машинное обучение не исчерпывается, но даже она порой очень хорошо работает. Настолько, что другие методы не могут выступить лучше. Классическое обучение работает чаще всего с табличными данными. Это значит, что есть объект, есть его некое описание, оно выполнено экспертом либо какой-то программой. Там написано, что встречаются такие-то паттерны, в таком-то количестве. И вот по этому вектору, который получается для каждой записи, модель машинного обучения должна научиться предсказывать интересующие нас величины.
Традиционные методы машинного обучения занимали в геномной биоинформатике основное место всего лишь несколько лет назад. «Биомолекула» уже рассказывала об одной из таких работ [1] с участием Дмитрия: «Фальшивые мотивы в ДНК: как геномные варианты меняют поведение транскрипционных факторов» [2]. — Ред.
— А глубокое машинное обучение, насколько я понимаю, — где есть несколько слоев? В чем их смысл?
Основная идея, что у вас каждый слой учится выделять какие-то интересные признаки. Если в классическом машинном обучении вы закладываете данные, уже обработанные программами либо экспертами, глубокое машинное обучение, как правило, работает с неструктурированными, сырыми данными. Машина пропускает их через первый слой нейронной сети и выделяет самые простые признаки. Потом следующий слой пытается из этих простых признаков сложить более сложные. И так далее. Получается такой поэтапный процесс. И, наконец, выходят финальные признаки, которые помогут решить задачу. Условно, если задача — классифицировать то, что у нас на картинке — не на два класса делить животных, а на тысячу объектов, то на последнем слое нейронной сети у нас будет как раз тысяча признаков, и каждый признак будет отвечать на вопрос, есть ли на этом изображении объект. А дальше мы просто их объединяем.
Дальше возникает вопрос, с какого момента у нас получается глубокое машинное обучение? Четкого ответа на этот вопрос нет. Вот два слоя, это много или мало? Очень теоретически, их уже для большей части задач должно хватать. И для каких-то задач, действительно, двух или трех слоев достаточно. А десять слоев? Это уж точно глубокое обучение, хотя бывает и сто слоев.
— Еще есть модели с учителем и без учителя?
Да. Ну, с учителем — это то, что обычно мы и имеем в виду, когда говорим о машинном обучении. Вот у вас задачка: вам на вход дали картинку кошки, и задача — определить, есть ли кошка на картинке. И есть датасет — 10 тысяч изображений, где написано, есть там кошка или нет. Он-то как раз и выступает в роли учителя. То есть, есть задача, на нее есть ответ, и модель учится выдавать этот ответ. А пример обучения без учителя — ну самое простое, это задача кластеризации. Представьте себе, что у вас есть миллион организмов, и вы их хотите как-то разбить на группы.
— А по каким признакам — неизвестно?
У машины как раз задача в том, чтобы найти какие-то общие черты и как-то их разделить — на иерархические группы или просто группы. Это, в принципе, то, что сводится к систематике, например, к современной классификации видов. Вы ответа не знаете, известно только то, что вот это вы называете рыбой, вот это называете змеей, а может, это и не змея вовсе... И у вас есть миллион последовательностей, всё, что вы насобирали. И методу нужно полагаться на какие-то там эвристики, на то, что у похожих объектов эти последовательности должны быть похожими, а у разных — разными... То есть, в этой истории у вас есть поставленная задача, но нет ответа.
Вообще, определение методов по тому, с учителем они, без учителя, довольно усложнилось в наше время. Например, сейчас часто модель учат на задаче, ответ для которой генерируется из самих данных (замазали часть текста — предскажи, что замазано). А далее уже обученную модель дополнительно учат решать более практические задачи. Это называется уже self-supervised learning. И часто отделяется от обучения без учителя. Хотя, по сути, во многом его подобласть.
— Давайте уточним — ваша конкретная область исследований — это нейронные сети в области регуляторной геномики? Можно привести примеры задач, которые в этой области приходится решать?
Одна из уже классических задач: есть регуляторный белок, который узнает какие-то короткие сигналы — текстовые паттерны в ДНК, — и нужно научиться предсказывать аналогичные участки в новой неизученной последовательности. Или сложнее — давайте научимся не просто предсказывать связывание, а предсказывать, как данная последовательность будет влиять на активность экспрессии рядом. Или шаг еще дальше — давайте такую последовательность сконструируем. И часто такие задачи не машинным обучением не решаются вовсе.
Вот любимый пример моего научного руководителя — когда пытались делать дизайн регуляторных областей по геному дрозофилы, и было точно известно из экспериментов, что вот эта область важна, эта область важна, эта область важна, а между ними, как будто, ничего важного нет. Из этого следует вывод, что мы можем всё это неважное выкинуть либо заменить чем-то другим неважным, и у нас по-прежнему эта регуляторная область будет работать. А оно так не получается, не работает. Это пример того, когда мы вроде бы глазами можем что-то пронаблюдать и сделать какие-то выводы, но они оказываются неполными. А нейронная сеть в такой ситуации вам прямо скажет, что это работать не будет, потому что вы правильно вычислили три главных определяющих кусочка, но оставшиеся кусочки тоже что-то делают, и без них регуляторная область не работает.
— Какое-то из наиболее ярких биоинформатических достижений можете назвать?
В истории биоинформатики одна из самых крупных побед — это AlphaFold2 : он точно работает лучше всего остального в предсказании пространственной структуры белков. Мы точно знаем, что там глубокое машинное обучение справилось лучше экспертов. Там есть еще несколько других обещаний, которые не сбылись.
AlphaFold — модель искусственного интеллекта на основе глубокого машинного обучения, разработанная Google DeepMind, которая выполняет предсказания пространственной структуры белка. В 2024 году руководители AlphaFold Демис Хассабис и Джон Джампер получили Нобелевскую премии по химии: «Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024)» [3]. См. также: «AlphaFold: нейросеть для предсказания структуры белков от британских ученых» [4], «Белковые галлюцинации: как справляется AlphaFold?» [5] и «От структуры к функции: революция ИИ в белковом дизайне» [6].
С чего начался бум машинного обучения — нейронные сети научились классифицировать картинки лучше человека . То есть сначала было так: условно, у человека был уровень ошибок 5%, а у моделей до нейросетей — 30%. А лет через семь появились модели, которые решали задачу в два раза лучше, чем человек . Вот с AlphaFold2 мы такую же картину наблюдаем, и в некоторых других областях биологии, что какие-то важные задачи получается лучше решать нейронными сетями. И с задачами регуляторной геномики мы видим, что в разных конкурсах по предсказанию эффектов вариантов, по предсказанию активности регуляторных последовательностей всегда побеждают нейронные сети. Если люди приходят с другими подходами, то их решения оказываются хуже любого сколь угодно простого метода на основе нейросетей.
Об этих достижениях рассказывает спецпроект «Биомолекулы» «Искусственный интеллект в биологии»: «История развития искусственного интеллекта и его пришествия в биологию» [7] и «Как языковые модели покорили мир белков» [8]. — Ред.
При этом надо сказать, что предсказания моделей машинного обучения для регуляторной геномики, конечно, находятся еще на академическом уровне. Например, хотим мы делать клиническую диагностику. Ну вот, от создателей AlphaFold есть нейронная сеть AlphaGenome . Так, если не читать пресс-релиз компании, а читать документацию к этой сети у них сайте, так там почти прямо написано, что для клинической практики она не особо применима. Ну или выпустила Illumina свою сеть для предсказания эффектов регуляторных мутаций — PromoterAI. Опять, в пресс-релизе, в статье — уже бороздим просторы вселенной и лечим людей от всего. А в лицензионном соглашении, которое должен подписать каждый, использующий их сеть, четко написано — не является инструментом диагностики.
AlphaGenome — модель искусственного интеллекта, разработанная Google DeepMind, направленная на анализ человеческого генома.
Поэтому предсказание влияния регуляторных мутаций, как они будут влиять на человека — до полного решения этой задачи нейронными сетями еще очень-очень далеко. Это пока еще на уровне обещания и рекламы.
— Вы в своей работе чаще используете готовые нейронные сети или создаете свои?
Зависит от задачи и ресурсов. Иногда первая проблема, с которой ты сталкиваешься — это вычислительные ресурсы. То есть, ты можешь быть бесконечно умным, и в принципе, ты можешь поставить задачу так же, как ее поставили авторы какой-то статьи, может, даже лучше придумать, как решить ее, но, чтобы обучить модель с нуля, тебе нужен сервер с большим числом видеокарт, и чтобы он три месяца был включен только в твою задачу. Это не всегда достижимо. В этом случае используй что есть.
Иногда ты не самый умный, и люди сделали хорошо. Или тебе нужно какое-то время, чтобы понять, как улучшить решение. Причем заранее понятно, что прям сильно не улучшишь. Такое тоже часто бывает. Вот в этих случаях мы вполне можем взять чужую нейросеть, например, есть AlphaGenome, который я упоминал, для наших задач он иногда работает, но с ним проблема, что его очень тяжело использовать из-за лицензии. А есть открытые модели, Flashzoi, например, которые не сильно хуже [9]. То есть вполне можно использовать чужие модели, обычно они публикуются с какими-то условиями человечными, то есть ты их можешь использовать, но не можешь коммерциализировать, например.
Ну и в принципе, даже если ты решил учить свою модель, тебе все равно нужно запустить чужую, чтобы понимать, какие подходы уже перебрали в этой области. Потому что, если ты смотришь литературу, то у тебя вываливается 50 статей, в каждой из которых говорится, что они эту задачу блестяще решают. Тебе надо хотя бы проверить, а реально ли эта задача кем-то решена. Хоть как-то. С кем сражаемся-то. Вторая история — ты не хочешь заниматься переделыванием чужой работы, которая нормально сделана. Но и применять ее в текущем виде не получится — учились авторы немного на других данных, или у тебя появились новые данные, которыми модель можно улучшить. В этом случае применяется так называемый fine tuning, дообучение. То есть, ты берешь модель и доучиваешь ее решать твою задачу; иногда для этого достаточно просто взять предсказание этой модели, сверху взять как раз линейную регрессию, и всё прекрасно работает. Такое мы тоже делаем.
Ну и, наконец, третий вариант — бывает просто, что задачу решают плохо. И ты понимаешь, что ты, действительно, можешь лучше. И с нуля делаешь свою модель. А еще иногда бывает «комбо» — из чужих моделей, из своих и из чужих моделей, адаптированных к задаче.
Зигзаги карьеры биоинформатика
— Дмитрий, с какого времени вы работаете в команде Ивана Кулаковского ?
Там была смешная история. Я учился на факультете Биоинженерии и биоинформатики МГУ, меня восхищало машинное обучение с первого курса, и в какой-то момент я поступил учиться в чудесный проект «Техносфера» от Mail.ru. Летом я думал продолжить стажировку в Mail.ru, набраться там опыта, благо результаты обучения позволяли. Всё прошло прекрасно, но на финальном собеседовании я достаточно долго думал над очень простой задачкой, хотя в итоге ответил. И вот прошел месяц — мне не пишут, и я решил, что, значит, не судьба, недотянул. И тут буквально через четыре дня после «смирения с судьбой» Ваня Кулаковский написал объявление, по-моему, в чат ФББ, что они ищут человека, который хочет заниматься машинным обучением для регуляторной геномики. Мы созвонились, я согласился. Мы уже начали работать, а через две недели мне написали из Mail.ru, что меня ждут и уже оформили. Пришлось написать — ну простите, не получилось. Но вышло, в итоге, даже лучше, чем могло быть, потому что в итоге я остался в биологии, заниматься регуляторной геномикой.
Иван Кулаковский — канд. физ-мат наук, доктор биологических наук, в.н.с. Лаборатории системной биологии и вычислительной генетики ИОГен РАН.
— Когда вы пришли в команду Ивана Кулаковского, в каких проектах вы участвовали? FANTOM еще был?
Нет, FANTOM я уже не застал. Когда я пришел, был конкурс DREAM , 2016 года, причем я пришел, когда конкурс уже закончился.
FANTOM — международный мегапроект по функциональной аннотации геномов млекопитающих. В проекте FANTOM 5 участвовали биоинформатики Лаборатории системной биологии и вычислительной генетики ИОГен РАН под руководством чл.-корр. РАН Всеволода Макеева [10].
DREAM (Dialogue on Reverse Engineering Assessment and Methods) — проект по предсказанию сайтов связывания транскрипционных факторов.
— А, это тот конкурс, где у российской команды стал талисманом Чебурашка! Напомните, в чем там была задача?
Там задача была — использовать информацию о том, насколько доступен хроматин, и о самой нуклеотидной последовательности, чтобы предсказать связывание транскрипционного фактора. Просто доступность хроматина легко померить, а каждый транскрипционный фактор в каждом типе клеток изучить нереалистично долго и дорого, и у авторов конкурса была идея, что мы сейчас обучим модель, которая берет доступность хроматина, берет последовательность ДНК, и мы за счет этого получаем как бы, сразу много «виртуальных» ChIP-Seq , и дешево. В конкурсе участвовала команда Всеволода Макеева и Ивана Кулаковского, и они заняли первое место . Но потом был второй этап, где они первого места не заняли. На тот момент (почти 10 лет назад!) оказалось, что на этой задаче классическое машинное обучение работает даже лучше, чем глубокое. И мы поэтому долбили в этом направлении, но тогда ничего существенного не надолбили.
ChIP-Seq (Chromatin Immunoprecipitation followed by Sequencing) — метод анализа ДНК-белковых взаимодействий, основанный на иммунопреципитации хроматина (ChIP) и высокоэффективном секвенировании ДНК. Почитайте: «Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра» [11] и «Путешествие внутрь клеточного ядра, или Системная биология хроматина» [12].
Всеволод Юрьевич Макеев, чл.-корр. РАН, экс-руководитель Лаборатории системной биологии и вычислительной генетики ИОГен РАН (сегодня ИО зав. лабораторией Алексей Ступников).
«Биомолекула» освещала участие команды Кулаковского и Макеева в этом конкурсе: «Мечту вызывали?» [13]. — Ред.
А дальше был мой дипломный проект по конкурсу CAGI5. Это целая последовательность конкурсов, которые акцентируются на предсказания эффектов мутаций в геноме человека.
— Ну это, прямо, глобальная задача!
Да, но они ее делят. Там обычно есть задачи про белок-кодирующие мутации, и несколько конкурсов на предсказание эффектов некодирующих мутаций. И мы участвовали в конкурсе Saturation Mutagenesis, то есть насыщающий мутагенез. Идея там была следующая. Взяли, померили эффект однонуклеотидных мутаций в каких-то клинически значимых регуляторных регионах человека. Было известно, что в этих регионах мутации приводят к каким-то болезням, и гены, которые рядом находятся, действительно важны. Они регионы вырезали, и в каждом полученном регуляторном элементе просто померили эффект каждой отдельной мутации. Мы участвовали в этом конкурсе, и нас сильно подвело то, что мы акцентировались на классическом машинном обучении, мы думали, что сейчас придумаем хорошие признаки, подберем их под задачу, и решим всё. И в итоге мы выступили откровенно плохо.
Это было темой моего преддиплома, а темой диплома, как ни странно, было доказать, что проиграли мы, «потому что конкурсы у вас дурацкие». Ну, я утрирую, конечно. Мы проиграли абсолютно честно, у нас модель ужасная получилась, но прикол был в том, что, когда начали анализировать решение победителей, мы заметили очень странную вещь. У вас есть регуляторный участок, на нем надо учить эту модель, и можно выбрать разные кусочки внутри него, чтобы на них учиться. И мы увидели, что от того, как вы эти кусочки выбираете, получается очень разный результат. И на каком-то кусочке качество модели очень резко проседает. Авторы при выборе того, на чем будут учиться участники соревнования, так мелко нарубили регуляторную область, что это давало очень хороший результат. Но если просто взять и как-то поменять разбиение, то качество классификатора падает до 0,5, до случайного уровня.
При этом, если взять некоторые публичные данные, на которых можно было проверить модель, которая победила и была обучена на разбиении организаторов, то ее качество тоже оказывалось 0,5. Так что мы показали, что в этом CAGI5 всё было не так чисто. Самое смешное из того, что мы показали, — что на их разбиении можно взять признаки, которые не несут никакого биологического смысла, абсолютно случайные, просто построены по определенному правилу, и они дают вам очень хорошее качество.
— Интересный поворот для дипломной работы.
Да, мы очень разозлили организаторов. К ним потом наша статья попала, видимо, на рецензию, и была очень некорректная критика с их стороны. С игнорированием многих наших вычислительных экспериментов. Ну а потом я к Ване пошел в аспирантуру, и так с ним и работаю. В эпоху ковида я в преподавание подался. Не могу сказать, что это был негативный опыт, мне это нравилось, даже совместно с коллегами магистратуру открыли, но от научной работы отвлекало. Потому с какого-то момента я решил, что наука всё же веселее.
«Если ты побеждаешь в независимом конкурсе, для репутации это важнее, чем статья в Nature»
Следующий этап — это был конкурс DREAM 2022 года.
— Какая там была задача?
Участникам выдавали маленькие регуляторные последовательности длиной 80 нуклеотидов, их активность померили в специальных условиях, чтобы их можно было сравнивать. То есть, для одной последовательности активность 7,8, а для другой — 6,5, и мы уверены, что одна более активна, чем другая.
— Активность регуляторной последовательности определяется по экспрессии генов?
Да, но вот вопрос, как ее мерить. Они мерили специфическим образом, он называется «массовые параллельные эксперименты с репортерами» . Они использовали схему, которая позволяла тестировать много случайных последовательностей или геномных фрагментов. Маленький случайный кусочек в 80 нуклеотидов вставляется в плазмиду, там есть минимальный промотор, тут у них стоит GFP (зеленый флуоресцентный белок), а в другом месте — YFP (желтый флуоресцентный белок). Дальше эту плазмидку вставляют в клетку дрожжей с разными последовательностями. Затем их на клеточном сортере сортируют по светимости, по отношению светимости разных белков, и так замеряют активность. Задача была построить модель машинного обучения, которая вот по этим 80 нуклеотидам сможет предсказать их активность, а проверяли ее потом на разных подзадачах, в том числе на задаче оценки изменения активности при замене одного нуклеотида.
Массовый параллельный репортерный анализ (MPRA, massively parallel reporter assay) — это молекулярно-биологический метод, позволяющий одновременно тестировать тысячи или миллионы коротких фрагментов ДНК, чтобы определить, какие из них и с какой эффективностью действуют как регуляторные элементы.
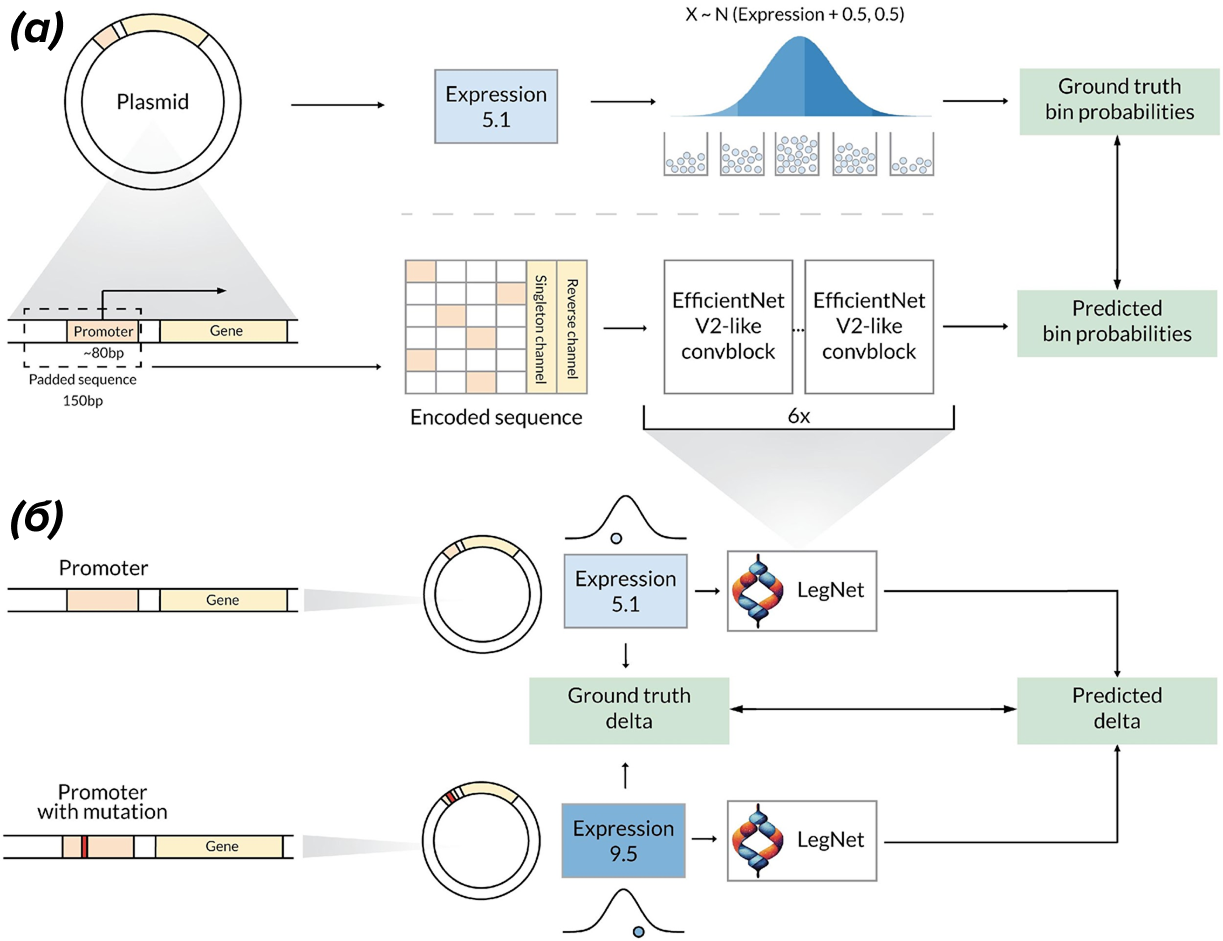
Рисунок 2. Обучение и прогнозирование экспрессии промотора и эффектов однонуклеотидных вариантов на основе массового параллельного репортерного анализа с помощью LegNet. (а) — общая схема процесса. Модель LegNet, основанная на нейросети для классификации изображений EfficientNetV2, обучается предсказывать вероятность попадания плазмиды с заданной регуляторной последовательностью в каждую из категорий активности. (б) — оценка эффекта варианта с помощью LegNet. Активность предсказывается для последовательностей с мутацией и без; эффектом мутации считается разница предсказаний.
Мы разработали свою модель, назвали ее LegNet (рис. 2), и она работала сильно лучше других. Она была сделана, по сути, с нуля, просто основываясь на том, что люди делают в обычном машинном обучении, не в биологии. И заняли первое место, причем с большим отрывом, нам еще до оглашения результатов конкурса организаторы написали, что у нас amazing performance. Потом оказалось, что даже часть подходов, которые мы предложили в своем решении, можно приложить к решениям других участников, и у них результат становится лучше. Мы эту историю далее продолжали, хотелось и свою статью написать, с акцентом на наших результатах [14]; затем организаторы конкурса написали статью, и мы пытались как-то эту статью сделать получше [15], а третья история была со статьей в Nature [16], о которой мы с вами прошлый раз говорили [17].
— Задача предсказания свойств регуляторных элементов по их последовательности, да? Где вы модифицировали вашу модель LegNet? Напомните эту историю.
Мы долго искали, где бы получше применить нашу модель, так как публикации только в Bioinformatics нам не хватало. А в своей статье организаторы «логично и законно» делали акцент на том, какие молодцы они. И вот мы увидели препринт статьи, где авторы для обработки результатов MPRA используют другую модель — совершенно непонятно, почему именно ее они взяли, потому что эта модель в конкурсе DREAM 2022 заняла почетное 20-е место из 25. И из результатов ее использования делают не совсем корректные выводы.
Мы удивились, обучили свою модель, быстренько адаптировали, всё проверили и написали пост в ныне запрещенной сети Twitter, что, ребята, всё, конечно, хорошо, только ваши выводы немножко не соответствуют действительности, и наша модель работает лучше, чем даже та модель, которая, как вы говорите, будет работать лучше за счет какой-то дополнительной информации. И вместо того, чтобы с нами ругаться в Twitter или просто проигнорировать, первый автор статьи Викрам Агарвал (Vikram Agarwal), замечательный человек, написал нам письмо: а давайте мы вас просто включим в статью, и там будет ваша модель. Но вам для этого, разумеется, надо сделать дополнительные анализы, потому что просто вашей одной картинки не хватает, нужно много еще поработать. И мы поработали, и сделали. Получилась с ними отличная коллаборация [16].
— А потом был конкурс Стэнфордского университета?
Да, с ним единственный нюанс — он был очень сторонним проектом и, по сути, не связан с тем, за что дали премию Сбера. Но да, сам конкурс был прикольный. В нем надо было предсказывать результаты эксперимента, который можно использовать для того, чтобы определить трехмерную структуру РНК. Я в этом участвовал отдельно от Вани, от всех взрослых коллег, вместе со своими студентами, и эти три месяца мы откровенно занимались тем, что никак не связано с регуляцией транскрипции (синтеза РНК), а влияет на пост-транскрипционную судьбу РНК. Сначала мы в этом конкурсе тоже пытались как-то наш LegNet адаптировать, но не получилось. И мы разработали другую модель, которую назвали в итоге ArmNet, ибо почему бы и нет? Это была тяжелая история, потому что там команд было даже больше, чем в DREAM. Кроме того, в DREAM не было денежного приза, а здесь он был, и поэтому люди боролись до последнего дня, но мы, в том числе за счет грамотного планирования, что и когда учить, как-то смогли победить.
В этой истории мы тоже разработали решение почти с нуля. Хотя нельзя сказать, что полностью с нуля, потому что почти в первый день конкурса было выложено публичное решение, которое давало некое стартовое качество. И мы поначалу опирались на него, хотя, стоит отметить, что это было, по сути, просто стандартное решение на основе Transformer. Мы это всё доработали, очень сильно видоизменили, добавили кучу историй, связанных именно с биологическими особенностями решаемой задачи, и добавили модели возможность использовать предсказания других биоинформатических алгоритмов.
Лично для меня главный выхлопом этого конкурса было, во-первых, то, что мы там победили, во-вторых, что мы действительно научились работать с этими архитектурами нового вида. А еще это был первый проект, когда за моей спиной никто не стоял, кроме того, он был явно перпендикулярен основной работе, а мы всё равно смогли победить.
Не только предсказание, но и дизайн
Когда умеешь предсказывать свойства последовательности, интересно научиться создавать последовательности с заданными свойствами. Люди в тот момент очень вдохновились историей, которая называется Diffusion — это способ обучить модель, которая генерирует картинки. Сейчас все это любят: ты задаешь текстовое описание, и она генерирует сценку. И то, на чем в тот момент начали строить эти модели, называлось Diffusion. И я увидел возможность — для нуклеотидных последовательностей никто этот подход на тот момент еще не применил. Мы можем быть первыми. И студентка под моим руководством, Лиза Аристова, начала работать над данным проектом.
— Почему Diffusion?
Потому что там процесс такой: у вас есть картинка в обучающем наборе, вы начинаете постепенно добавлять в нее нормальный шум. Чем больше шума добавляете, тем больше картинка неотличима от шума. Дальше ваша задача — обучить модель на каждом шаге игнорировать шум и возвращаться к незашумленной картинке. А дальше оказывается, что, если вы так обучили модель, можно специальной процедурой начать с шума и получить картинку. То, что я описал, позволяет генерировать картинки очень высокого качества. И люди от этого просто фанатели.
А я понял, что для нуклеотидов в последовательности это еще никто не применил. И очевидно, что кто-то применит, потому что свято место пусто не бывает. Почему бы это не быть нам? У нас же есть LеgNet, которая хорошо работает для предсказания активности регуляторных последовательностей. Давайте попробуем ее использовать, чтобы их дизайнить, то есть делать новые последовательности с заданной активностью. Когда вы ей даете случайную последовательность, а она должна вам выдать последовательность с заданной активностью. То есть она какие-то буковки поменяла, и вот выдала последовательность, в которой активность, скажем, 7,2. И мы этому ее обучили.
Но есть проблема — сигнал картинки можно представить как числа (к примеру, от 0 до 255), а нуклеотиды — это категориальные переменные, нет такого, что А больше Т. Поэтому нельзя добавлять нормальный шум. И мы сделали так: окей, давайте вместо нормального шума просто берем последовательность, у которой активность 8,9, вносим несколько одиночных случайных мутаций, а дальше просим модель: «Слушай, вот у этой последовательности была активность 8,9, мы внесли три мутации, пожалуйста, поменяй три нуклеотида и получи исходную последовательность». Оказывается, если так обучить, тоже потом получается, что можно стартовать со случайной последовательности и специальной процедурой получать последовательность с заданной активностью.
Конечно, пока мы всё адаптировали, пришлось очень много всего перепробовать. Бедная Лиза:) Во многом вспоминается анекдот про Брежнева и кур. Мы это опубликовали в той же статье, где и LеgNet, отдельно. У нас получилась первая статья [14], которая была опубликована в рецензируемом журнале про диффузию на нуклеотидных последовательностях. За счет того, что мы придумали очень простой способ, у нас получилось сделать это быстрее, чем у людей, которые пытались придумать теоретическое обоснование. Можно сказать, получился грязный метод, не имеющий статистического обоснования. Но по факту то, что мы сейчас видим: все эти методы, с обоснованием и без, которые были опубликованы после, генерируют приблизительно на одинаковом уровне. Потому у нас получился хороший метод.
Дальше встал вопрос: а что дальше? В случае с мРНК, конкретно – с мРНК-вакцинами, интересно генерировать последовательность не просто заданной активности, а чтобы они активно экспрессировались только в определенных группах клеток. Нам нужна клеточная специфичность. И, собственно, мы решили адаптировать нашу модель к данной задаче. За это взялись аспирант Вани Арсений Зинкевич и уже упомянутая студентка Лиза Аристова. В итоге было показано, что наша модель PARADE может генерировать последовательности, которые сильно экспрессируются в одной клеточной линии и слабо экспрессируются в другой. Более того, за счет того, что было имплементировано сразу несколько методов генерации, не только диффузия, получилось добиться большого разнообразия получаемых последовательностей.
Сейчас статья на рецензии [18]. К сожалению, здесь мы столкнулись то ли с халатностью части ревьюеров, то ли с конкурентами. Один ревьюер написал замечаний 30, часть из которых, я подозреваю, была сгенерирована нейросетью. Где-то замечания были просто фееричными: «Вы используете в своей статье метод X, а на самом деле есть еще новые методы Y, Z. Почему вы их не используете?». Хотя я точно знаю, что наш метод — один из самых новых для данной подзадачи. А дальше идешь по ссылкам, которые он дал, и понимаешь, что человек либо не читал, что ему выдала модель, либо осознанно валил. Потому что наш метод опубликован в 2019 году, а те ссылки, которые он дает, — 2016 и 2017 года. Причем в реальности это использованный нами метод X есть обобщение методов Y и Z. В итоге отбивание от рецензентов занимает много времени. Ибо (от других) есть замечания по делу, а есть вот такое вот. Тоже время отнимающее.
Вскочить в уходящий поезд или решить задачу тысячелетия?
— Откуда вы берете задачи, которые надо решать?
Самый простой способ — ты просто смотришь в область и видишь, какие есть нерешенные проблемы, пытаешься их решить. Или, как в случае с диффузией — какие есть нерешенные проблемы, которые еще не решали методом X. Второй вариант, если не угадал тренды до того, как они появились, — посмотреть, что люди сейчас публикуют. Можно пытаться запрыгнуть в вагон уходящего поезда. Та же работа с предсказанием структуры РНК, конечно, была достаточно новой задачей, но, во-первых, ее предложили другие люди нам в конкурсе, а во-вторых, кто-то уже ее делал. Казалось бы — зачем делать то, что другие уже сделали. Но ты прямо видишь, что люди делают хуже, поэтому ты можешь сделать лучше. Во-первых, иногда просто хочется сделать лучше, чтобы был нормальный метод, а не то, что обучили «на коленке», опубликовали в Nature, а использовать никто не сможет. Во-вторых, когда ты это делаешь, иногда получается emerging capabilities — что модель лучше другой на 20 процентов не просто за счет лучше выученных закономерностей, а за счет того, что учит какую-то другую, дополнительную биологию. И это действительно важно, что ты можешь дополнительно что-то интересное биологическое найти и исследовать.
Ну и третья история — это когда к тебе приходят коллеги и спрашивают: «А можно сделать это?». Иногда ответ: «Да, это кто-то делает». Очень часто ответ: «Это кто-то пытался делать, оно не работает. Давайте попробуем мы сделать». То есть получается комбинация первых двух. К тебе пришли люди, может, они сами придумали эту задачу — и это новая задача, которой в литературе нет. А может, они пришли с задачей, не зная, что она в литературе есть, а на самом деле, кто-то ее решает.
А бывает, что люди к тебе приходят, и это задача, которую все пытаются решить, но никто не может. Тогда это уже не вагон уходящего поезда, а, наоборот, задача тысячелетия. Я бы сказал, что таких задач в нашей области две, и они обе важны для клинической практики. Одна — это научиться предсказывать активность элемента тканеспецифично. А вторая, с ней связанная и более сложная, — это предсказать эффект индивидуальной мутации для конкретного человека. Вот у вас есть регуляторная мутация в каком-то геноме — какой у нее будет эффект на экспрессию генов вокруг, какой будет эффект на фенотипы? То есть, если у вас регуляторные варианты что-то рушат так, что они везде всё рушат, тогда и бог с ним, это, как правило, легко предсказать. А бывает, что они тканеспецифично рушат. Тогда у вас получается комбинация проблем: текущие модели мало того, что плохо работают с тканеспецифичными эффектами, они еще и плохо учитывают эффекты нуклеотидных мутаций.
Наши модели, обученные на MPRA, учитывают лучше, но и им далеко до клинической практики. Я подозреваю, что тот, кто эту задачу действительно решит, у него будет какой-нибудь AlphaGenomе10.
— А что нынешний AlphaGenomе?
Его разрабатывала та же компания, что и AlphaFold, и они пытались сделать такую нейросеть, что за нее тоже Нобелевку дадут. Но оказалось, что если AlphaFold отрывался от коллег в два раза, то AlphaGenomе там, дай бог, три процента где-то наскреб, ну, в одной задаче 10 процентов выбил. И что еще «веселее» — чем более задача близка к клинической, тем меньше получался прирост. Вот если хочется понять, за что будут давать Нобелевскую премию — читайте пресс-релиз AlphaGenomе. А далее можно прочитать советы по использованию AlphaGenome на гитхабе, где выложена модель, и понять, насколько мы все далеки от Нобелевки. Там очень большой разрыв. Причем, чем больше опыта в области и чем больше умеешь читать между строк — тем больше увидишь разрыв.
«Они на нас разозлились, но всё равно взяли в конкурс»
Да, я не рассказал еще историю с продолжением конкурса CAGI. Как я говорил, в CAGI5 мы проиграли, потом показали, что конкурс плохой. Но в этом году организаторы объявили очередной конкурс CAGI7. Там была история, похожая на CAGI5, но более качественная: они во всем геноме взяли регуляторные области, которые в исследованиях отмечены как регуляторные, и в этих областях набрали клинически значимых мутаций. И для каждой такой мутации померили ее эффект. У них была табличка: на такой-то хромосоме в такой-то позиции замена A на T приводит к уменьшению активности этого регуляторного элемента на 0,29, условно. Они дали информацию о мутациях в участках нескольких хромосом, а остальные хромосомы были скрыты от участников, и на них производилась оценка моделей. То есть у вас есть информация об эффектах мутаций в восьми хромосомах, вам нужно обучить на этом свою модель и сделать предсказания на оставшихся мутациях. А авторы конкурса оценят, насколько ваши модели хороши.
И мы в этом конкурсе решили участвовать. Причем после истории с CAGI5 у нас было два варианта: либо мы вообще не участвуем, либо, если участвуем, то надо побеждать. Прочитали условия — вроде сейчас конкурс правильно сделан, значит, надо побеждать. И мы с ребятами мутузили эту проблему как только могли. У нас получилось, что два студента внесли наибольший вклад (Ева Зубова и Алексей Василенко). И мне пришлось руками поработать. Эти четыре месяца в целом работали очень-очень много. Еще и организаторы продлевали конкурс три раза в последнюю ночь. После последнего продления Ева написала: «Два часа ночи, Дима прислал опять четыре варианта того, что надо протестировать. Живем. Когда-нибудь это закончится» (рис. 3). В итоге, наконец, конкурс закрыли, а вскоре оказалось, что мы победили.
Рисунок 3. «Я биоинформатик, я так вижу» — некоторые впечатления от участия в конкурсе CAGI
мем из личного канала студентки Евы Зубовой
— Это было недавно?
Седьмого декабря.
— Так у вас прямо свежая победа. Поздравляю!
О том, что мы победили, по идее, еще нет опубликованной информации, но это объявили на конференции по результатам конкурса. Там есть разные метрики нашего метода — есть корреляция, насколько то, что мы предсказываем, соответствует экспериментальной величине. А еще, насколько хорошо мы отличаем мутации, которые действительно обладают каким-то клиническим эффектом, то есть вредные, от невредных. Мы очень сложную процедуру разрабатывали, чтобы всё это правильно считать. В том числе, кстати, вдохновленную подходами из AlphaGenome. В итоге у нас качество, которое оценивается площадью под кривой точность–полнота, — 0,4, что очень много для этой задачи. У следующей команды 0,3, потом 0,28, ну а другие вообще плохо справились. Самое смешное: наши конкуренты, которые тоже на этих данных работают, заняли третье место. А второе место заняли люди, которые использовали нашу модель LegNet. Просто у нас была на руках доработанная нами же модель, совмещенная с опытом использования других моделей в области. Но, по сути, мы заняли и первое, и второе место.
— А там были еще российские команды?
Нет, на этот раз не было. С этим конкурсом CAGI есть большая проблема в том, как они распространяют информацию о нем. Они публикуют ее на своем сайте. Если ты увидел этот сайт — ты молодец, если не увидел... Так, чтобы просто сделать по-человечески объявления в Twitter и так далее, они, по-моему, додумались, только когда поняли, что... собственно, почему они дедлайн один раз перенесли — что команд откровенно мало собралось для участия. В общем, организация от боженьки. Но данные очень хорошие, они действительно в этот раз их по-человечески разбили, с ними было очень удобно работать, и было понятно, на что ты смотришь. В итоге лично для меня закольцевалась эта история — в CAGI5 мы нашли ошибку организаторов, но не победили, в CAGI7 победили. Причем с огромным отрывом от ближайших преследователей.
— А что вам будет за то, что вы победили? Публикация в крутом журнале?
Ну, как бы да, публикация. Но самое главное — репутация. Потому что есть миллион статей, которые рассказывают, что мы в регуляторных областях прям собаку съели и всё прекрасно предсказываем. Но тут как: «не важно, как проголосовали — важно, как подсчитали» — в своих статьях все герои. А если ты побеждаешь в независимом конкурсе, который оценивали третьи люди, не аффилированные с участниками, это куда более ценно. Также должна быть публикация с организаторами. Не совсем понятно, когда она выйдет, потому что они достаточно долго это делают. И с этим связана вторая проблема: мы хотим наш метод отдельно тоже опубликовать. Так что сейчас мы ждем разрешения использовать данные конкурса, потому что у нас много чего интересного получилось, и в том числе не связанного с конкурсом.
Что мне в этом конкурсе еще адски понравилось — эти данные используют все. CAGI5 с 2019 года, когда его опубликовали, использовался уже в миллиарде статей и будет продолжать использоваться. Данные CAGI7 еще лучше. И их, несомненно, все будут использовать. Значит, и наш метод тоже — как минимум для сравнения. И это очень полезная для видимости вещь. А еще мне понравилось, что датасет получился очень биологически интересный. На нем можно было проверить много гипотез, мы их проверили прямо в ходе соревнования, и получились очень нетривиальные результаты. И их было сложно заметить на других датасетах. На том же CAGI5 они воспроизводятся — но там их можно объяснить еще десятью способами. А здесь данные куда менее шумные.
Эти данные, в частности, помогают увидеть, что с моделями машинного обучения в области регуляторной геномики явно что-то происходит очень странное, и меня это сильно напрягает (в хорошем смысле — надо исследовать и исправлять причины). То, что мы видим на геномных моделях, в том числе на этом конкурсе, говорит о том, что эти модели работают вообще не так, как многие думают. Они явно в себя засовывают очень много какой-то лишней информации, которую чуть поломай — и их предположения, которые они используют, причем предположения там абсолютно дурацкие, нарушатся. И модель будет работать никак или в три раза хуже. И дальше возникают вопросы: первое, почему так? А второе, как эту модель использовать. Потому что ее можно применить к задаче десятью способами. Не умозрительными — в почти каждой статье, что я вижу, модель применяют еще с каким-то подвывертом.
При этом часто получается такая история, как с ЕГЭ по некоторым предметам: чем больше ты знаешь, тем хуже напишешь. То есть если ты биолог, ты понимаешь, как этот эксперимент проводится, ты все это учел при подаче в модель — ты молодец, конечно, держи шоколадную медальку, но у тебя модель выдаст худшее качество, чем если ты абсолютно дундук в этом плане. Так быть не должно. И очевидно, модель выучила что-то не то. На каких-то моделях мы такое поведение видим больше, на других — меньше. Но в целом история очень поучительная, и очень хочется это публиковать, хочется подискутировать с научным миром на эту тему. А пока авторы не опубликуют свои данные и не ответят мне на email, когда же они их опубликуют, мы сидим и ждем.
— Удачи вам. А 9 декабря вы получили Научную премию Сбера, с чем я вас еще раз поздравляю! Как она была организована? Туда подавались какие-то конкретные ваши работы?
Да, публикации. У них, насколько я понимаю, три этапа отбора. Предварительная фильтрация осуществляется очень большим количеством людей из науки, потому что много заявок. Потом идет этап с более сжатым экспертным советом. А потом последний этап, где уже финально выбирают победителя в каждой номинации.
— В вашей номинации «Artificial Intelligence в науках о жизни» большая была конкуренция?
Я знаю только, что на шесть номинаций было 700 заявок, а как оно дальше распределялось, мы не знаем. Конкурс точно был серьезный. Насколько он был серьезный, знают только в Сбере.
Немного о будущем геномики
— Вот такой вопрос. Вы используете возможности нейросети, решаете разные задачи и как бы добавляете кусочки пазла к общей картине геномной регуляции. А как вы считаете, можно ли предсказать, когда в науке будет полное понимание того, как работает геном? И вообще, эта задача осуществима?
Мне кажется, она точно осуществима. Вопрос, сколько нужно ресурсов для ее осуществления. Потому что даже про AlphaFold мы знаем, что он не всё решил, и нужны дополнительные эксперименты. И при этом для AlphaFold2 была сравнительно очень чистая база PDB (за утверждение о чистоте базы, меня, конечно, побьют структурные биологи), на которой можно было учиться. А в нашей области есть, конечно, результаты консорциумов ENCODE и FANTOM, где собраны какие-то эксперименты. Но… непонятно даже, сколько и какого рода экспериментов надо еще накопить, чтобы получилась история как с AlphaFold. Для того же ENCODE мы точно видим, что очень много данных там то ли избыточны, то ли модель выучивает только некое среднее этих данных, а больше выучить не может. А скорее играет роль комбинация этого.
ENCODE (энциклопедия элементов ДНК) — международный исследовательский консорциум с целью построения атласа функциональных элементов генома человека.
Поэтому я бы очень осторожно сказал, можно ожидать, что через 10 лет мы подвинемся в этой истории достаточно сильно. То, что мы точно сейчас знаем, что вот эти эмпирические эксперименты — массовые параллельные эксперименты с репортерами — они, как минимум, позволят нам работать с биотехнологиями и с дизайном мРНК-вакцин. Эта история, по-видимому, рабочая. И в комбинации с нейронными сетями на рубеже 5–10 лет, скорее всего, это очень сильно поможет в генной инженерии и генной терапии. Если делать какую-то терапию на этом уровне — замена регуляторных участков, целенаправленное внесение мутаций — в этом плане поможет. А вот до того, когда мы сможем просто взять геном конкретного индивида, загрузить его, и нам скажут все его предрасположенности и так далее, еще очень долго. Может быть, через 20 лет это решат, может быть, быстрее, может быть, медленнее. Опять же, это зависит от того, появятся ли принципиально новые методы экспериментальной оценки всего этого. Потому что постоянно придумывают новые методы — новые single-cell-эксперименты придумывают, недавно был придуман новый метод для измерения доступности хроматина. Хотя пока это proof of concept скорее методы, на них модели учатся, как я понял, не очень хорошо.
20 лет — это с условием, что будет происходить накопление данных и будут появляться какие-то эксперименты, позволяющие последовательно решать дополнительные задачи. А может быть, что-то бахнет, и окажется, что можно делать какой-то ультраэксперимент через пять лет, который по уровню сравним с тем, что научились секвенировать геном человека за $1000. Тогда будет просто: взяли тысячу разных клеток у человека, на каждой применили этот эксперимент, и дальше на этом учишь модельку, и все идеально. Такое может быть, но я пока к этому скептически настроен.
Тем более, что финансирование науки, в принципе, по миру переживает спад, достаточно тяжелый. Одним из ключей создания AlphaFold было такое объединение людей в международное сообщество в целом, и в соревновании CASP в частности, когда люди со всего мира пытались решить задачу предсказания структуры белков. Можно ли это делать в условиях, когда у каждой страны своя наука, и с другой наукой мы не взаимодействуем? Тут, опять же, надо понять, что это везде происходит, вот эта суверенизация науки. Не только в России, но и в Америке — сейчас на рассмотрении закон, чтобы всех ученых, которые взаимодействовали с русскими или китайскими учеными в течение пяти лет, лишить грантов. В итоге это реально приведет к тому, что у всех будет своя суверенная наука, и они изредка, обложившись десятью бумажками, все-таки будут друг с другом общаться.
— Очень грустная перспектива…
Если эта грустная перспектива не произойдет, то будет побыстрее. Если она произойдет, то я не представляю, как можно проблему уровня AlphaFold2 или AlphaGenomе решить на уровне одной страны любыми доступными средствами.
— Но, по крайней мере, международные конкурсы проходят.
Да, научное сообщество, по большей части, успешно сопротивляется, особенно если конкурсы организованы именно на деньги, которые дали научному сообществу и сказали — делайте, что надо. В ситуациях, когда спонсирует и организует фарма или похожее, проблемы есть. Например, есть организация Arc Institute — по сути, коммерческая инициатива, пусть так себя публично и не позиционирующая. Они проводили конкурс Virtual Cell Challenge, кажется. Там решали задачку с предсказанием результатов single-cell экспериментов, это близкая проблема к той, которую мы решаем. И вот они, в связи с какими-то регуляциями, которые накладываются на частных лиц, не могли выплачивать приз русским ученым, и более того, они запретили участвовать в конкурсе ученым из России, Ирана и так далее. А если конкурс организуют сами ученые, без участия частных компаний, то проблем чаще всего удается избегать.
Научпоп не всегда развивает критическое мышление
— И под конец хочу вас спросить про такую сторону вашей деятельности, которую можно назвать научным пранком. 1 апреля сего года вы с коллегами опубликовали на «Биомолекуле» «доклад младшего научного сотрудника Никиты Трофимовича Лисенко «Как ИИ способствует развитию современной науки» [19]. То, что это розыгрыш, помимо даты публикации, подсказывает ФИО докладчика. Но придумано, на самом деле, очень круто. Что вас сподвигло на это?
Начать надо с того, что я — один из главных админов паблика Labrats в Телеграме. В нем мы обыгрываем разные вещи, касающиеся биологической науки, в основном. Юмор у нас достаточно специфический, иногда довольно жесткий. Паблик появился в 2018 году, мы его сделали с моими студентами, которым я читал курс. Мы как-то сдружились и поняли, что нам всем нравятся научные мемы. И начали шутить про биоинформатику, про биоинженерию, про молекулярную биологию, в какой-то момент — про машинное обучение тоже. С какого-то момента паблик завирусился, так что нас многие теперь читают.
— Сколько у вас подписчиков?
15 тысяч. Так как у нас ниша ученых и студентов, получается достаточно много. Причем мы рекламу в основном не продаем, ибо в этой ситуации заказчик — барин, и это может вредить репутации. Потому рекламируем дружественные паблики и то, что нам самим интересно читать.
При этом у нас несколько админов, и разобраться, кто за что отвечает, людям со стороны, особенно пришедшим просто «прорекламироваться» — сложно. И вот написали мне сообщение — не могли бы связать нас с главным админом, хотим пригласить выступить на конференции, посвященной проблемам искусственного интеллекта? Я посмотрел на это и понял, что это так называемые инфоцыгане. Я, естественно, это перекинул в общий чат с вопросом: «Ребята, мы можем это просто проигнорировать, а можем... есть у кого-то желание выступить?». И админ, Никита, согласился в этом участвовать. Они ему скинули условия. И дальше помогло то, что Никита очень хорошо умеет торговаться. Потому что сначала они хотели, чтобы он еще и деньги заплатил, чтобы поучаствовать, но он сторговался за бесплатно. Дальше они у него спрашивают: «А как вас представить?». Мы вместе сочинили — Никита Трофимович Лисенко. Никита очень долго не хотел представляться так. Думал, что сейчас они загуглят, и вся наша веселая шутка закончится, потому что они поймут, что их стебут. Но нас спасло то, что они, по-видимому, абсолютно пропустили школьную программу, и кто такой Трофим Лысенко, они не знали. Это был такой тест, насколько они... Да, настолько. В общем, где-то на этом моменте мы уже поняли, что они все съедят, что мы дадим. И нам было заранее очень смешно. Это правда. И мы где-то три или четыре дня, если не неделю, сидели и писали шутки, которые надо в эту презентацию вставить, голосовали, что пойдет, что не пойдет, как это все сократить до десяти минут и так далее. И как это сделать звучащим максимально научно, но при этом абсолютно бредово для любого, кто вслушается и имеет хоть какой-то минимальный набор компетенций. Какие-то шутки мы даже исключали, потому что там было понятно, что Никита может и сам заржать в процессе выступления.
Мы это всё сварганили, и, забегая вперед, получилось даже лучше, чем мы ожидали. Потому что мы потом это пересылали адекватным людям, которые просто не знакомы с областью. И они сказали, что главной их ошибкой было, что они это слушали фоном, и когда слушали фоном, они не выкупали, что это очень жесткий стеб. Никита выступил с этим докладом, там было четыре других докладчика, которые рассказывали, как они используют ИИ в постоянной жизни. И Никита Трофимович Лисенко наш. Мы все думали, что кто-то хотя бы из тех, кто смотрит это, выкупит. Кто-то выкупал, отчасти, наши подписчики, прошедшие по ссылке на трансляцию, но организаторам это не сильно помогало, они не понимали по-прежнему, что над ними стебутся.
— Смешно. А был ли в этой истории какой-то просветительский момент?
В нулевые—десятые годы набрала обороты история, вера отчасти даже, что сейчас у нас из научной и околонаучной среды выйдет звездная плеяда популяризаторов, расскажет людям про все тайны мира, и люди резко начнут во всем разбираться. Критически мыслить в любой ситуации и, разумеется, ни за что и никогда не покупать у шарлатанов мифические препараты. Называлась эта идеология и, вроде, называется «просветительством». Мое же мнение, и мнение админов Labrats, как людей, испорченных работой в науке, что если ты не специалист в области, то просмотр научпопа поможет тебе развлечься и, может быть, если научпоп очень качественный (такого мало, ибо может исходить исключительно от людей, работающих в науке активно, или от журналистов, с ними тесно общающимися при составлении материала), сформировать очень примерную картину того, что происходит в области. Но далеко не факт, что это у тебя разовьет критическое мышление. Ибо, в отличие от мира розовых пони, в реальности критическое мышление очень жестко привязано и зависит от вашего знания области. И ученые это прекрасно понимают — недаром почти любая статья (во всяком случае, по естественным наукам) делается усилиями десятков людей, являющихся экспертами в своих узких нишах.
Мы посылали выступление Лисенко людям, у которых, несомненно, критическое мышление есть, просто оно развито в своей области, они это слушали вполуха и далеко не сразу понимали, в чем нюанс. Если им сказать: «Посмотри, тут есть подвох», наверное, они подвох бы заметили. И то, по их же признанию, не все. Но даже так — мы же в жизни очень редко въедливо воспринимаем каждую крупицу информации. Никаких ресурсов не хватит. Поэтому у нашего Лисенко был своего рода «антипросветительский посыл» — в реальных ситуациях обращайтесь к экспертам, не думайте, что по каким-то трем роликам в интернете вы всё узнаете. Силами человека из области или даже просто получившего высшее образование по области X затереть лютейшую дичь про эту область можно с очень высокой эффективностью — и прослушивание популяризации поможет очень слабо. А при условии, что сами «звездные» популяризаторы склонны всё упрощать — иногда можно их же материалы для одурачивания и использовать.
— Вы продолжили дело «Корчевателя».
Ну, «Корчеватель» Гельфанда стал очень известным мемом. Мы до этого уровня еще не дошли. Но да, может быть, появится еще Никита Лисенко, может, его еще куда-то позовут, и будет вторая итерация, посмотрим.
— Ну, успехов ему в его нелегком труде. И вам успехов — в исследованиях и в следующих конкурсах!
«Полнота — вещь очень относительная и вряд ли достижимая»
Мы также задали несколько вопросов научному руководителю Дмитрия Пензара, доктору биологических наук Ивану Кулаковскому.
— Как вы считаете, в чем секрет успеха российских бионформатиков? У вашей команды целая коллекция выигранных международных конкурсов. И вообще, российские бионформатики всегда выглядят очень достойно на международном уровне. По сравнению, например, с экспериментальной генетикой, которая испытывает большие сложности. Чем это можно объяснить?
Тут много факторов. Во-первых, очень талантливые люди — увлеченные педагоги и ученые — вложились в то, чтобы построить в России биоинформатическое образование. Сейчас это стало уже мейнстримом, но импульс был еще в девяностые, что вылилось, в частности, в создание Факультета биоинженерии и биоинформатики МГУ в 2002. Об этом написал Михаил Гельфанд в статье [20], где, помимо взвешенной критики неприлично-знаменитого мусорного обзора по истории биоинформатики в России, есть и ключевой абзац про становление биоинформатического образования в России. И, конечно, кроме этого, уже была сильная математическая и айтишная школа. Оказалось, что есть хорошие кадры — те, кто знает статистику, умеет хорошо программировать, интересуются какими-то применениями и развитием вычислительных алгоритмов. И притом этим людям скучно было идти в бизнес и не хотелось заниматься абстрактной математикой. А тут такая замечательная прикладная область, которая, благодаря развитию высокопроизводительных экспериментальных методов, стремительно обзавелась потоком «больших данных». И вот так удачно сложилось, что люди, которые хотели в этом направлении развиваться, нашли своим навыкам новое полезное применение, теперь это не только математика-IT-физика, но и «компьютерная биология» .
См., например: «Я б в биоинформатики пошёл, пусть меня научат!» [21] и «12 методов в картинках: „сухая“ биология» [22].
Кроме того, к сожалению или к счастью, биоинформатика была и остается существенно дешевле, чем экспериментальная молекулярная биология. Анализом последовательностей и моделированием успешно занимались и до того, но момент резкого взлета приходится, условно, на начало 2000-х годов (окончание проекта «Геном человека» [23] и стремительный рост доступных для анализа молекулярно-биологических данных [24]). Появилось действительно новое поле, в котором можно было без больших финансовых вложений (например, анализируя большие и кем-то еще опубликованные данные) делать хорошие работы. Оказалось, что есть квалифицированные люди, есть растущая предметная область, да еще и дешево можно этим заниматься. Как замечательно! Сейчас, конечно, и получать большие данные дорого, и обсчитывать большие данные дорого, то есть, если раньше разрыв в стоимости с экспериментальной наукой был астрономический, то сейчас он потихонечку сокращается. Оборудование для обучения нейросетей — большая статья расходов, а долгосрочное хранение данных секвенирования скоро (если не уже) будет дороже, чем непосредственно секвенирование.
— Ваша команда активно работает с зарубежными коллегами, вы участвуете в международных конкурсах. Испытываете ли вы сейчас трудности с международным научным общением? Изменилось ли что-то с 2022-го года?
К сожалению, да. Можно сказать две вещи. Первое, что люди увлеченные, и с этой стороны, и с той стороны, конечно, контакты сохранили и пытаются их поддерживать. В этом смысле, персональное общение, оно так быстро не разрывается. Но есть какие-то формальные и бюрократические критерии. Например, большой конкурс проводили наши знакомые коллеги из Университета Санта-Круз в Калифорнии. Серьезный конкурс, крупные коммерческие партнеры вложили в организацию какие-то свои ресурсы. И в этом прекрасном мероприятии (сайт которого не открывается с российских IP адресов), очень современном, интересном, на предсказание ответа клеток на удаление генов, было просто запрещено участвовать из России. Прямо на старте. Ну и нет никакого смысла участвовать как-то подпольно, поэтому мы не участвуем вообще. Итого: стало, конечно, сложнее поддерживать связи. И этот процесс неостановимый с обеих сторон. Конечно, хочется дожить до точки, когда все эти связи, которые сейчас начинают рушиться, будут восстанавливаться. Хочется дожить до этого момента. Планы такие есть.
— Да, мы все этого очень хотим. И последний вопрос — я его задавала Дмитрию, и вам тоже хочу задать. Вы с помощью нейросетей решаете разные задачи в области геномной регуляции. А по вашему мнению, когда в науке будет полное понимание того, как работает геном?
Мы недавно с коллегами спорили, можно вообще говорить в современной биологии, что какой-то список или ресурс, например, полный. Можно заявить, что мы знаем все гены у какого-то организма, и что больше их список не будет меняться? Но вот выясняется, что нет. Находятся какие-то кусочки, которые тоже зачем-то нужны. Гены это или не гены, если они все-таки прочитываются? Или наоборот — известные гены переводят из одной категории в другую. Например, оказывается, что это какая-нибудь ошибочная копия или просто плохо аннотированный кусочек генома. То есть, к сожалению, полнота — вещь очень относительная и вряд ли достижимая. Что такое «полностью понять», трудно сказать.
Мне больше нравятся конструктивные вопросы. Вот какие задачи мы бы хотели решать? Мы бы хотели рационально конструировать управляющие последовательности с нужными свойствами, чтобы хорошо работал нужный ген и включался в нужных тканях или типах клеток. Можно это сегодня сделать? Можно. Можно это было сделать 10 лет назад? Ну, с большим трудом и ограничениями, и проще было взять кусочки готовые из генома и как-то переиспользовать, да и это тоже не всегда работало. То есть, в принципе, за 10–20 лет прогресс в том, чтобы искусственно конструировать полезные для таких биотехнологических задач последовательности, произошел колоссальный и радикальный. То есть, по сути, решена неразрешимая раньше задача. Так же и с белками. Сворачивать белки — нельзя сказать, что задача полностью решена, что больше не надо этим заниматься. Но достигнут прогресс, который тоже еще недавно казался невозможным. И в этом смысле, если не каждый год, но каждые пять лет приносят какие-то невероятные новости. Вот, например, с реконструированием генома древнего волка, то, что сделала компания Colossal [25]. Это, с одной стороны, «обман», потому что это реконструирование не генома и не биологического вида, а яркого фенотипа. С другой стороны — это демонстрация того, что мы уже можем сделать инструментами геномного редактирования. И это красиво.
Ну и, наконец, биологические организмы и системы прекрасны и тем, что постоянно меняются. Отчасти поэтому биология неисчерпаема — мы тут изучаем организм в деталях, это процесс небыстрый, а пока его изучаем, тут уже и клетки сами «отрастили» себе очередной новый молекулярный механизм.
Литература
- Sergey Abramov, Alexandr Boytsov, Daria Bykova, Dmitry D. Penzar, Ivan Yevshin, et. al.. (2021). Landscape of allele-specific transcription factor binding in the human genome. Nat Commun. 12;
- Фальшивые мотивы в ДНК: как геномные варианты меняют поведение транскрипционных факторов;
- Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024);
- AlphaFold: нейросеть для предсказания структуры белков от британских ученых;
- Белковые галлюцинации: как справляется AlphaFold?;
- От структуры к функции: революция ИИ в белковом дизайне;
- История развития искусственного интеллекта и его пришествия в биологию;
- Как языковые модели покорили мир белков;
- Johannes C Hingerl, Alexander Karollus, Julien Gagneur. (2025). Flashzoi: an enhanced Borzoi for accelerated genomic analysis. Bioinformatics. 41;
- PCR: «FANTOM. Молекулярный зоопарк»;
- Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра;
- Путешествие внутрь клеточного ядра, или Системная биология хроматина;
- Мечту вызывали?;
- Dmitry Penzar, Daria Nogina, Elizaveta Noskova, Arsenii Zinkevich, Georgy Meshcheryakov, et. al.. (2023). LegNet: a best-in-class deep learning model for short DNA regulatory regions. Bioinformatics. 39;
- Abdul Muntakim Rafi, Daria Nogina, Dmitry Penzar, Dohoon Lee, Danyeong Lee, et. al.. (2025). A community effort to optimize sequence-based deep learning models of gene regulation. Nat Biotechnol. 43, 1373-1383;
- Vikram Agarwal, Fumitaka Inoue, Max Schubach, Dmitry Penzar, Beth K. Martin, et. al.. (2025). Massively parallel characterization of transcriptional regulatory elements. Nature. 639, 411-420;
- PCR: «Активность регуляторных элементов в геноме человека научились точно предсказывать по их нуклеотидной последовательности»;
- Matvei Khoroshkin, Arsenii Zinkevich, Elizaveta Aristova, Hassan Yousefi, Sean B. Lee, et. al. A generative framework for enhanced cell-type specificity in rationally designed mRNAs — Cold Spring Harbor Laboratory;
- ИИ и генетика: как российские нейросети учатся читать ДНК и создают персонализированную медицину;
- Mikhail S Gelfand. (2025). On “Bioinformatics in Russia: history and present-day landscape” by M.A. Nawaz, I.E. Pamirsky, and K.S. Golokhvast. Briefings in Bioinformatics. 26;
- Я б в биоинформатики пошёл, пусть меня научат!;
- 12 методов в картинках: «сухая» биология;
- Геном человека: как это было и как это будет;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Фантасты в лабораторных халатах: как безумные идеи превратились в бизнес будущего.











