
От структуры к функции: революция ИИ в белковом дизайне
08 декабря 2025
От структуры к функции: революция ИИ в белковом дизайне
- 288
- 0
- 5
Генеративный дизайн белков.
Рисунок в полном размере.
иллюстрация автора, созданная с помощью DALL·E 3
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Белки — это молекулярные машины, отточенные эволюцией на протяжении сотен миллионов и даже миллиардов лет. Мы пока не способны воспроизвести этот длинный путь естественной эволюции, но уже активно учимся направлено конструировать белки под конкретные задачи. И помогают нам в этом большие массивы данных и искусственный интеллект (ИИ). Сначала он научился предсказывать трехмерную структуру природных белков, а теперь на очереди — дизайн: создание новых функций и уверенная оптимизация уже существующих. В статье разбираем, как генеративные модели придумывают форму будущего белка, подбирают к ней аминокислотную последовательность и как вместе с лабораторными экспериментами проверяется, что такой белок действительно работает.
Конкурс «Био/Мол/Текст»-2025/2026
Эта работа опубликована в спецноминации «Искусственный интеллект в биологии» конкурса «Био/Мол/Текст»-2025/2026.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Партнер номинации — компания Центральный университет в лице совместной с институтом AIRI Лаборатории ИИ, анализа данных и моделирования.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Эволюция на протяжении миллионов лет скрупулезно настраивала работу белков, подгоняя их форму и функцию под нужды клетки. Но ее нужды — это довольно ограниченный набор задач. Человеческие же запросы куда экзотичнее. Нам нужны катализаторы для синтеза лекарств в органических растворителях; ферменты, стабильно работающие при 70–80 °C или в кислых средах; высокоспецифичные байндеры (связывающие белки) и лиганды; а также белки, разлагающие стойкие техногенные соединения — вроде пластиков, появившихся в биосфере лишь с развитием химической промышленности. Неудивительно, что далеко не под все такие задачи у естественного отбора вообще был запрос: часть нужных нам реакций в природе попросту не катализируется, а многие природные ферменты вне своих родных условий почти не работают.
Для решения этой проблемы биохимики еще в конце прошлого века начали переносить дарвиновскую эволюцию в пробирку. Сначала из гена исходного фермента делают гигантскую библиотеку мутантов: с помощью ПЦР [1] с искусственно выкрученной на максимум ошибочностью копирования вносят точечные мутации и произвольно рекомбинируют фрагменты. Иногда к ПЦР добавляют короткие ДНК-заготовки с заменами в целевой позиции, и копирование идет уже с ними (так исследователи могут немного управлять мутагенезом) [2]. Затем полученные варианты экспрессируют и отбирают те, которые работают чуть лучше. Лучшие мутанты попадают в следующий раунд: их снова мутируют, снова тестируют и повторяют этот цикл, пока улучшения не прекратятся [3].
Не случайно Нобелевская премия по химии 2018 года досталась Франсис Арнольд за направленную эволюцию ферментов и Джорджу Смиту и Грегори Уинтеру — за фаговый дисплей пептидов и антител — по сути, еще один вариант направленной эволюции, только примененный к байндерам. К этому моменту направленная эволюция уже стала обычным способом улучшить белок [4].
Но у направленной эволюции есть и серьезные минусы. Чтобы вытащить из случайных библиотек несколько по-настоящему удачных вариантов, приходится проверять десятки миллионов и более мутантов, при этом выстраивая сложные и дорогие схемы отбора. И даже после этого активность новых ферментов не дотягивает до природных [3]. Направленная эволюция трудоемкая и медленная, а поиск, по сути, идет только по близким к оригиналу последовательностям и в конце может привести в некий «эволюционный» тупик, из которого практически невозможно выбраться малыми мутациями.
Поэтому логичным следующим шагом становится попытка перенести эволюцию из пробирки в компьютер.
Первая серия: компьютерный дизайн Rosetta
Еще до того, как в белковом дизайне появились диффузионные и графовые нейросети, трансформеры и языковые модели, у исследователей уже было вполне работающее «конструкторское бюро» — классический компьютерный дизайн с помощью Rosetta, которую уже более двух десятилетий развивает лаборатория Дэвида Бейкера в Вашингтоне. Очень подробный разбор этого мира есть на «Биомолекуле» в статье «Конструкторское бюро белков» [5].
Если кратко, то Rosetta — это, по сути, алгоритм Монте-Карло «на стероидах» [6]. Программа берет исходную структуру белка и многократно вносит случайные небольшие изменения по одному и тому же набору заранее запрограммированных действий: поворачивает связи, гнет остов (каркас белка) и перестраивает боковые цепи. А в режиме дизайна белков к этим движениям добавляются еще и мутации [5].
Такие изменения выбираются случайно, но не абы как: генератор не предлагает совсем уж бессмысленные шаги. Например, гидрофильные аминокислоты располагаются преимущественно на внешней стороне (в соответствии с принципами фолдинга глобулярных белков [7], [8]), а гидрофобные на внутренней — такую управляемую случайность в Rosetta называют «умной рандомизацией». После каждого шага пересчитывается энергия получившейся структуры: «хорошие» шаги, понижающие энергию, она оставляет, «плохие» — отбрасывает . Повторяя этот цикл тысячи раз, Rosetta строит всё более правдоподобные конформации и последовательности, а затем отбирает те, которые выглядят энергетически наиболее выгодными [5].
Или всё же принимает, но с вероятностью, уменьшающейся пропорционально разнице энергий: такой принцип в методах Монте Карло называется критерием Метрополиса [9].
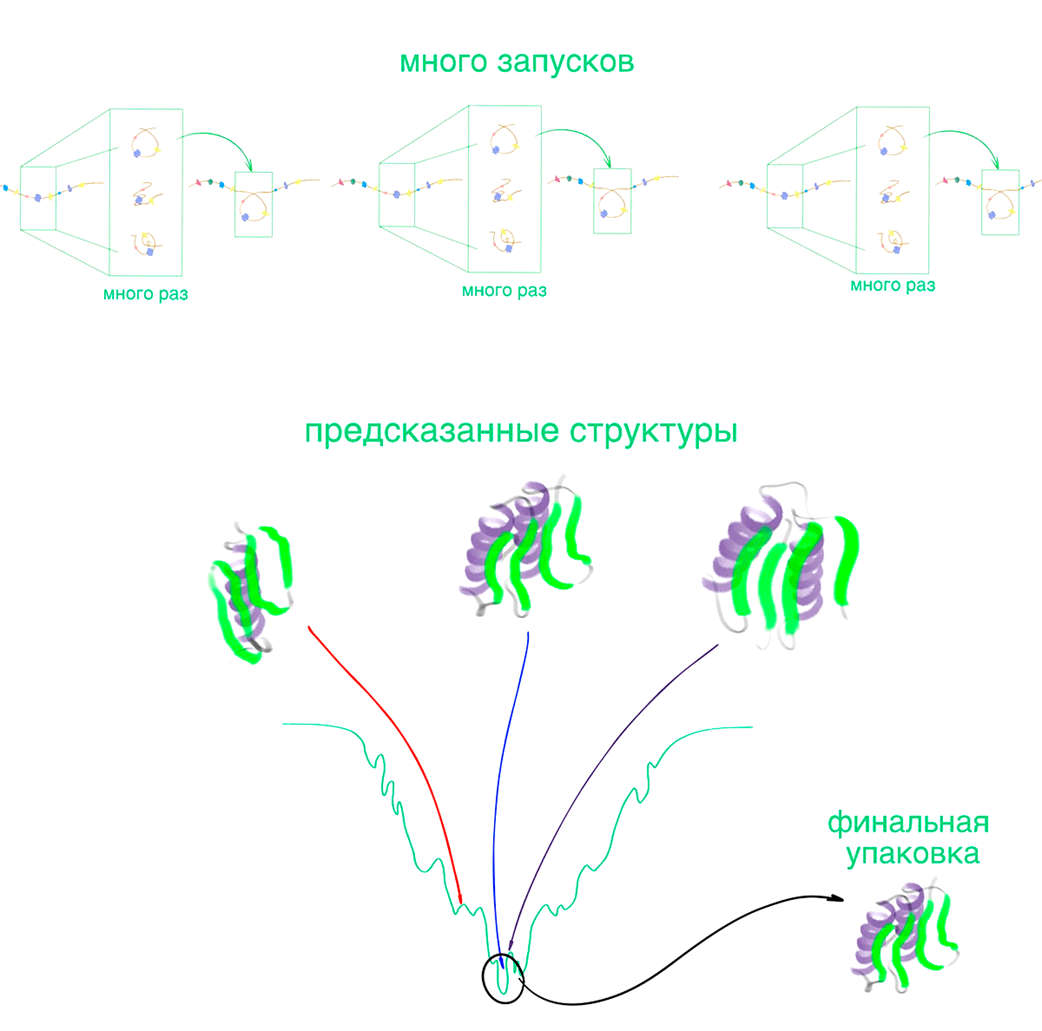
Рисунок 1. Алгоритм Rosetta: многократный запуск «умного» поиска Монте Карло порождает множество кандидатных структур белка. Затем эти варианты сортируются по энергии, и из дна энергетического ландшафта отбирается наиболее стабильно упакованная конформация.
рисунок Васили Сулеймановой из статьи «Конструкторское бюро белков» [5]
Еще в 2003 году группа Дэвида Бейкера спроектировала Top7 — небольшой белок с новой упаковкой, который в эксперименте сложился почти так же, как в компьютере [10]. Позже с помощью Rosetta спроектировали десятки дизайнерских белков. Среди них — искусственные ферменты (например, KumaMax), минибелки-ингибиторы гемагглютинина как лекарство от гриппа, биосенсоры на малые молекулы, «замок—защелка—ключ» для контроля экспрессии, светящиеся репортёры на заказ, белки-менеджеры для работы с метаболизмом, адаптеры для CAR-T [5], [11]. Подробнее про них можно почитать в статье «Конструкторское бюро белков» [5].
В качестве более раннего, еще до эпохи Rosetta, примера de novo дизайна часто вспоминают альбебетин — искусственный белок с заранее заданной трехмерной структурой, который российская группа спроектировала практически «на коленке» с помощью теории структуры белков, буквально карандашом на бумаге, задолго до эры суперкомпьютеров [10].
В 2024 году за развитие вычислительного дизайна белков Дэвид Бейкер получил половину Нобелевской премии по химии. Подробно за что именно — можно (и нужно) прочитать на «Биомолекуле» в статье «Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024)» [10].
На момент 2020 года, если оглянуться на все достижения Rosetta, складывалось весьма оптимистичное ощущение: «С ее помощью ученым удалось справиться с целиакией, создать лекарство широкого спектра от вируса гриппа и научить иммунные клетки находить и убивать раковые» [5]. И это действительно впечатляет! Кажется, что найдено решение для большинства биотехнологических прихотей. Но это не волшебная палочка, а тяжелый инженерный инструмент: мощный, но далеко не всегда доводящий красивый in silico дизайн до рабочего лекарства или фермента.
Расчеты энергии Rosetta неизбежно упрощают реальную физику, а жестко фиксированный каркас плохо учитывает гибкость белка и различные конформации. В результате подавляющее большинство in silico дизайнов в пробирке либо вообще не сворачивается, либо оказывается плохо растворимым и нестабильным, а у немногих, выживших после проверки в пробирке (обычно это менее 0,1%), каталитическая активность и аффинность обычно далеки от природных. Лаборатории компенсируют это «грубой силой» — отбором по большим библиотеками из сотен тысяч конструкций и парой раундов направленной эволюции вокруг удачных зернышек.
И даже если конкретный дизайн проходит через это сито и в пробирке выглядит почти образцовым — стабильный, активный, хорошо работающий на модельных субстратах, — это все равно только середина пути: впереди устойчивость в организме, фармакокинетика, токсичность, иммуногенность и неожиданные сюрпризы на клинических стадиях.
На уровне биохимии KumaMax (потенциальное лекарство от целиакии) выглядел как демонстрационный успех Rosetta: в фазе I фермент реально переваривал 97–99% глютена в желудке у людей [5], [12]. Но в фазе II все оказалось гораздо менее эффектным: по гистологии слизистая в группе фермента выглядела даже хуже. Вместо того, чтобы обезвредить глютен, пациенты получили, по сути, мощный иммуногенный белок: так, в группе принимавших KumaMax, антитела нашли у 90% участников, причем у большинства ответ оказался стойким. До «лекарства от целиакии» эта красивая конструкция так и не дошла.
Похожая судьба постигла и некоторые другие проекты Rosetta. И дело здесь не в том, что с самими конструкциями или с Rosetta что-то не так — напротив, уже тот факт, что искусственные белки вообще работают и иногда работают очень хорошо, сам по себе впечатляет. Причина в том, что подавляющее большинство кандидатов в лекарства, независимо от платформы, отсеивается на поздних стадиях клиники или по бизнес-соображениям.
Так, минибайндеры к гемагглютинину обеспечивали у мышей вполне приличную защиту от гриппа, но их разработка так и не дошла до клиники и реального препарата. Антиамилоидные мини-белки в пробирке и на животных моделях хорошо тормозят рост амилоидных фибрилл [13], однако таким конструкциям все равно нужно как-то попасть в мозг через гематоэнцефалический барьер, который пропускает в мозг менее 0,1% циркулирующих белков [14].
Есть и более удачный пример. В вакцине против коронавируса SKYCovione используют небольшую белковую наночастицу, заранее спроектированную в Rosetta, к которой пришит фрагмент S-белка коронавируса. В 2022 году такую вакцину одобрили в Южной Корее и внесли в список ВОЗ, так что это один из первых зарегистрированных препаратов, где de novo-спроектированный белок служит носителем антигена [15].
Кроме того, многие из этих конструкций уже стали лабораторными инструментами: флуоресцентные β-бочки превратились в метки для визуализации, белковые переключатели управляют экспрессией и логикой сигналов (система «замок—защелка—ключ») в клеточных моделях и CAR-T у мышей [5], [16].
Также далеко не к любой мишени вообще удается подобрать хороший байндер. Если целевой участок слишком плоский или требует неудобной ориентации, компактный белок просто не может одновременно хорошо упаковываться и дотянуться до нужной поверхности. А так как Rosetta жестко фиксирует мишень, то шанс на успешный дизайн падает почти до нуля.
И все же именно Rosetta впервые показала, что белок, целиком спроектированный в компьютере, в реальности может складываться, катализировать незнакомые природе реакции и даже лечь в основу вакцинной наночастицы, дошедшей до препарата. Да, у подхода есть ограничения и порой мучительный клинический путь, но именно Rosetta первой показала, что с белками можно обращаться как с объектами инженерного дизайна [5], [10].
Тем не менее, смоделировать эволюцию и «настоящую» физику одновременно очень сложно: либо мы грубо упрощаем расчеты, либо теряемся в астрономическом количестве последовательностей. Поэтому следующий шаг был почти неизбежен — сместить акцент с физики Rosetta на статистику: пытаться с помощью моделей ИИ учиться у самой эволюции на больших массивах белковых данных.
Вторая серия: эпоха ИИ — от предсказания формы к ее генерации
Переход от физики к статистике начался не сразу с генерации новых белков, а с более скромной задачи — научиться предсказывать форму уже существующих. Первым громким героем здесь стал AlphaFold: нейросеть, которая по одной аминокислотной последовательности выдает трехмерную структуру с точностью, сравнимой с экспериментом [17]. После успеха AlphaFold2 предсказание структуры для многих хорошо упорядоченных белков превратилось из отдельного проекта на месяцы работы в повседневный инструмент: им пользуются, чтобы анализировать последствия мутаций, собирать комплексы, прикидывать, где могут быть карманы и гибкие петли. Не случайно вторую половину Нобелевской премии по химии 2024 года за предсказание структуры белков с помощью AlphaFold получили Демис Хассабис и Джон Джампер . Подробно про то, как ИИ ворвался в мир белков, «Биомолекула» уже рассказывала в спецпроекте «ИИ в биологии» [18].
В интернете есть классные документльный фильм, посвященный Хассабису, и видео-подкаст с участием Джампера (оба — выпущены DeepMind), где они рассказывают о своем пути и гордости за то, что их творение стало одним из основных рабочих инструментов биоинформатиков по всему миру.
При этом первые поколения AlphaFold-подобных моделей решали прежде всего «прямую» задачу: по последовательности построить наиболее правдоподобную структуру [17]. Если запустить предсказание несколько раз, мы почти всегда получаем один и тот же детерминированный снимок молекулы с небольшими вариациями, а не целый спектр возможных форм. Для дизайна это удобно как строгий фильтр качества, но не очень подходит, когда хочется не только проверять готовые последовательности, но и придумывать новые [18].
Логичным следующим шагом стало научиться решать задачу в обратную сторону: не только спрашивать «какая форма у этой последовательности?», но и «какую форму вообще можно придумать под наши условия?» — отсюда и новая волна генеративных моделей для белков [18]. Здесь в игру входят диффузионные модели — те самые, которые рисуют картинки по тексту, только вместо пикселей у них координаты атомов. Модель, стартуя с хаотичного шума, шаг за шагом восстанавливает из него реалистичные остовы (каркасы) белка. Каждый запуск начинается с нового «шумного облака» атомов, поэтому одна и та же сеть может порождать целое семейство правдоподобных каркасов, а не один и тот же вариант.
Так из разных нейросетевых моделей собирается конвейер генеративного дизайна белков. Обычно он состоит из трех основных шагов. Сначала диффузионная модель (мы рассмотрим RFdiffusion) генерирует подходящий остов белка под нашу задачу (например, с нужным карманом или мотивом). Затем модель вроде ProteinMPNN заселяет остов аминокислотами так, чтобы последовательность надежно сворачивалась именно в эту форму. И, наконец, предсказатель структуры вроде AlphaFold проверяет, что получившийся белок действительно складывается так, как было задумано.
На практике это уже целый ансамбль нейросетей: мы комбинируем разные модели между собой, переиспользуем их архитектурные блоки и идеи, при необходимости подмешиваем классические подходы направленной эволюции и физического расчета — в сумме все это заметно повышает шансы на успешный дизайн.
На фоне такого разнообразия инструментов для начала давайте разберемся с диффузионными моделями для белкового дизайна — например, RFdiffusion.
RFdiffusion: Скульптор формы из шума
RFdiffusion — диффузионная модель, которая работает по тем же принципам, что и другие диффузионные модели. Поэтому, чтобы проще понять, что делает RFdiffusion, сначала посмотрим на те самые диффузионные ИИ, которые генерируют картинки в интернете.
В диффузионных моделях вся магия происходит с одним большим вектором — длинным списком чисел. В этом векторе в сжатом виде хранится текущее состояние объекта. Для картинок это латент — в нем хранится информация о содержимом будущего изображения (грубо говоря, вектор содержит большое множество чисел-предшественников пикселей в виде матрицы). В RFdiffusion вместо латента сразу будут готовые координаты белкового остова.
Теперь представим наш большой вектор как точку на векторном поле (рис. 2). В каждой точке поля нарисована стрелка — она показывает, в какую сторону нужно сдвинуться, чтобы результат стал более правдоподобным. В начале генерации наш вектор заполнен шумом, и точка случайно попадает куда-то на это поле. После этого мы много раз подряд прогоняем ее через одну и ту же нейросеть-«денойзер». На каждом шаге сеть слегка меняет числа в векторе — а значит, чуть-чуть сдвигает точку по этому полю. С каждым таким шагом точка все ближе подбирается к области, где живут реалистичные картинки или физически правдоподобные белки: траектория постепенно по стрелочкам идет к какому-то правдоподобному положению.
Шаги диффузии пронумерованы, и на каждом шаге сеть учитывает текущий номер t (это число подмешивается в сеть как отдельный аргумент). Из-за этого меняются вычисления нейросети, а за ними и векторное поле (рис. 2Б). В начале пути поле «крутое»: стрелки длинные и все направлены в центр — сеть делает грубые движения, быстро убирая грубый шум. Ближе к финалу поле выравнивается, шаги становятся маленькими, и сеть уже не перекраивает структуру, а лишь аккуратно шлифует детали (рис. 2Б).
Сама по себе диффузионная модель умеет делать только одно: двигать точку от абсолютного шума к какому-то реалистичному объекту. Если ее ни о чем не просить, она просто блуждает среди реалистичных структур.
Чтобы это превращалось в управляемый дизайн, мы добавляем условия (и получаем conditional diffusion): симметрию, форму и т.д. На каждом шаге сеть получает не только зашумленное состояние и номер шага t, но и это условие. Внутри модели условие сначала кодируется в отдельный вектор, а потом используется, чтобы подтягивать движения основного вектора по полю в сторону выполнения этого условия.
Без условий сеть задает базовое векторное поле: «куда двигаться, чтобы прийти к любому правдоподобному белку». Это отражает ее априорный «вкус» — какие структуры она в принципе считает реалистичными. Если добавить условие «симметрия» или «форма», к базовому шагу добавляется еще один вектор — поправка в сторону условия. Взятые вместе, эти два вклада ведут вектор и к реализму, и к нужным свойствам (рис. 4А) [19].
Чем больше коэффициент поправки на условие, тем сильнее вклад условия: тем агрессивнее «точка-вектор» выталкивается в сторону заданных свойств (стрелки от условий как бы перевешивают базовое поле и активнее тянут вектор к условию), иногда ценой разнообразия. За это отвечает популярный прием guidance (управление). Точно так же можно складывать несколько условий: текст, картинку-подсказку, маску областей и т.д.
Некоторые условия можно задавать мягко (форма, симметрия) — через guidance, то есть добавляя к движению по полю аккуратное «подталкивание» в сторону нужных структур. Например, если мы хотим получить симметричный олигомер, мы подаем в модель симметричный шум и добавляем поправки на условия, которые мягко стягивают траекторию к симметричным конфигурациям (рис. 4).
Однако мягкого условия часто оказывается недостаточно: нам важно не только наметить общую форму структуры, но и жестко зафиксировать конкретный фрагмент. Так устроено, например, проектирование байндеров к заранее заданным участкам мишени или белков с определенным мотивом.
Диффузионная модель по определению работает со всей структурой сразу. Если просто предоставить ей свободу, она будет пытаться восстановить структуру целиком, включая мотив или мишень, и постепенно начнет искажать их. Поэтому соответствующие атомы фиксируемого участка просто перезаписывают каждый раз заданными координатами.
Решают эту проблему методом Монте-Карло (SMC-Diff — Sequential Monte Carlo Diffusion): запускают множество диффузионных траекторий, генерируют несколько вариантов остова и отбирают только те, которые хорошо стыкуются с мотивом без разрывов и геометрических артефактов.
В итоге комбинация мягкого условия и жесткой фиксации мотива превращает хаотический процесс диффузии в управляемый процесс генерации структур, которые одновременно и реалистичны, и удовлетворяют всем заданным условиям (рис. 5) [20].
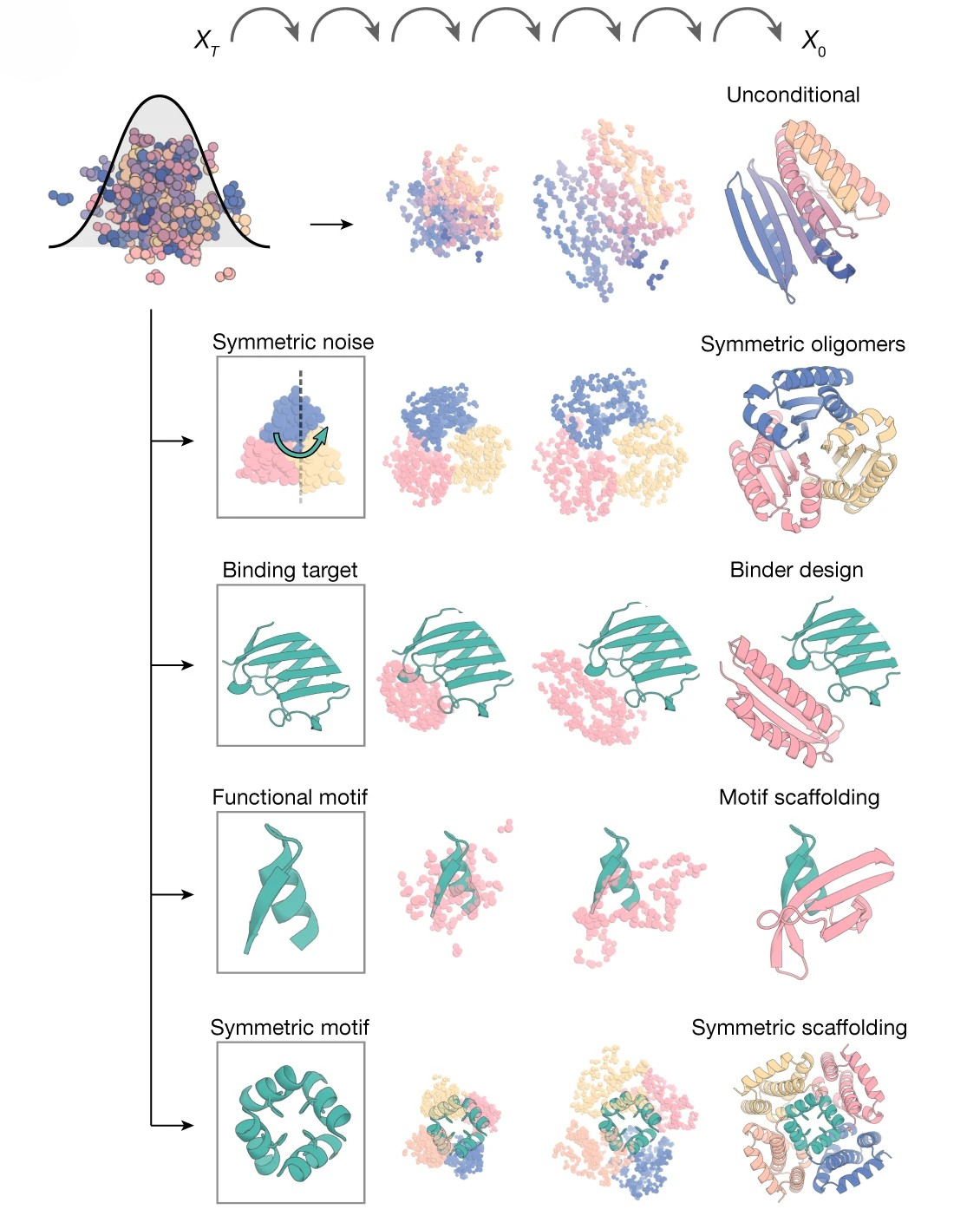
Рисунок 5. Различные способы управления генерацией RFdiffusion. Сверху показана генерация без заданных условий: шаг за шагом собирается произвольный белок. Ниже — один и тот же диффузионный процесс под разными условиями. Когда задают симметрию, модель получает симметричный шум и дополнительные вектора-поправки, которые стягивают генерацию к симметричным олигомерам. Если нужно встроить структуру мишени или функционального мотива (зеленые фрагменты), эти фрагменты жестко фиксируются на каждом шаге, а диффузия постепенно формирует вокруг них новый каркас. Для симметричного мотива оба приема просто складываются: фиксированный мотив и симметрия. Этим и хорош хаотический процесс диффузии, что им можно управлять множеством различных способов: можно сохранить мотив, встроить его из другого белка, сразу вырезать карман под определенную молекулу (лиганд, ион металла или даже другой белок) или задать симметрию и форму.
Похожим образом в качестве условия можно «прибить гвоздями» не только целый мотив, но и отдельные каталитические остатки и их функциональные группы, расположенные вокруг переходного состояния молекулы, вплоть до атомарного описания активного центра (рис. 6) На этом принципе построена RFdiffusion2: по заданной конфигурации таких групп она подбирает каркас фермента, который удерживает эту геометрию с атомарной точностью [21].
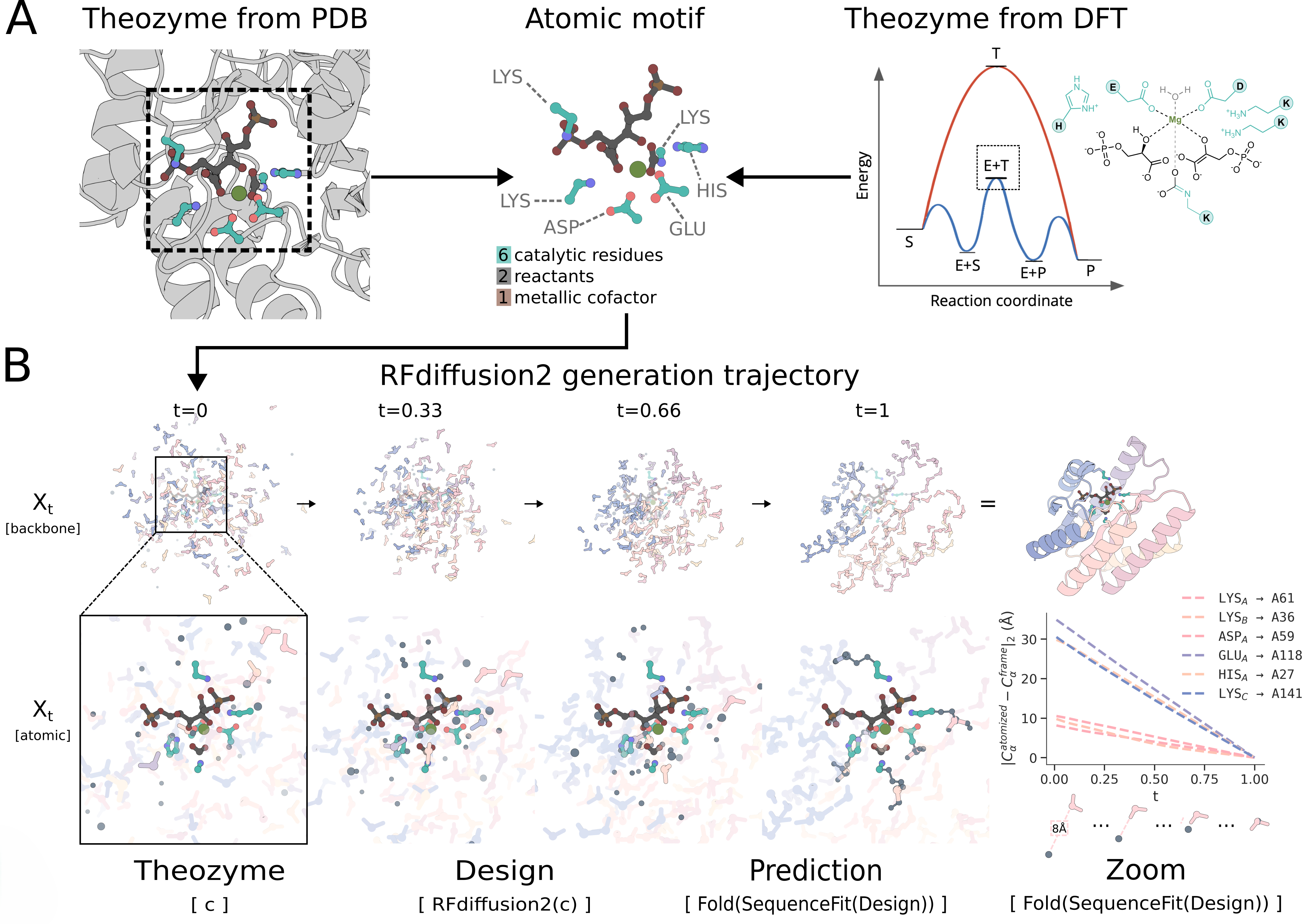
Рисунок 6. Проектирование фермента по атомарному описанию активного центра в RFdiffusion2 (а) — каталитический теозим извлекают из PDB-файла. Фиксируют расположение остатков активного центра (atomic motif). (б): слева — исходный теозим, используемый как условие [c]; в центре — один из дизайнов, сгенерированных RFdiffusion2(c); справа — предсказанная структура Fold(SequenceFit(Design)). График справа показывает, как по мере хода диффузии расстояния между каталитическими атомами и остатками дизайненного белка уменьшаются и, по сути, сходятся без разрывов и сумасшедших углов. Видно, как по мере диффузии Xt [backbone] постепенно расшумляется, а заданные остатки активного центра Xt [atomic] не меняются на протяжении всего процесса.
В RFdiffusion в роли денойзера выступает модифицированный RoseTTAFold2, который Минки Бэк с коллегами разработали в группе Бейкера. RoseTTAFold2 по устройству очень близок к AlphaFold2 (рис. 7) [22], но здесь его используют как специальный блок для очистки структуры от шума.
На вход ему дают пустую аминокислотную последовательность и только зашумленные координаты остова, поэтому выравнивание последовательностей (MSA) здесь просто не работает — нечего выравнивать. Модель смотрит на это хаотичное облако остова и пытается предсказать более правдоподобную структуру, то есть сдвинуть наш большой вектор по векторному полю в сторону реалистичного белка.
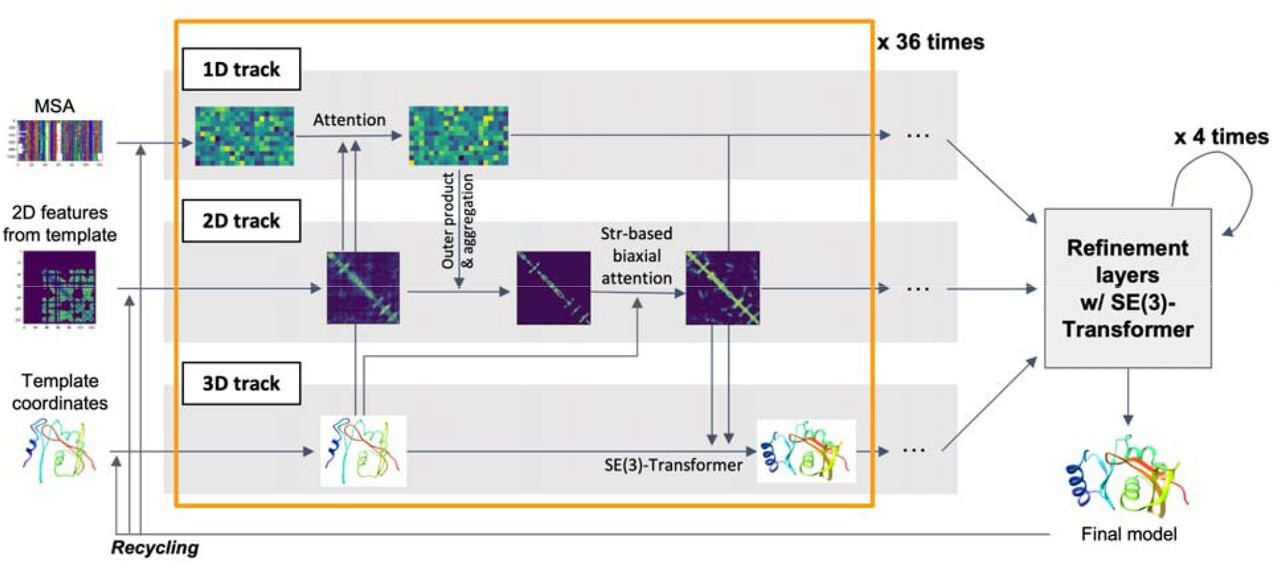
Рисунок 7. Архитектура RoseTTAFold2. Создатели RFdiffusion прямо вдохновлялись идеями AlphaFold2, поэтому внутреннее устройство RoseTTAFold2 во многом напоминает его: трехтрековая архитектура (1D-трек с MSA, 2D-трек с попарными расстояниями и ориентациями, 3D-трек с координатами остова), несколько проходов recycling. Обучалась модель не только на экспериментальных структурах, но и на предсказаниях AlphaFold2 (distillation). Качество локального расположения фрагментов вокруг каждого остатка контролируют через лосс FAPE (Frame Aligned Point Error) — тот же прием, что и в AlphaFold2: он заставляет сеть особенно тщательно согласовывать относительное положение фрагментов, а не только «в среднем» попадать в структуру. 2D-трек обновляется за счет структурно-зависимого biaxial attention, а 3D-трек — через SE(3)-Transformer, эквивариантный аналог IPA: при повороте входной молекулы все ее координаты на выходе поворачиваются вместе с ней.
Дальше после каждого шага денойзинга RoseTTAFold2 к предсказанным координатам специально добавляют небольшой случайный шум. На векторном поле это выглядит как случайные прыжки точки после каждого шага (на рис. 8А: голубая линия — правка RoseTTAFold2, серая — смещение из-за шума).
Зачем это нужно? Благодаря этим смещениям результат зависит не только от самого начального шума, но и от случайностей вдоль всей траектории: при одних и тех же условиях мы получаем целое семейство разных структур, а не одну-единственную. Если убрать внесение шума, то любая точка будет скатываться в центр векторного поля (см рис. 8Б).
Амплитуда вносимого шума зависит от шага t: в начале траектории шум заметный и помогает растрясти структуру и исследовать разные варианты структуры, что повышает разнообразие. Ближе к концу траектории шум почти исчезает, оставляя только мелкие точечные правки, которые дошлифовывают уже вполне правдоподобную конформацию.
В целом вся процедура выглядит так: остов, полученный на данном шаге, слегка зашумляют и снова отправляют в RoseTTAFold2. Чем ближе к концу траектории, тем меньше шума и тем короче шаги, которыми сеть правит координаты. Шаг за шагом шумное облако блоков остова превращается в стабильную структуру (рис. 9).
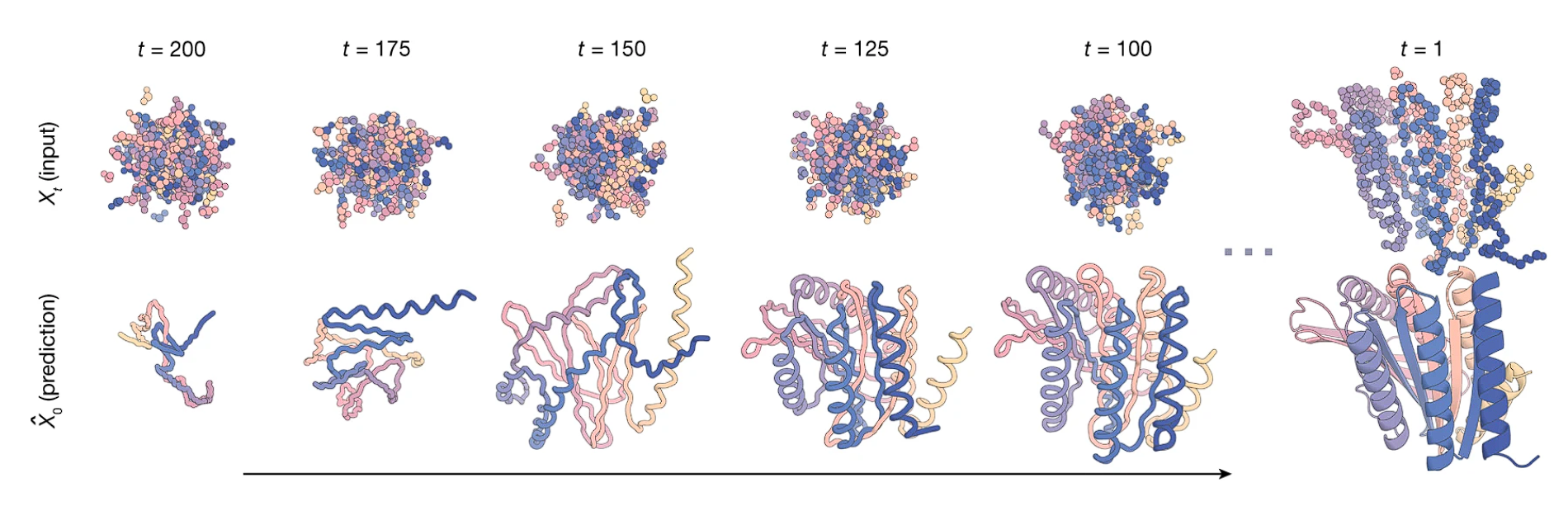
Рисунок 9. Работа RFdiffusion: на каждом шаге предсказывается правдоподобная структура белка X̂0 (prediction), сразу вносится шум и возвращается в RoseTTAFold в качестве Xt (input).
Чтобы все это не оставалось чистой абстракцией, на рис. 10А—В показано, как RFdiffusion шаг за шагом «выковывает» байндеры из шума — сначала для совсем простой мишени, потом для реального рецептора.
Те же диффузионные идеи уже нашли место и в классических моделях предсказания структуры. Прием «взять зашумленный объект и шаг за шагом его очищать» сегодня используют не только в дизайнерских моделях вроде RFdiffusion, но и в современных предсказателях, например, в AlphaFold3, где диффузионный модуль занял место старого структурного блока на базе Invariant Point Attention (IPA) из AlphaFold2. Поскольку диффузионные нейросети более гибкие, это позволяет более точно восстанавливать атомную структуру не только белков, но и молекул самых разных химических классов [23], [24].
Диффузия перестала быть чисто «игрушкой для de novo-дизайна»: это уже общая парадигма генерации трехмерных координат белков. Один и тот же принцип можно использовать и чтобы придумать новый фолд, и чтобы собрать структуру для известного семейства по заданной последовательности и эволюционному сигналу из MSA.
Важно, что на всех этих картинках RFdiffusion занимается исключительно формой. Модель генерирует остов, но не знает, какие именно аминокислоты там будут. Такие белки еще не существуют: это геометрические заготовки, абстрактные фолды, где вместо аминокислот пустые маски — то есть пока только геометрия, без конкретной последовательности.
Это было осознанное ограничение первого поколения диффузионных генераторов вроде RFdiffusion. Вместо того чтобы сразу решать все — и форму, и последовательность, и химию активного центра, — команда Бэйкера сфокусировалась на самой тяжелой геометрической части пайплайна: придумать новый фолд под заданные условия. Последовательности, энергии и тонкая химия отданы на откуп другим моделям, которые подключаются на следующем шаге дизайна.
ProteinMPNN: заселяем каркас аминокислотами
На предыдущем шаге RFdiffusion сгенерировала нам «голый» остов белка — каркас основной цепи и петель без конкретных аминокислот. Теперь нужно сделать обратную операцию: подобрать такую последовательность аминокислот, которая будет надежно складываться именно в этот каркас. Этот тип задач называют обратным фолдингом. В прямом фолдинге по последовательности мы предсказываем, как реальный белок свернется в трехмерную структуру [25], а при обратном, наоборот, к уже заданной форме подбираем подходящую последовательность (рис. 11) [26].
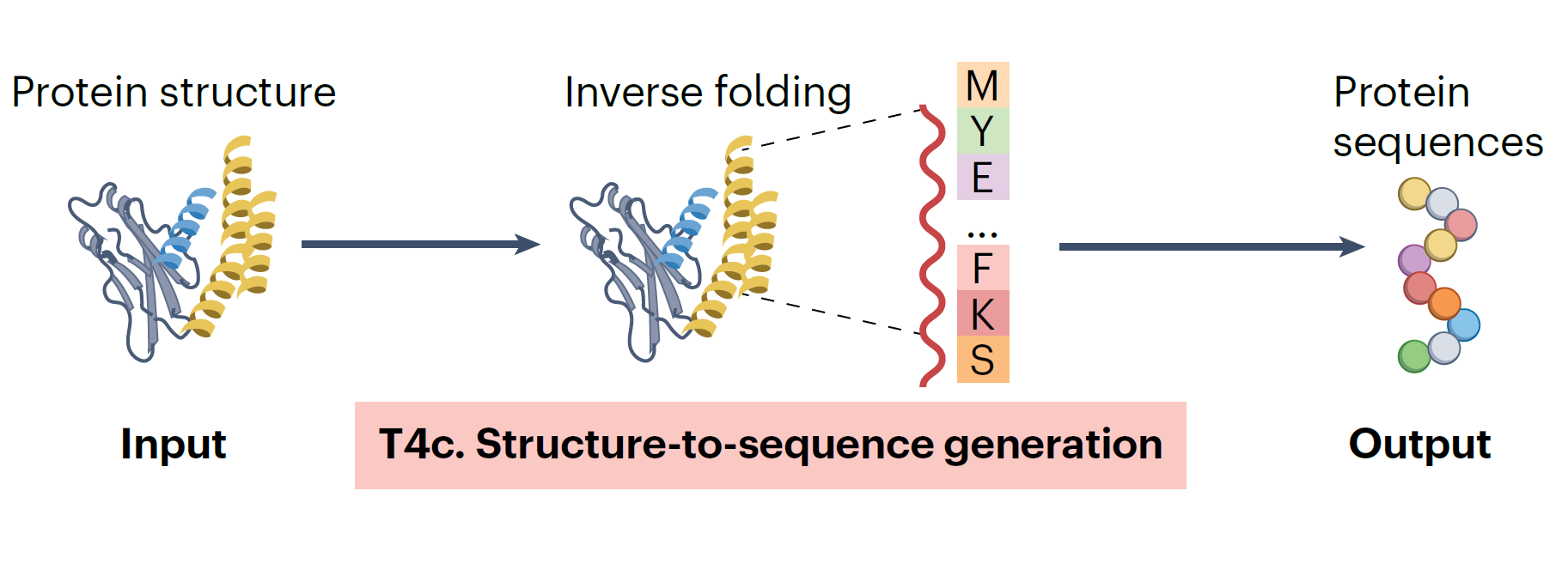
Рисунок 11. Обратный фолдинг (inverse folding): по заданной структуре (protein structure) модели генерируют подходящие аминокислотные последовательности (protein sequences).
До эпохи нейросетей обратный фолдинг осуществляли с помощью Rosetta [5]: перебирали мутации, считали энергии и пытались, насколько позволяет упрощенная физика, подобрать «энергетически выгодную» последовательность. Такой подход работал, но был тяжелым вычислительно и часто давал конструкции, которые на компьютере выглядели идеально, а в пробирке не сворачивались или оказывались нестабильными.
В Институте дизайна белков в Сиэтле Дэвид Бейкер вместе с Юстасом Даупарасом, Ильей Анищенко и коллегами пошли другим путем и разработали ProteinMPNN — нейросетевую модель, которая решает проблему обратного фолдинга статистически, опираясь на реальные структуры. Модель обучали на десятках тысяч экспериментально определенных пар «готовая структура белка → его природная аминокислотная последовательность» [27].
По сути, ProteinMPNN выучивает негласные правила эволюции: какие аминокислоты обычно сидят внутри гидрофобного ядра, какие комбинации остатков стабилизируют ɑ-спираль, а какие — β-листы, как выглядят типичные швы между доменами и контакты между цепями. Благодаря этому ей не нужно явно считать энергетику и вращать каждую боковую цепь — просто генерируют целую библиотеку таких вариантов и in silico отбирают несколько лучших по метрикам нейросети.
До появления ProteinMPNN лаборатория Бейкера за годы работы с Rosetta накопила целое кладбище неудачных каркасов, которые смотрелись прекрасно на компьютере, но плохо экспрессировались, не сворачивались или оказывались нестабильными в эксперименте. В то же время, ProteinMPNN гораздо лучше восстанавливает последовательность, чем Rosetta — в среднем около 52% аминокислотных позиций против примерно 33% у Rosetta (эти проценты — sequence recovery) [27].
Пропустив такие «проблемные» остовы — мономеры, циклические гомоолигомеры, наночастицы, байндеры — через ProteinMPNN, оказалось, что значительная часть этих каркасов была вполне жизнеспособна: новые последовательности лучше экспрессировались и сворачивались в заданную форму. Иными словами, старые геометрические идеи оказались хорошими, проблема была именно в последовательности — и нейросеть смогла подобрать более удачные варианты [27].
AlphaFold: рентген без рентгена
Однако нейросеть все еще остается лишь алгоритмом и вполне может ошибаться. Чтобы не проводить месяцы в лаборатории, тестируя десятки неудачных конструкций, следующий шаг мы тоже частично переносим в in silico. AlphaFold настолько дешево и быстро отбрасывает плохие варианты, что сегодня почти ни один проект по дизайну белков не обходится без такой in silico-проверки [28]. Неудивительно, что именно за решение задачи надежного предсказания структуры белков с помощью AlphaFold в 2024 году Демис Хассабис и Джон Джампер получили Нобелевскую премию по химии [10].
Внутреннее устройство AlphaFold2 уже хорошо разобрано на «Биомолекуле» в статье «Как языковые модели покорили мир белков» [18] (хоть AF — это и не языковая модель), поэтому здесь напомним лишь основную идею (рис. 13). Сначала модель ищет эволюционных «родственников» белка и подходящие структурные шаблоны, а затем переводит все это в два внутренних представления: MSA (множественное выравнивание последовательностей) и pair representation, где кодируются расстояния и относительные ориентации остатков. Затем блок Evoformer многократно гоняет информацию между этими двумя треками, уточняя, какие пары аминокислот должны оказаться рядом. Наконец, структурный модуль на базе Invariant Point Attention (IPA) подбирает координаты атомов, согласуя их с предсказанными картами расстояний и ориентаций, и выдает итоговую 3D-структуру вместе с оценками уверенности. (Одновременно напомним, что наиболее современная версия программы — AlphaFold 3 — работает уже во многом иначе, в частности, вместо IPA используя диффузионный структурный модуль.)
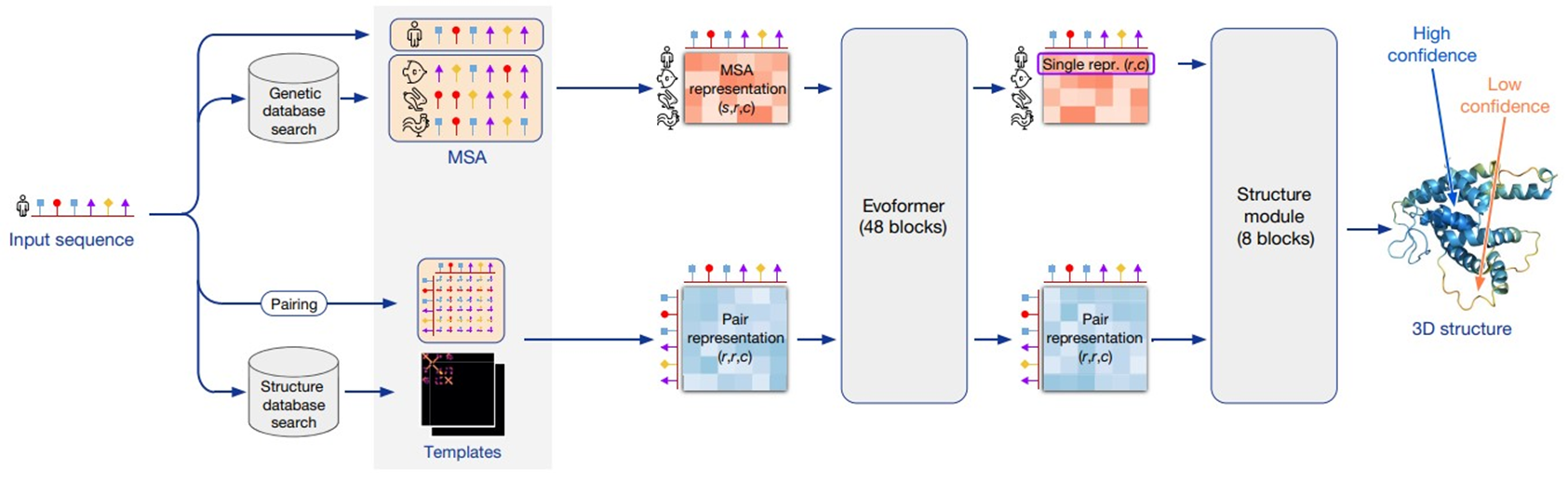
Рисунок 13. Устройство AlphaFold2. Входная последовательность по результатам поиска гомологов и структурных шаблонов превращается в два трека — MSA- и pair-репрезентации, которые многократно обновляются в блоках Evoformer и затем подаются в структурный модуль IPA, предсказывающий 3D-структуру и метрики уверенности (синий — высокая уверенность, оранжевый — низкая).
Когда мы говорим о проверке дизайнов, нас интересуют именно эти оценки уверенности, которые модель возвращает вместе со структурой (рис. 14). Каждый остаток получает оценку уверенности локальных расстояний pLDDT от 0 до 100, а для пар остатков строится карта ожидаемой ошибки PAE (predicted alignment error). Если в дизайне несколько цепей, дополнительно появляются интерфейсные метрики — например, показатели уверенности во взаимном расположении цепей (pTM) и качестве межцепочечных контактов (ipTM).
Если предсказанная структура в целом совпадает с каркасом RFdiffusion, а зоны белка выглядят «синими» по шкале уверенности (рис. 14), такой кандидат идет дальше; если остов упрямо складывается во что-то иное или метрики на интерфейсе проваливаются — последовательность отправляется в корзину.
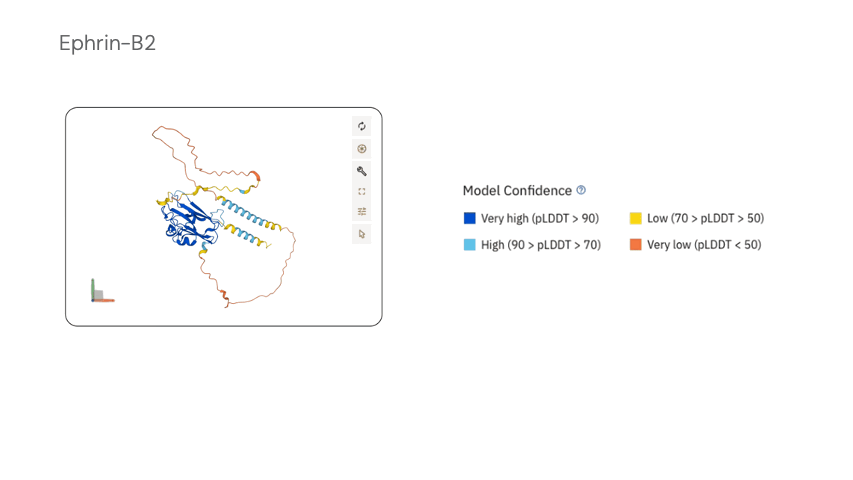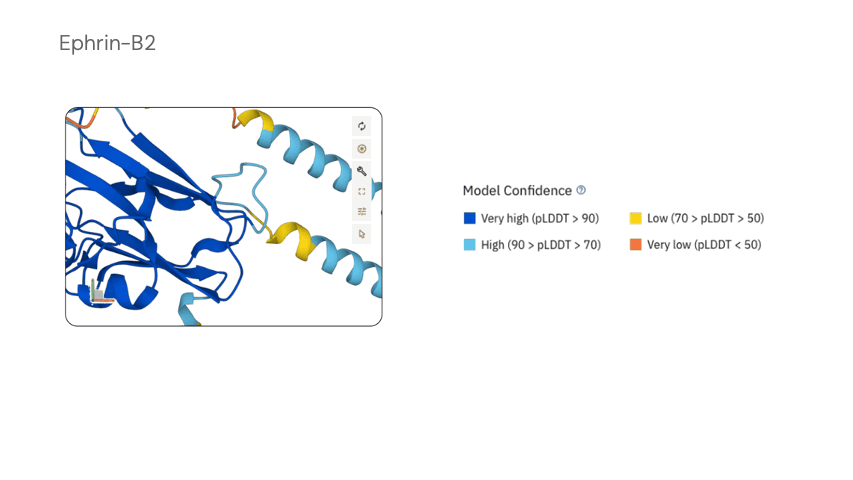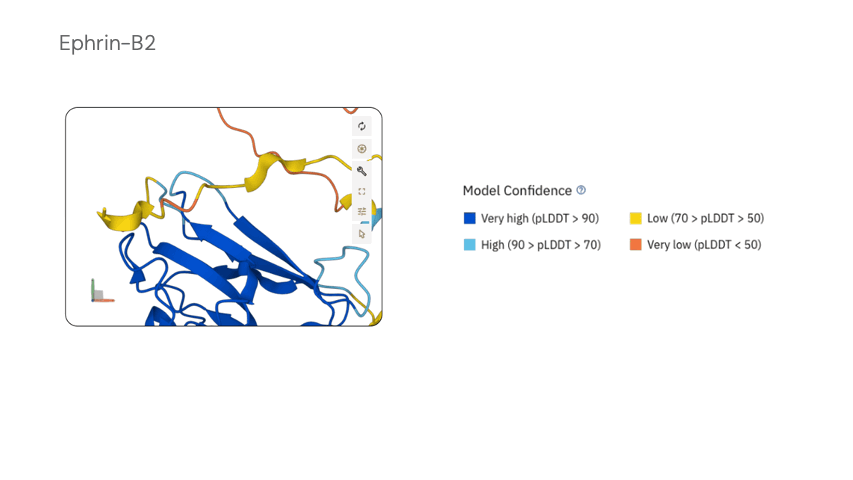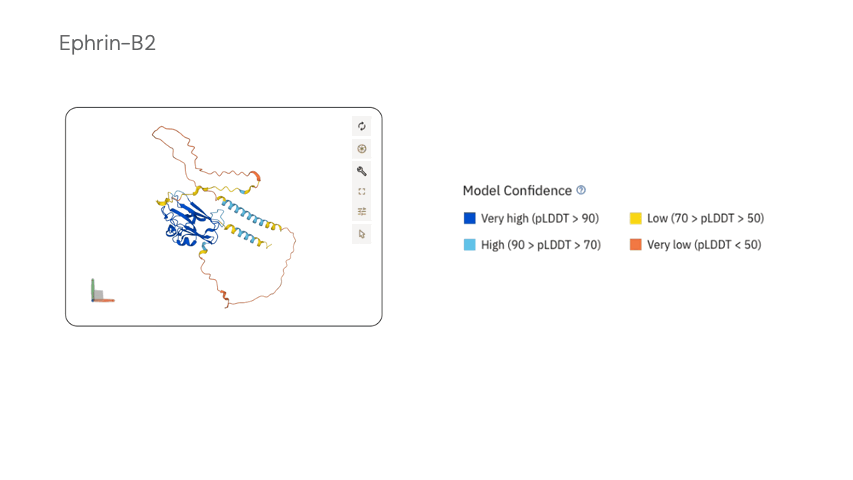
Рисунок 14. Метрика локальной уверенности модели (pLDDT) как измеритель «качества» модели. Темно-синий — очень высокая (pLDDT > 90), голубой — высокая (90 > pLDDT > 70), желтый — низкая (70 > pLDDT > 50), оранжевый — очень низкая (pLDDT < 50; таким участкам обычно доверять не стоит).
Отдельно важно подчеркнуть, что все эти метрики, как и сама структура, все еще являются предсказаниями модели, но, тем не менее, именно они сегодня де-факто используются как фильтр качества, который можно (и нужно) применять еще на стадии компьютерного проектирования, до передачи в лабораторию. По профилю pLDDT, PAE, pTM и ipTM можно уже на in silico этапе отсекать явно слабые варианты и сосредотачиваться на последовательностях, в геометрии которых модель уверена. Компьютеру безразлично, сколько тысяч таких кандидатов он «выбросит» (ну, если есть хороший компьютер), а вот для эксперимента в пробирке каждая лишняя попытка обходится дорого.
Помимо AlphaFold, есть целый ряд моделей, которые работают очень похоже и выдают примерно тот же набор метрик уверенности. OpenFold и Uni-Fold реализуют ту же архитектуру, но есть в открытом доступе, поэтому их можно дообучать под свои задачи [30], [31]. RoseTTAFold нередко используют как денойзер. ESMFold опирается на большую языковую модель и умеет быстро считать структуры без MSA, что удобно для массового скрининга (подробнее его разбирали в статье «Как языковые модели покорили мир белков» [18]). OmegaFold и HelixFold3, как и AlphaFold3, сразу предсказывают комплексы белков с лигандами и нуклеиновыми кислотами [32], [33]. А более специализированные модели вроде Boltz фокусируются на взаимодействии белка с лигандом и потому активно используются в задачах дизайна байндеров и in silico скрининга [34]. В итоге формируется целая единая экосистема: различные модели выдают сопоставимые метрики, поэтому их можно обрабатывать одними и теми же программами, легко сравнивать, взаимозаменять и комбинировать в одном конвейере
Эти метрики можно использовать не только в качестве фильтра дизайнов. В BindCraft, например, AlphaFold2 выступает таким «наставником», который по своим же метрикам указывает, где нужно подправить последовательность байндеров (рис. 15). Модель смотрит на pLDDT, PAE, ipTM и другие показатели интерфейса, чуть-чуть редактирует аминокислотную последовательность в «проблемных» зонах и снова прогоняет ее через AF2 — так цикл повторяется много раз. Интуитивно процесс похож на многократное переписывание участка белка с оценкой того, насколько эта последовательность нравится AlphaFold2: становится ли зона контакта все более «синей» (см. рис. 14) и аккуратно скомпонованной с лигандом. Со временем так удается «дотянуть» байндер до варианта, который, по мнению модели, уверенно и специфично садится на нужную мишень [35].
При этом белок, который «нравится» AlphaFold2 на картинке, еще не обязательно будет устойчивым в пробирке. Поэтому в BindCraft дополнительно через ProteinMPNN пересобирают только ту часть байндера, которая не взаимодействует с мишенью — под уже найденный каркас подбирают более физически реалистичную и устойчивую последовательность, а участок связывания с мишенью оставляют неизменным.
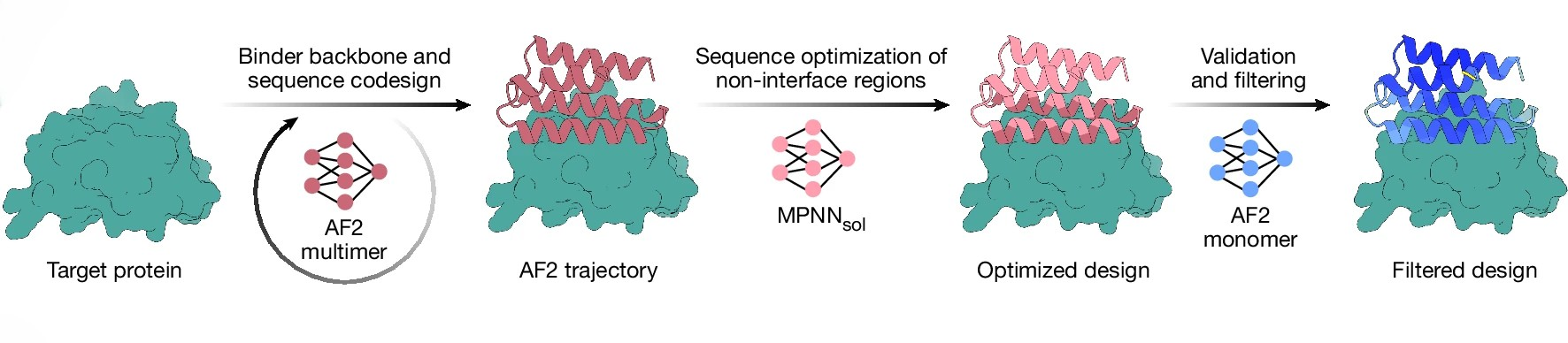
Рисунок 15. Схема работы BindCraft. AlphaFold2 в режиме multimer по собственным метрикам шаг за шагом «дотягивает» каркас и последовательность байндеров под заданную мишень. Затем ProteinMPNN пересобирает неинтерфейсные (не участвующие в связывании) участки в более физически реалистичную и стабильную последовательность, после чего финальные варианты еще раз прогоняются через AF2 и отбираются по качеству модели. Этот пайплайн заточен на максимальную долю успешных дизайнов в лаборатории (success rate), поэтому его крайне строгие фильтры часто могут и не пропустить ни одной молекулы, заставляя программу бегать по зачарованному кругу и тратя компьютерные ресурсы.
В 2024 году Google DeepMind представила AlphaFold3. Ключевое нововведение — это диффузионный модуль, который за счет своей гибкости точнее достраивает атомарное расположение всех компонентов комплекса. Благодаря этому AlphaFold3 уверенно работает с широким спектром химических классов молекул — от белков и нуклеиновых кислот до малых органических соединений и ионов [23].
Несмотря на то, что метрики AlphaFold позволяют ранжировать тысячи кандидатов и быстро отбрасывать заведомо слабые, они все равно остаются лишь приближением реальности. По ним нельзя напрямую узнать, насколько белок устойчив, активен, токсичен и вообще пригоден ли для работы в живой системе. Поэтому после любого ИИ-конвейера неизбежно всегда следует — и еще долго будет следовать — «мокрый» этап.
«Мокрый» этап: испытание лабораторией
К моменту, когда дело доходит до экспериментов, компьютер уже сильно сузил воронку: из тысяч, а иногда десятков и даже сотен тысяч in silico-вариантов остаются единицы наиболее многообещающих конструкций. Перебирать сотни тысяч in silico-вариантов для компьютера ничего не стоит, но окончательный вердикт все равно выносит пробирка: получается ли белок в живой системе, стабилен ли он и делает ли то, ради чего его проектировали?
Сначала мы проверяем, а можно ли вообще получить спроектированный белок в клетке. Для этого синтезируем ген, вставляем его в экспрессионный вектор и запускаем продукцию (часто в E. coli, для более сложных белков используют дрожжи). После очистки часто оказывается, что белок может резаться протеазами, оказывать токсическое действие на клетку, не растворяться и выпадать в осадок или вовсе не экспрессироваться (например, если мРНК образует «неудобные» для рибосомы структуры) [36], [37]. Это первый, самый дешевый фильтр: неудачные дизайны отсеиваются еще до сложных и дорогих тестов.
Затем смотрим на устойчивость. Для этого используют дифференциальную сканирующую флуориметрию DSF (Differential Scanning Fluorimetry) и ее «бескрасительную» версию nanoDSF (nano Differential Scanning Fluorimetry). В классическом DSF белок нагревают в присутствии гидрофобного красителя: по мере разворачивания белка его гидрофобные участки приоткрываются, краситель связывается с ними и начинает светиться сильнее — по изменению флуоресценции можно оценить стабильность белка [38].
В nanoDSF внешний краситель уже не нужен: прибор регистрирует тонкие изменения собственной флуоресценции ароматических аминокислот (триптофана и тирозина) при разворачивании белка [39]. У этого подхода свои ограничения: для nanoDSF в белке должны быть подходящие остатки (Trp/Tyr), а DSF может давать артефакты с примесями, связывающими краситель. Поэтому выбор метода зависит от задачи: для массового скрининга удобнее классический DSF, а когда белка мало или он богат ароматическими остатками, предпочтительнее nanoDSF.
И наконец, мы проверяем главное — работает ли белок как задумано. Катализирует ли реакцию фермент? связывается ли лиганд с рецептором? активирует ли он его? насколько крепко и избирательно связывается байндер с мишенью (токсин, вирусный белок, маркер и пр.)?
Если цель — катализ, ставят реакцию и измеряют каталитическую активность: скорость превращения субстрата при заданных условиях. Если цель — связывание, используют методы, которые позволяют отслеживать этот процесс в реальном времени. Чаще всего для таких измерений используют два похожих метода — поверхностный плазмонный резонанс (SPR) и биослойную интерферометрию (BLI). В обоих случаях белок или его мишень фиксируют на специальном сенсоре. Когда белок связывается с мишенью, меняются характеристики отраженного от сенсора света (угол отражения и показатель преломления для SPR [40] или положение пика изменения длины волны для BLI). Прибор по этим измерениям считывает, насколько быстро и насколько крепко белок связывается со своей мишенью [41].
В итоге хороший дизайн проходит двойное испытание — на устойчивость и функциональность. Кандидаты, у которых есть приемлемая экспрессия, приличная устойчивость, нормальная каталитическая или связывающая активность — идут дальше. Цифры из лаборатории помогают подправить связывающие карманы, контакты, переподобрать последовательность или, когда удачному варианту чуть-чуть не хватает до идеала, пройти пару этапов направленной эволюции [42].
Третья серия: горизонт будущего — куда мы движемся?
Если еще недавно дизайн белков опирался в основном на тяжелые физические расчеты и огромные библиотеки мутантов, то сейчас роль ИИ быстро растет. В первичном поиске доля мокрой биологии снижается — модели все точнее генерируют белки, а доля неудачных дизайнов падает [20], [43].
BindCraft, например, уже показывает, что функциональные байндеры можно получать почти в один заход (конечно, для «простых» мишеней). Нейросеть оптимизирует последовательности прямо по метрикам AlphaFold2 и в ряде случаев дает 10–100% экспериментальных попаданий без массивного высокопроизводительного скрининга (а это очень много, если вспомнить, что начиналось все с success rate 0,1% и меньше). Это серьезный шаг вперед по сравнению с прежними подходами, но пока его высокий процент успешных дизайнов ограничен тщательно подобранными мишенями — до универсальной «кнопки дизайна белков» еще далеко [44]. Модель BoltzGen идет еще дальше и объединяет дизайн и предсказание структуры в одной всеатомной генеративной модели, способной придумывать белки и пептиды-байндеры к самым разным мишеням [45].
Обновленная RFdiffusion (RFdiffusion2) научилась проектировать ферменты напрямую по атомарному описанию активного центра: модель сразу строит остов белка под несколько каталитических остатков. В таком режиме модель справляется даже со сложными активными центрами: в тестах на 41 задаче она построила рабочие каркасы во всех случаях (против 16 из 41 у предыдущих DL-методов). В экспериментах такие ферменты давали прирост каталитической активности на порядки по сравнению с конструкциями, полученными старыми ИИ-подходами [21].
Chroma, в свою очередь, объединяет диффузионную генерацию формы с моделью, по духу похожей на ProteinMPNN, причем каркас может генерироваться нефиксированными областями, а последовательность проектироваться под него сразу после генерации каркаса — из-за этого аминокислоты меньше прибиты к единственному положению в пространстве, а сама диффузия лучше учитывает природу белка. Благодаря этой особенности взаимодействия двух модулей повышается согласованность формы и последовательности, что увеличивает шансы на успешный дизайн [19].
Кроме того, тонкая доводка сгенерированного белка все так же делается классическими приемами — направленной эволюцией удачных вариантов. Разница в том, что это уже не основной путь поиска, а финальный штрих: ИИ сужает воронку до нескольких десятков разумных кандидатов, а эволюция в пробирке доводит их до нужной устойчивости, активности или специфичности.
Практическая значимость
Сегодня уже создано множество белков с заранее заданными функциями. За последние годы области их применения постепенно начали выходить за рамки демонстрационных примеров и становиться основной для лекарств, противоядий, вакцин, диагностических тестов и промышленных ферментов. Обо всем этом можно послушать самого Дэвида Бейкера — общепризнанного отца белкового дизайна (видео 1):
Видео 1. Выступление Дэвида Бейкера на TED.
Лекарства и таргетные белки с нуля. Небольшие стабильные белки способны садиться в заданный карман вирусных белков и блокировать их. Один из наиболее показательных примеров — минибелки, спроектированные под шиповидный S-белок коронавируса: конструкции длиной ~50—60 аминокислот с очень высокой аффинностью связывают и полностью перекрывают участок взаимодействия с рецептором ACE2 (angiotensin-converting enzyme 2), через который коронавирус попадает в клетки дыхательных путей и ряда других тканей [5]. Такие минибелки эффективно нейтрализовали вирус in vitro и у животных [46]. Высокая скорость получения таких белков, их термостабильность, высокая аффинность и возможность локальной доставки (например, назальной) позволяют довольно быстро собирать на их основе новые противовирусные средства под очередную вспышку.
Похожая стратегия сработала и для ядов змей: недавно команда Бэйкера с помощью RFdiffusion спроектировала небольшие байндеры к токсинам яда кобр. В экспериментах такие белки эффективно блокировали связывание нейротоксинов с ацетилхолиновыми рецепторами и защищали мышей от смертельных доз токсина, обеспечивая 80–100% выживаемости, иногда даже при введении после начала действия токсина. Благодаря малому размеру, стабильности и относительно простому производству, такие противоядия потенциально могут быть быстрее и дешевле классических антител-противоядий [47]. Сейчас это еще доклиника: работа вышла в Nature совсем недавно, вокруг нее много обсуждений, но впереди еще годы работы, прежде чем такие белки смогут стать настоящими лекарствами и противоядиями.
Новое поколение вакцин. Самособирающиеся белковые частицы, на поверхности которых выставлены десятки копий одного и того же антигена, воспринимаются иммунной системой заметно сильнее, чем тот же белок в растворе. Для B-клеток это плотный «ковер» повторяющихся сигналов, поэтому уровень антител получается выше, а иммунная память держится дольше: в экспериментах после однократного введения антитела сохранялись месяцами. На одной такой частице можно одновременно разместить несколько разных антигенов — например, варианты одного и того же вирусного белка разных штаммов или фрагменты разных патогенов, — поэтому на этой платформе уже делают прототипы универсальных и мультивалентных вакцин [48].
Биосенсоры. Белковые рецепторы с высокой селективностью легли в основу множества простых экспресс-тестов — от полосок на вирусные антигены и сердечные маркеры до систем контроля пищевой безопасности и загрязнения среды [49], [50]. Чаще всего используют антитела или другие связывающие белки. Их закрепляют на тест-полоске или электроде и пропускают образец. В первом случае все работает как привычный экспресс-тест: если есть целевая молекула, на полоске появляются линии. Во втором случае белок сидит на электроде, а сигнал считывает прибор; такой сенсор часто можно использовать многократно — достаточно промыть поверхность и провести новый анализ. Упрощение создания и производства таких биосенсоров позволяет постоянно придумывать новые быстрые тесты под конкретные задачи. Во многих случаях результат можно получить за 10–30 минут — причем сразу «в поле»: у врача на приеме, на линии производства или прямо на месте отбора проб [49], [50].
Биокатализ для переработки отходов. Отдельное направление — ферменты для «разборки» пластиков. Современные ПЭТ-гидролазы работают при довольно широком диапазоне температур, около 30–50 oC, и за несколько дней превращают пластиковую упаковку обратно в мономеры. Например, фермент FAST-PETase почти полностью разложил необработанный ПЭТ из 51 разных термоформованных изделий всего за неделю при 50 oC, после чего из полученных мономеров снова собрали пластик бутылочного качества. Такие белки устойчивы, хорошо работают в технологичных условиях и уже доступны как готовые конструкции, которые можно встраивать в промышленные процессы переработки [51].
Все эти примеры показывают: ИИ в биологии постепенно превращается в обычный рабочий инструмент. Вместо того, чтобы каждый раз заново проходить всю историю эволюции, мы сразу переходим к финалу: сначала описываем, каким должен быть белок, а потом модель предлагает несколько подходящих вариантов.
Такой подход заметно снижает нагрузку на лаборатории, делает дизайн белков доступным все большему числу групп, а значит, в ближайшие годы, вероятно, мы увидим еще больше лекарств и биотехнологических решений, изначально спроектированных с помощью ИИ. И по мере того, как такие ИИ-инструменты входят в повседневную лабораторную работу, дизайнерские белки из редких демонстраций будут постепенно превращаться в привычный способ создавать новые молекулы.
Литература
- 12 методов в картинках: полимеразная цепная реакция;
- Lara Sellés Vidal, Mark Isalan, John T. Heap, Rodrigo Ledesma-Amaro. (2023). A primer to directed evolution: current methodologies and future directions. RSC Chem. Biol.. 4, 271-291;
- Дизайнерские ферменты на службе общества;
- Черный ящик изобилия. Нобелевская премия по химии 2018 года;
- Конструкторское бюро белков;
- Новые успехи в предсказании пространственной структуры белков;
- Проблема фолдинга белка;
- Роль слабых взаимодействий в биополимерах;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024);
- Kyeongseok Song, Haekang Ji, Jiwon Lee, Youngdae Yoon. (2025). Microbial Transcription Factor-Based Biosensors: Innovations from Design to Applications in Synthetic Biology. Biosensors. 15, 221;
- Ingrid Swanson Pultz, Malcolm Hill, Joanne M. Vitanza, Clancey Wolf, Lasse Saaby, et. al.. (2021). Gluten Degradation, Pharmacokinetics, Safety, and Tolerability of TAK-062, an Engineered Enzyme to Treat Celiac Disease. Gastroenterology. 161, 81-93.e3;
- Kevin A. Murray, Carolyn J. Hu, Sarah L. Griner, Hope Pan, Jeannette T. Bowler, et. al.. (2022). De novo designed protein inhibitors of amyloid aggregation and seeding. Proc. Natl. Acad. Sci. U.S.A.. 119;
- Peng Zhao, Ningyan Zhang, Zhiqiang An. (2022). Engineering antibody and protein therapeutics to cross the blood–brain barrier. Antibody Therapeutics. 5, 311-331;
- Joon Young Song, Won Suk Choi, Jung Yeon Heo, Jin Soo Lee, Dong Sik Jung, et. al.. (2022). Safety and immunogenicity of a SARS-CoV-2 recombinant protein nanoparticle vaccine (GBP510) adjuvanted with AS03: A randomised, placebo-controlled, observer-blinded phase 1/2 trial. eClinicalMedicine. 51, 101569;
- Guohao Zhang, Chuanyang Liu, Wenying Li, Jiajie Lu, Ang Li, Lingyun Zhu. (2025). Beyond evolution: De novo designed protein toolkit rewriting the rules of synthetic biology. Biosafety and Health. 7, 306-311;
- AlphaFold: нейросеть для предсказания структуры белков от британских ученых;
- Как языковые модели покорили мир белков;
- John B. Ingraham, Max Baranov, Zak Costello, Karl W. Barber, Wujie Wang, et. al.. (2023). Illuminating protein space with a programmable generative model. Nature. 623, 1070-1078;
- Joseph L. Watson, David Juergens, Nathaniel R. Bennett, Brian L. Trippe, Jason Yim, et. al.. (2023). De novo design of protein structure and function with RFdiffusion. Nature. 620, 1089-1100;
- Woody Ahern, Jason Yim, Doug Tischer, Saman Salike, Seth M. Woodbury, et. al. (2025). Atom level enzyme active site scaffolding using RFdiffusion2. BioRXiv;
- Baek M., Anishchenko I., Humphreys I., Cong Q., Baker D., DiMaio F. (2023). Efficient and accurate prediction of protein structure using RoseTTAFold2. BioRXiv;
- Josh Abramson, Jonas Adler, Jack Dunger, Richard Evans, Tim Green, et. al.. (2024). Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature. 630, 493-500;
- SciNat за июнь 2024 #3: как бактерии устраняют конкурентов, в чем преимущества AlphaFold 3 и неожиданная связь ожирения и иммунотерапии;
- Торжество компьютерных методов: предсказание строения белков;
- Huan Yee Koh, Yizhen Zheng, Madeleine Yang, Rohit Arora, Geoffrey I. Webb, et. al.. (2025). AI-driven protein design. Nat Rev Bioeng. 3, 1034-1056;
- J. Dauparas, I. Anishchenko, N. Bennett, H. Bai, R. J. Ragotte, et. al.. (2022). Robust deep learning–based protein sequence design using ProteinMPNN. Science. 378, 49-56;
- Jue Wang, Joseph L. Watson, Sidney L. Lisanza. (2024). Protein Design Using Structure-Prediction Networks: AlphaFold and RoseTTAFold as Protein Structure Foundation Models. Cold Spring Harb Perspect Biol. 16, a041472;
- John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, et. al.. (2021). Highly accurate protein structure prediction with AlphaFold. Nature. 596, 583-589;
- Gustaf Ahdritz, Nazim Bouatta, Christina Floristean, Sachin Kadyan, Qinghui Xia, et. al.. (2024). OpenFold: retraining AlphaFold2 yields new insights into its learning mechanisms and capacity for generalization. Nat Methods. 21, 1514-1524;
- Ziyao Li, Xuyang Liu, Weijie Chen, Fan Shen, Hangrui Bi, et. al. Uni-Fold: An Open-Source Platform for Developing Protein Folding Models beyond AlphaFold — Cold Spring Harbor Laboratory;
- Ruidong Wu, Fan Ding, Rui Wang, Rui Shen, Xiwen Zhang, et. al. High-resolutionde novostructure prediction from primary sequence — Cold Spring Harbor Laboratory;
- Liu L. et al. (2024). Technical Report of HelixFold3 for Biomolecular Structure Prediction. ArXiv;
- Jeremy Wohlwend, Gabriele Corso, Saro Passaro, Noah Getz, Mateo Reveiz, et. al. Boltz-1 Democratizing Biomolecular Interaction Modeling — Cold Spring Harbor Laboratory;
- Martin Pacesa, Lennart Nickel, Christian Schellhaas, Joseph Schmidt, Ekaterina Pyatova, et. al.. (2025). One-shot design of functional protein binders with BindCraft. Nature. 646, 483-492;
- Germán L. Rosano, Eduardo A. Ceccarelli. (2014). Recombinant protein expression in Escherichia coli: advances and challenges. Front. Microbiol.. 5;
- Grzegorz Kudla, Andrew W. Murray, David Tollervey, Joshua B. Plotkin. (2009). Coding-Sequence Determinants of Gene Expression in Escherichia coli. Science. 324, 255-258;
- Frank H Niesen, Helena Berglund, Masoud Vedadi. (2007). The use of differential scanning fluorimetry to detect ligand interactions that promote protein stability. Nat Protoc. 2, 2212-2221;
- Jie Wen, Harrison Lord, Nicholas Knutson, Mats Wikström. (2020). Nano differential scanning fluorimetry for comparability studies of therapeutic proteins. Analytical Biochemistry. 593, 113581;
- Миграция энергии плазмонного резонанса: вторая жизнь оптической спектроскопии;
- Ana Jug, Tomaž Bratkovič, Janez Ilaš. (2024). Biolayer interferometry and its applications in drug discovery and development. TrAC Trends in Analytical Chemistry. 176, 117741;
- Euan J. Hossack, Florence J. Hardy, Anthony P. Green. (2023). Building Enzymes through Design and Evolution. ACS Catal.. 13, 12436-12444;
- SciNat за ноябрь 2025 #2: «уставший» хроматин, ИИ-антитело и атлас развития мозга;
- Martin Pacesa, Lennart Nickel, Christian Schellhaas, Joseph Schmidt, Ekaterina Pyatova, et. al. BindCraft: one-shot design of functional protein binders — Cold Spring Harbor Laboratory;
- Hannes Stark, Felix Faltings, MinGyu Choi, Yuxin Xie, Eunsu Hur, et. al. BoltzGen: Toward Universal Binder Design — Cold Spring Harbor Laboratory;
- Longxing Cao, Inna Goreshnik, Brian Coventry, James Brett Case, Lauren Miller, et. al.. (2020). De novo design of picomolar SARS-CoV-2 miniprotein inhibitors. Science. 370, 426-431;
- SciNat за январь 2025 #3: ИИ разрабатывает противоядия, новый редактор генома и секрет болезни Хантингтона;
- Sebastian Ols, Klara Lenart, Rodrigo Arcoverde Cerveira, Marcos C. Miranda, Natalie Brunette, et. al.. (2023). Multivalent antigen display on nanoparticle immunogens increases B cell clonotype diversity and neutralization breadth to pneumoviruses. Immunity. 56, 2425-2441.e14;
- Tim Peto, Dominic Affron, Babak Afrough, Anita Agasu, Mark Ainsworth, et. al.. (2021). COVID-19: Rapid antigen detection for SARS-CoV-2 by lateral flow assay: A national systematic evaluation of sensitivity and specificity for mass-testing. EClinicalMedicine. 36, 100924;
- Wu Pengcheng, Song Jiaren, Sun Caixia, Zuo Wanchao, Dai Jianjun, Ju Yanmin. (2023). Recent advances of lateral flow immunoassay for bacterial detection: Capture-antibody-independent strategies and high-sensitivity detection technologies. TrAC Trends in Analytical Chemistry. 166, 117203;
- Hongyuan Lu, Daniel J. Diaz, Natalie J. Czarnecki, Congzhi Zhu, Wantae Kim, et. al.. (2022). Machine learning-aided engineering of hydrolases for PET depolymerization. Nature. 604, 662-667.






