
Медицинская мультивселенная: получится ли у омиксов людей лечить?
22 марта 2024
Медицинская мультивселенная: получится ли у омиксов людей лечить?
- 921
- 0
- 7
Биомедицина похожа на бескрайнее море. Ученым предстоит собрать целую флотилию кораблей из разных омиксов и узнать, какие успешно преодолеют все шторма и достигнут земли, где их ждут благодарные пациенты, а какие затонут под грузом данных, которые невозможно интерпретировать.
Рисунок в оригинальном разрешении.
иллюстрация Карины Илиопуло
-
Автор
-
Редакторы
-
Иллюстратор
Логичным этапом стремительного развития алгоритмов искусственного интеллекта, увеличения вычислительных мощностей и развития методов молекулярного анализа стало появление (мульти)омиксных технологий. Считается, что за ними будет победа в битве со сложными мультифакторными и социально значимыми патологиями, но пока это, как говорится, не точно. Так что давайте лучше разберемся, как и зачем используют (мульти)омиксы в биомедицине прямо сейчас — в заключительной публикации нашего спецпроекта.
Мультиомиксные технологии
На «Биомолекуле» уже разбирались методы и технологии молекулярной биологии, в том числе протеомные и геномные. В связи с технологическим прогрессом всё чаще применяют их комбинацию, причем к ним добавляются и другие «омики». В спецпроекте «Мультиомиксные технологии» мы расскажем не столько о самих методах, сколько об их применении в самых разных областях — фундаментальной науке, медицине, экологии, сельском хозяйстве. Мы рассмотрим, как калейдоскоп множества методов позволяет лучше увидеть общую картину, ответить на интересующие ученых вопросы и продвинуться в решении практических задач.
Партнер спецпроекта — аффилированные компании «Филип Моррис Интернэшнл» в России, ФМИ (PMI Affiliates in Russia).
Основная цель многих биомедицинских исследований — найти маркеры заболеваний, которые позволили бы определить болезнь на ранней стадии. Представим себе пациента, у которого ничего пока не болит, и он, полный сил, бегает и пляшет. Но сбой уже случился, и патологический процесс запущен. На каких молекулах это отразится прежде всего, и что позволит засечь невидимую глазу болезнь? За последние десятилетия проведено море исследований, в которых говорится об океане молекул, претендующих на роль таких маркеров. Но можно ли их использовать с такой целью на самом деле, не подвергнем ли опасности здоровье человека, положившись на биохимические параметры? Голова кругом.
К счастью, прогресс шел не только в биологических лабораториях, но и у биоинформатиков [1]. Появление специализированных баз данных, компьютерных кластеров при медицинских центрах и новых вычислительных методов вдохнули свежую струю в биомедицинские исследования. Появился новый тренд — изучать не отдельную молекулу, сигнальный путь или тип клеток, а процесс или систему целиком — это так называемая системная биология. Новые технологии подталкивают описывать сложные биологические системы так, чтобы вычленить самое интересное, а потом еще и найти этому практическое применение.
В самой первой статье Спецпроекта «Мультиомиксные технологии» [2] мы подробно рассказали об источниках данных, которые стали частью омиксной мультивселенной. Технологии секвенирования [3] предоставили ученым безбрежные океаны последовательностей ДНК, которые можно связать с первичной структурой белков. Структурная биология позволяет узнать их пространственное строение [4]. Технологии визуализации и микроскопии прояснили подробности покрытых ранее пеленой неизвестности клеточных и молекулярных процессов [5]. Добавим к этому новые аналитические технологии — масс-спектрометрию [6], микрофлюидику и так далее — и получим стремительно расширяющуюся базу данных всея молекул.
Знания о каждом из процессов, происходящих в человеческом организме, — это, безусловно, хорошо. Но раздельное развитие геномики, протеомики, метаболомики и других -омик [7] не могло продолжаться долго, ведь все процессы жизни взаимосвязаны. Говоря языком героев кинофильма «Облачный атлас»: «Все границы условны. Они созданы лишь для того, чтобы их переступать». Что и случилось — но давайте же попробуем разобраться, дало ли что-то большое мультиомиксное слияние для насущных задач биомедицины?
Часть 1. Начнем с основ: как объединение разнородных данных поможет медицине
Интеграция данных стала попыткой лучше понять молекулярные и клеточные процессы, разобраться в том, как и почему в них происходит сбой, к чему он приводит и как можно его исправить. Смешение омиксных данных стало основой, на которую пытаются опираться в персонализированной медицине (рис. 1) [8], [9]. Одна из ее задач — найти биомаркеры, позволившие бы выявить болезнь в момент, когда ее лечение потребует минимальных усилий со стороны врачей и столь же небольших затрат жизненных ресурсов пациентов [10].
Вспомним, что одна из основных проблем смертности от онкологии — слишком позднее ее определение, когда метастазы уже распространились по разным органам, и помочь пациенту гораздо сложнее. А что, если мы будем раз в год сдавать некий анализ, который покажет — нет, в организме всё хорошо и стабильно; или да, некий патологический процесс запущен — лечите, пока не поздно! Вот она, голубая мечта исследователей, занятых неоднозначным анализом (мульти)омиксов.
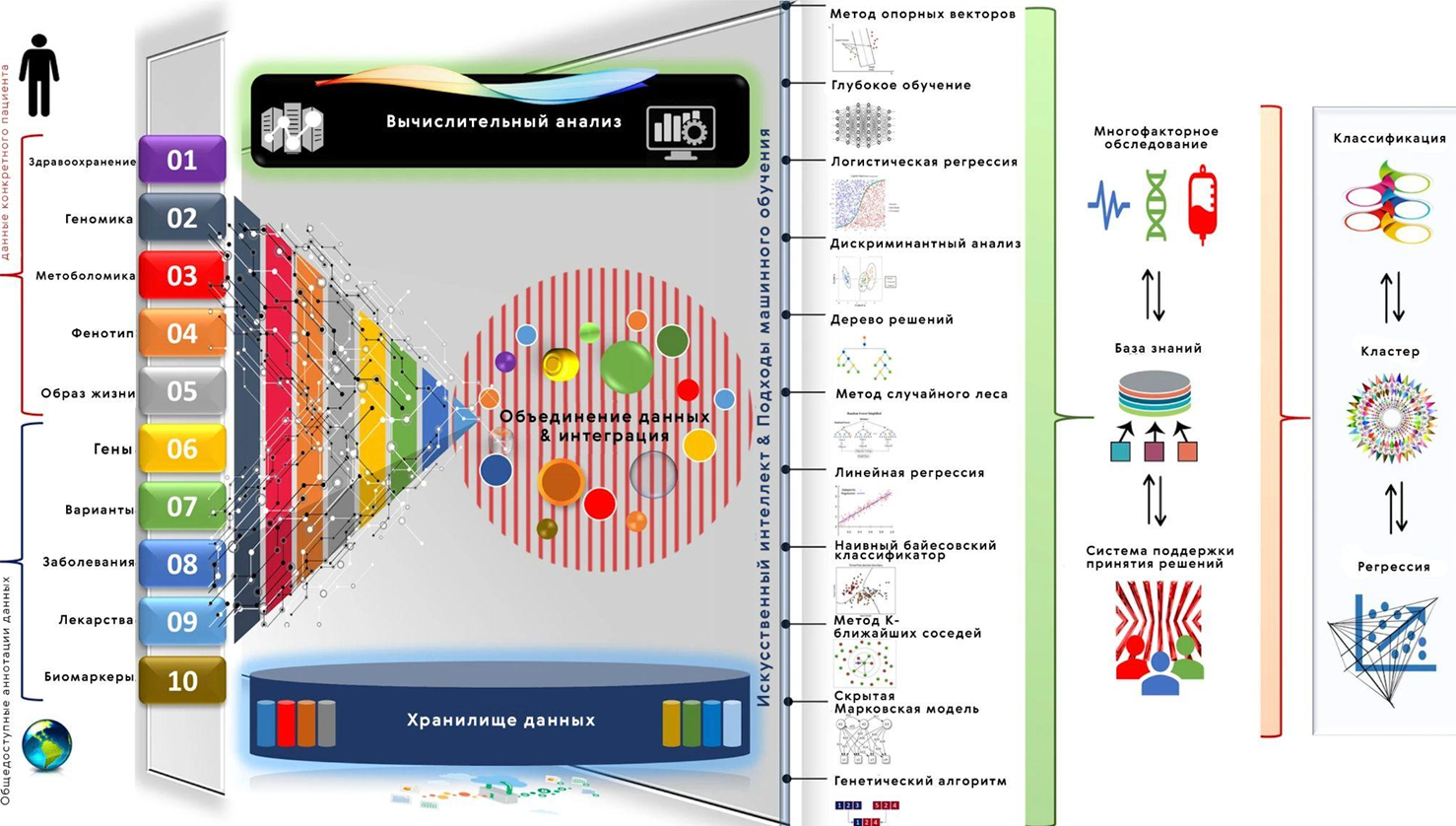
Рисунок 1. Алгоритмы обработки биомедицинских данных постоянно должны обучаться на новых потоках информации. Таким образом индивидуальные (мульти)омиксные данные о конкретном пациенте будут одновременно объединяться и сравниваться с существующей информацией. Алгоритмы ИИ направлены на извлечение полезных сведений, пополнение выборки и собственно обучение на ней. Извлеченные сведения предоставляют важную информацию для мультифакторного анализа, пополняют базу данных и служат источником помощи для других пациентов.
Конечно, есть не менее важные задачи — например, найти способ замедлять развитие заболеваний или разработать более эффективные фармпрепараты. Однако пока что мечты о спасении в лице (мульти)омиксных технологий остаются мечтами, и мы только-только подбираемся к тому, чтобы извлечь из обилия данных что-то полезное. Генерировать данные пока получается гораздо лучше.
Сложность возрастает при осознании того, что бóльшая часть этих омиксов динамическая и меняется в пространстве/времени. Ожидания возлагают на комбинации нескольких омиксов, чтобы создать более полный молекулярный профиль заболевания или отдельного пациента. Исследователи надеются, что этот профиль послужит трамплином для развития методов компьютерной диагностики; идентификации подтипов заболеваний; подходов к обнаружению сложных молекулярных паттернов, связанных с заболеванием; понимания регуляторных процессов, участвующих в патогенезе заболевания; и повышению точности прогнозов на медикаментозное лечение [12].
Перед исследователями, посягнувшими на омиксы, стоят следующие вопросы:
- какую их комбинацию рассматривать?
- какие базы данных использовать?
- какие алгоритмы для анализа данных привлечь?
- как интегрировать разные данные друг с другом?
- как на практике проверить гипотезы, полученные в компьютерных моделях?
От общего обозначения задач и проблем перейдем к более конкретным примерам того, на каком этапе находятся (мульти)омиксные технологии в разных областях биомедицины.
Часть 2. Осветят ли омиксы темную сторону нейробиологии?
Мультиомики уже широко применяются в нейробиологических исследованиях, начиная от обнаружения изменений в генах, мРНК, белках и отдельных метаболитах, до определения их локализации в конкретных отделах головного мозга (рис. 2) [13]. Ученые строят атласы мозга и изучают его динамику путем мониторинга изменений в отдельных клетках.
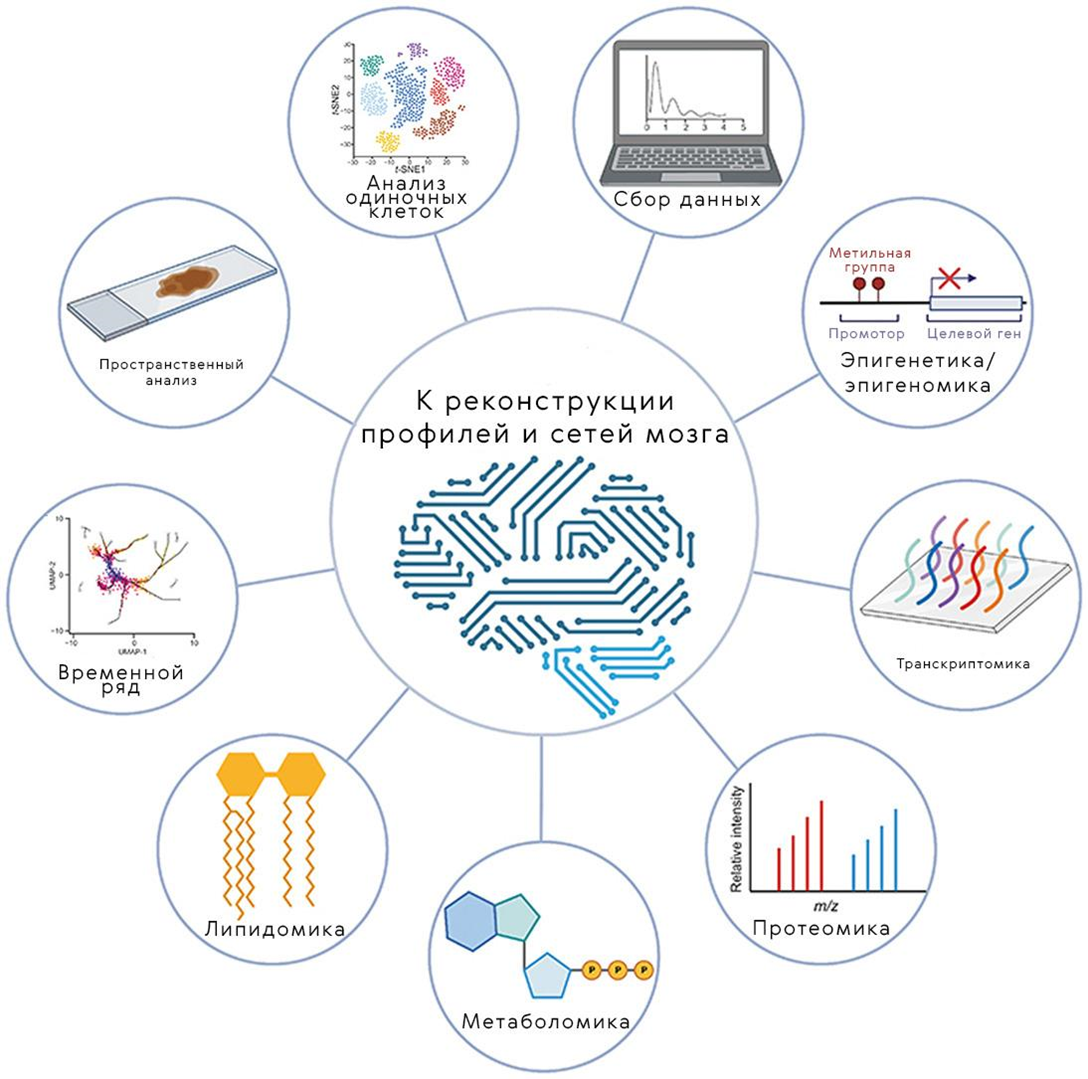
Рисунок 2. Путь к реконструкции нейронных сетей мозга вымощен мультиомиксным анализом. Отдельные МО-технологии уже не одно десятилетие пытаются применить к изучению особенностей развития и работы мозга; внесен весомый вклад и в изучение причин некоторых патологий. Например, геномика раскрыла, какие гены задействованы в развитии болезни Альцгеймера или насколько гетерогенна может быть нейробластома. Целью «многослойного» (мульти)омиксного анализа стал поиск новых, неизвестных ранее, закономерностей, способных внести вклад в прикладную науку.
Анализ массивов мультиомиксных данных открывает новые возможности для более глубокого понимания особенностей клинических фенотипов, невропатологических особенностей, причин прогрессирования заболеваний и механизмов, стоящих за этими процессами. В первую очередь усилия направлены на изучение наиболее социально значимых патологий, таких как болезни Альцгеймера и Паркинсона, рассеянный склероз, расстройства аутистического спектра, шизофрения [14] — особое внимание уделяется процессам нейровоспаления, нейродегенерации и нейроиммунной дисрегуляции (рис. 3).
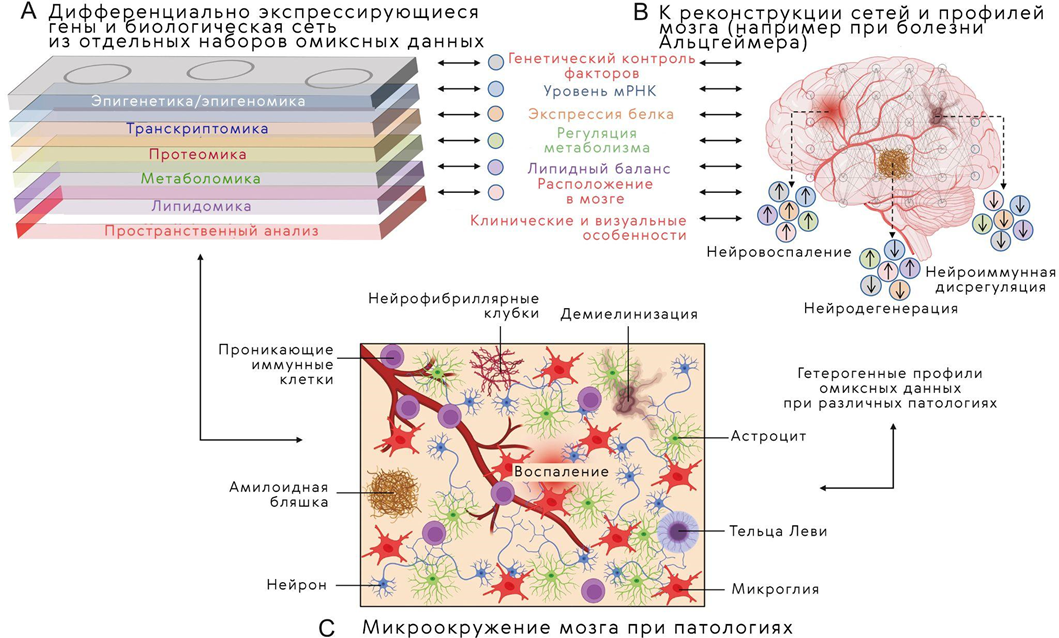
Рисунок 3. Интеграция омиксных данных вместе клиническими особенностями образуют патологический профиль заболевания головного мозга. Причем разные сочетания мультиомиксных слоев могут по-разному определить особенности состояния при нейровоспалении, нейродегенерации и нейроиммунной дисрегуляции.
В дополнение можно привести несколько примеров открытых баз данных, в которых представлены (мульти)омиксные данные (их много, мы сократим примеры до четырех):
- Allen Brain Atlas — один из первых мультиомиксных ресурсов по мозгу [15]. Есть профили экспрессии генов в приложении к разным процессам (старение, деменция, черепно-мозговая травма и т.д.), данные МРТ, данные секвенирования РНК одиночных клеток.
- Seattle Alzheimer’s Disease Brain Cell Atlas — есть транскриптомные, эпигеномные, геномные данные, полученные от пациентов с болезнью Альцгеймера.
- PsychENCODE — здесь представлены данные геномики и эпигеномики человеческого мозга для изучения нервно-психических расстройств.
- BRAINcode — представлены данные генома, эпигенома и транскриптома для изучения функции нейронов головного мозга, уязвимых к нейродегенеративным заболеваниям (например, дофаминовых нейронов при болезни Паркинсона, пирамидных нейронов при болезни Альцгеймера).
Давайте перейдем от общих слов к конкретным патологиям. Начнем с такого расстройства нервной системы, как шизофрения [16]. У пациентов с ней наблюдаются галлюцинации, нарушение когнитивных функций, психоз. Первые симптомы обычно появляются во взрослом возрасте. Известно более 100 геномных локусов, связанных с шизофренией [17]. Полногеномное секвенирование образцов разных пациентов приводит к выявлению всё новых генов, ассоциированных с заболеванием. В сочетании с транскриптомным анализом два источника омиксных данных позволили обнаружить неизвестные ранее изоформы белков. Оказалось, что не только отдельные мутации, но и особенности альтернативного сплайсинга стали причиной возникновения патологических изоформ. Подобные изменения сразу с двух сторон коснулись таких генов, как AS3MT (играет роль в развитии и дифференцировке нейронов), CACNA1C (кодирует субъединицу кальциевого канала, который играет важную роль в развитии дендритов и синаптической пластичности) и NRXN1 [18], [19]. Изменения в последовательности последнего гена связаны со многими нарушениями развития нервной системы и психическими расстройствами. Разные изоформы кодируемого NRXN1 белка участвуют в синаптической передаче сигналов между нейронами [20].
В качестве примера также рассмотрим значение (мульти)омиксных данных в изучении расстройств аутистического спектра (РАС) — наследуемого расстройства нервного развития. У РАС есть множество форм, в разных случаях у пациентов могут развиваться другие патологии нервной системы. Для изучения столь сложной по своей природе патологии, казалось бы, омиксные данные могут сильно помочь. Так, крупномасштабное исследование экзома 35 584 образцов, из которых 11 986 принадлежали пациентам с РАС, привело к обнаружению 53 генов, изменения в которых могут вносить вклад в развитие РАС [21]. Большинство из этих генов участвуют в развитии нейронов, нейронной коммуникации и нейрофизиологии. Некоторые из них особенно активны в возбуждающих и тормозящих нейронах, что подтверждает одну из гипотез происхождения РАС, основанную на дисбалансе активности этих типах нейронов [22].
В свою очередь, транскриптомный анализ помог определить тысячи некодирующих РНК (нкРНК), регуляция которых нарушена в мозге пациентов с РАС [23]. Данные о нкРНК получены при помощи методов NGS [3] и идентифицируют биомаркеры развития и вероятные терапевтические мишени [24]. Подобный множественный анализ проводит связь между фундаментальной нейробиологией и клиническим применением.
Несмотря на то, что процесс идет и появляются новые атласы, базы данных и всевозможные варианты их объединения, есть омиксные данные, которые несправедливо обошли вниманием — например, метилирование ДНК и экспрессия микроРНК.
Помимо этого, исследователи отмечают большую вариабельность (мульти)омиксных данных по полу, расе и другим переменным, однако в большинстве общедоступных баз эта вариабельность не особо отражена. Так, на 2019 год около 78% данных GWAS получены от людей европейского происхождения [25]. Это одна из причин, по которой методы терапии и лекарства, разработанные на базе этих данных, могут не сработать для людей другого происхождения. Похожие проблемы изучает особая область фармакологии — фармакогеномика [26].
Отметим также рост популярности изучения процессов нейробиологии при помощи органоидов [27], [28]. Уже больше десяти лет ученые выращивают в лабораториях мини-мозги из стволовых клеток, моделируют разные условия для изучения механизмов тех или иных патологий. Органоиды — это удобно: сокращается время и стоимость проведения экспериментов. Конечно, у всех подходов есть свои недостатки, но для пополнения мультиомиксных баз данных органоиды — неисчерпаемый источник.
О мультиомиксах в нейробиологии можно написать еще несколько статей; но давайте перейдем к не менее обширному полю, где такие данные могут дать ценные всходы, — онкологии.
Часть 3. Изучить опухоль со всех ракурсов — и снова при помощи «омиксов»
Возможно, именно онкология — лучшее приложение для (мульти)омиксных подходов. Гетерогенность опухоли и ее окружения [29] могут поставить в тупик в случае, если исследователь подступается к раку только с одним типом данных (рис. 4). Однако появление клеточных атласов опухоли поможет создать универсальную платформу для работы с индивидуальными случаями [30].
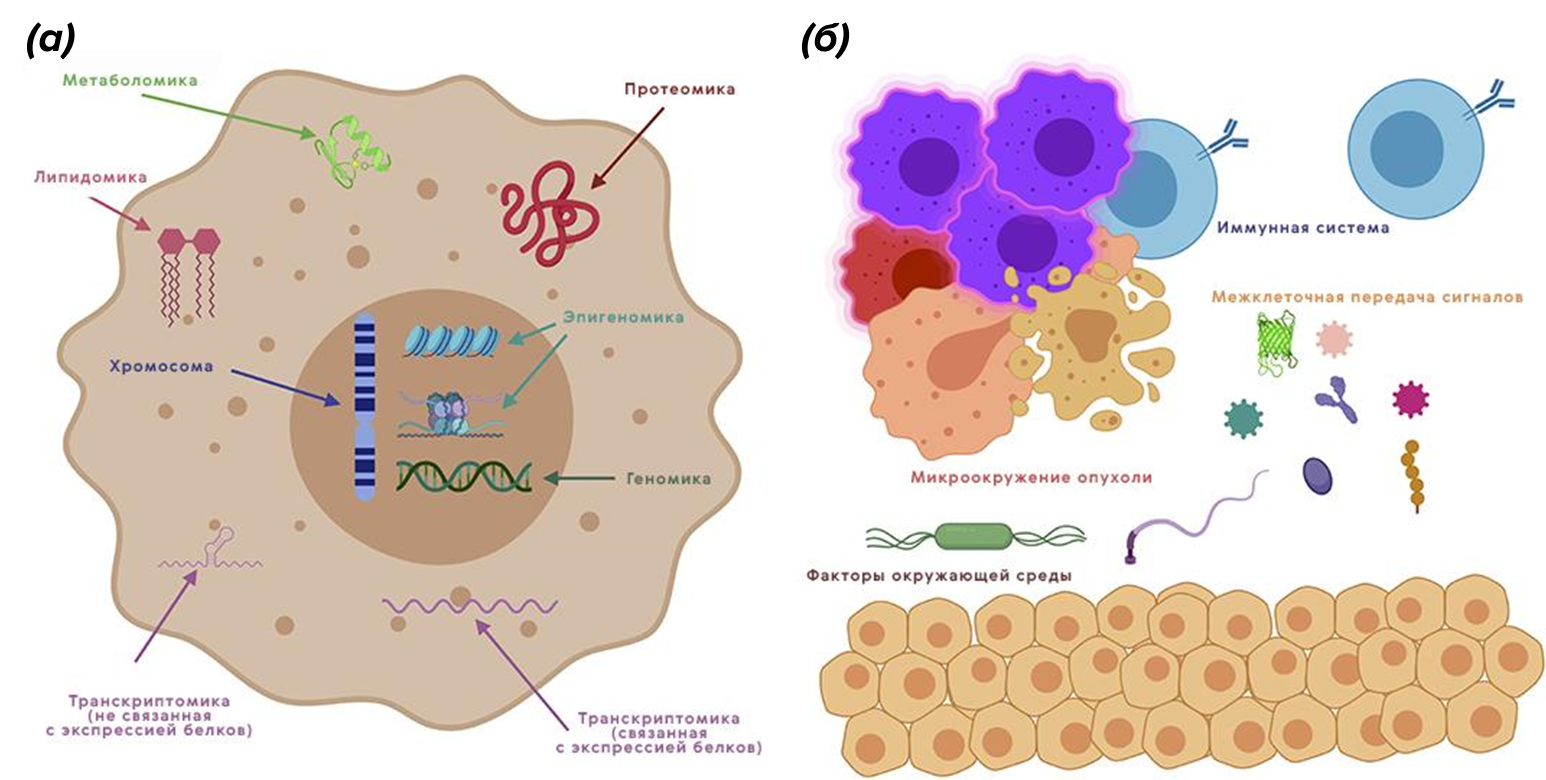
Рисунок 4. Внутриклеточные взаимодействия внутри опухолевой клетки можно описать при помощи нескольких омиксов: геномики, протеомики, транскриптомики, метаболомики и липидомики (а). Помимо этого, не стоит забывать о межклеточных и микробных взаимодействиях (здесь в игру вступает еще и метагеномика) (б).
Ученые уже рассматривают опухоль как сложную систему взаимосвязанных компонентов (рис. 5). Вместе они образуют сложную сеть — обширное поле для применения разных вычислительных алгоритмов. Также на рак воздействуют такие факторы, как окружающая среда и образ жизни. Они способны изменить развитие опухоли и процесс появления метастазов [31].
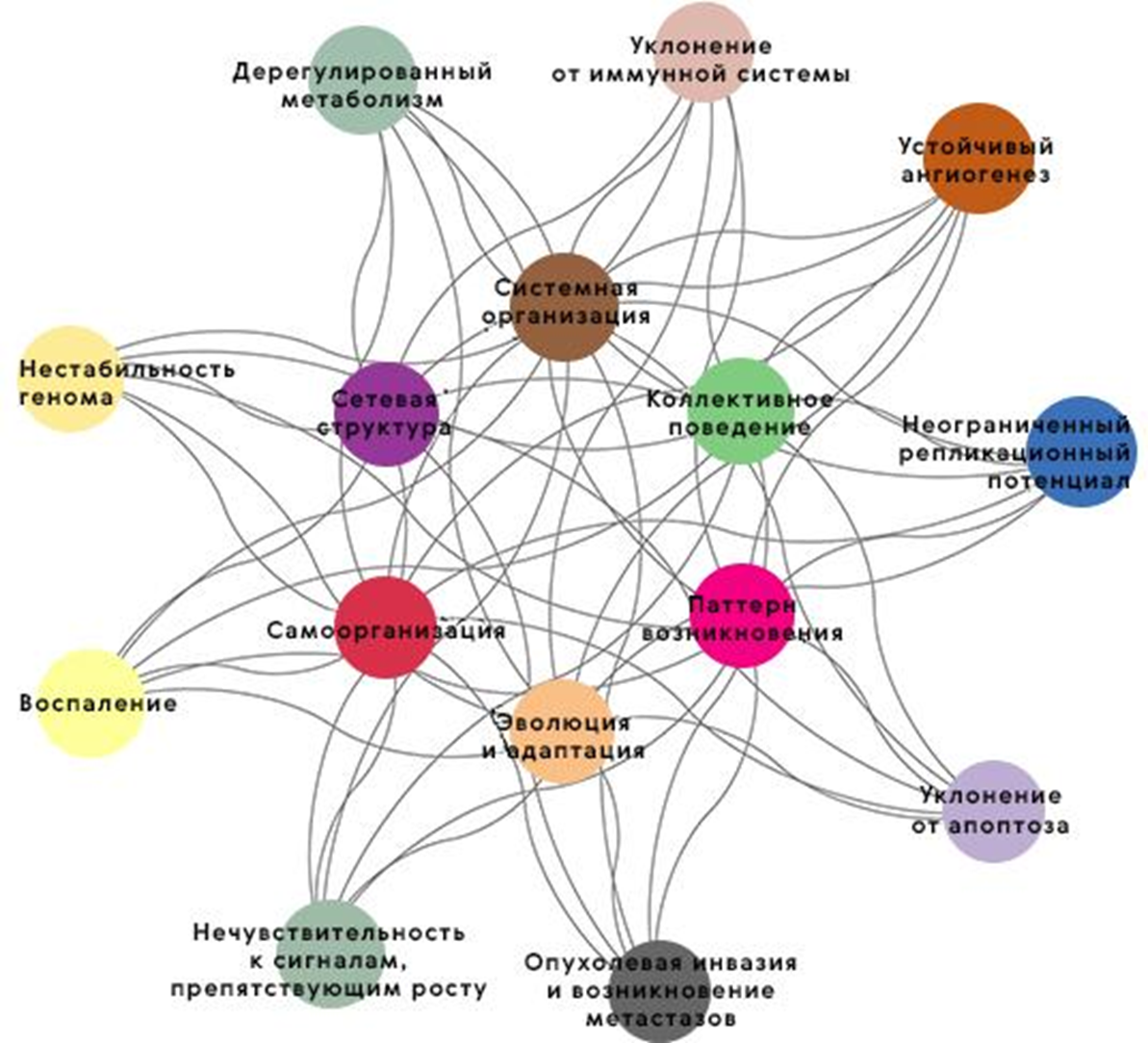
Рисунок 5. Многогранность свойств опухоли — гетерогенность, особенности адаптации к терапии, уклонение от иммунного ответа и многое другое. Поразительно напоминает ключевые признаки рака, которые мы разбирали в статье «От медицинской онкологии к молекулярной биологии рака» [29]!
Обработка омиксных данные о раке, само собой, требует мощного биоинформатического конвейера. Большое количество инструментов для анализа доступно в виде пакетов Bioconductor на языке R [32]. Самая первая из омиксов — геномика — в случае рака фокусируется на вариациях отдельных нуклеотидов (SNV), инделах, вариациях числа копий (CNV) и так далее. Уже существует огромный массив информации о связи изменений последовательности конкретного гена и развития опухоли.
В свою очередь, эпигеномика позволяет иначе взглянуть на особенности опухоли и расширяет взгляд, основанный на геномике. Так, например, обстоит дело с высокопроизводительными анализами иммунопреципитации, такими как ChIP-Seq. Данные транскриптомики также дополняют другие омиксы. Профиль экспрессии генов можно использовать как штрих-код биологического состояния клеток и изменений в них при раке [33].
Исследователи из разных стран уже предприняли попытки создать (мульти)омиксные профили для пациентов с раком. Один из них — The Cancer Genome Atlas (TCGA) — предоставляет подробные клинические, геномные, транскриптомные и протеомные данные по более чем 20 000 образцам. Составители атласа планируют расширить его, пополнив данными о разных типах онкологических заболеваний. И в который раз ученые сталкиваются с тем, что необходимо создание вычислительных алгоритмов, включающих интеграцию данных и статистические методы. Также следует привлекать алгоритмы искусственного интеллекта для того, чтобы из кучи данных можно было извлечь информацию для практического применения [34]. Малопредставленными в области изучения онкологии остаются метаболомные данные, к тому же доступ к многим омиксным данным всё еще ограничен [35].
Существует несколько подходов к работе с (мульти)омиксными данными в онкологии; приведем пример платформы SALMON, использующей глубокое обучение. Она объединяет транскриптомные данные, клинические характеристики и сведения о биомаркерах рака [36]. Результаты на пациентах с карциномой молочной железы показали улучшение способности прогнозировать выживаемость по сравнению с одиночными омиксами.
Часть 4. И еще об «омиксах» в борьбе с патологиями
О заболеваниях, которые мы обсудим дальше, наслышаны даже люди, далекие от биомедицины. Регулярно появляются новости о новых препаратах от этих заболеваний, но часто это только попытка СМИ привлечь внимание броским заголовком. На самом деле всё обстоит сложнее, да и сами болезни — сложные. А для того, чтобы разобраться в сложном, и методы используют непростые, как, например, (мульти)омиксные технологии. Дальше мы рассмотрим их приложение к таким патологиям, как сахарный диабет II типа, болезнь Альцгеймера и боковой амиотрофический склероз.
Все эти патологии вызваны сочетанием множества факторов окружающей среды и генетики, а их распространенность стала причиной серьезной нагрузки на здравоохранение и привлекла силы многих врачей и ученых к поиску эффективной терапии. К тому же, в контексте старения населения высока вероятность, что распространенность патологий будет только расти. Омиксы могут помочь обнаружить новые биомаркеры заболеваний, мишени для лекарственных средств и даже пути перепрофилирования существующих лекарственных препаратов (рис. 7) [38].
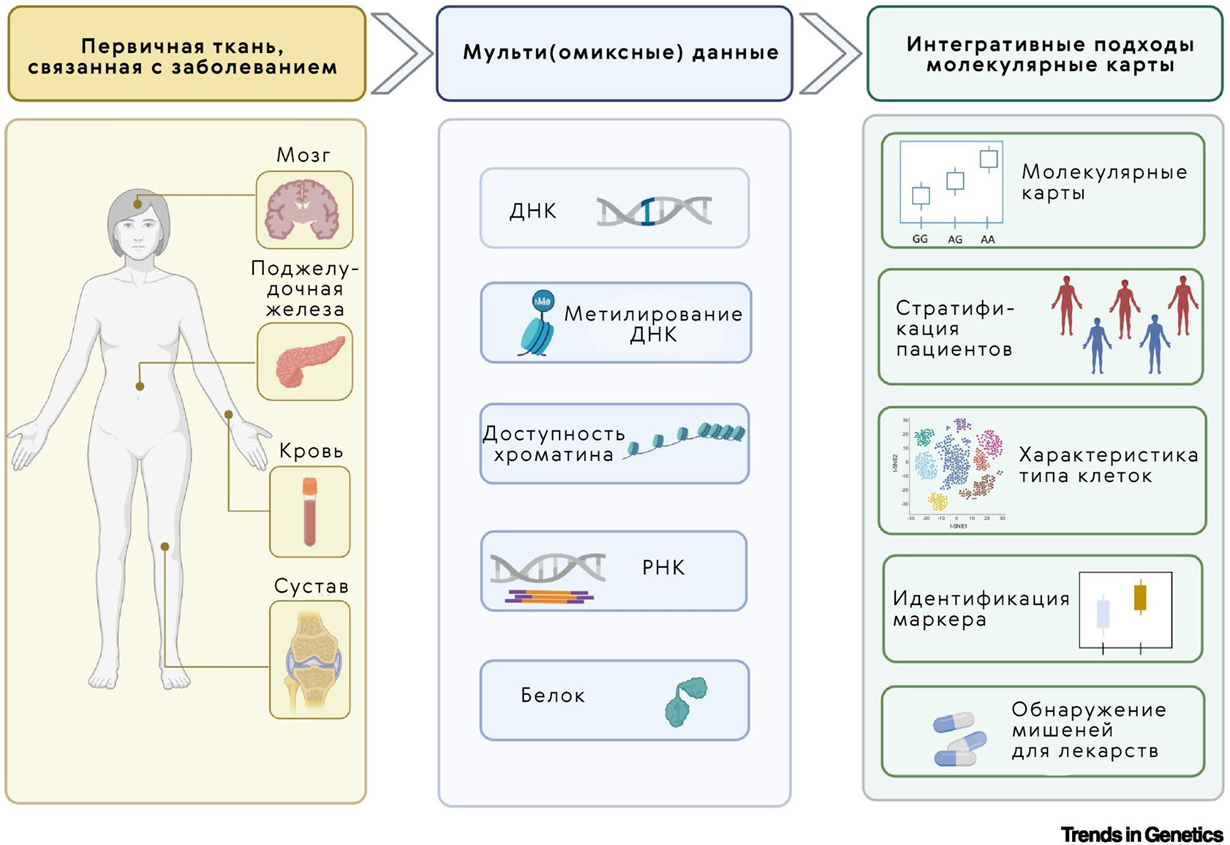
Рисунок 7. Ткани мозга — источник (мульти)омиксных данных о болезни Альцгеймера, поджелудочной железы — сахарного диабета, суставов — остеоартрита. Все они становятся источником (мульти)омиксных данных.
Сахарный диабет II типа
Сахарный диабет второго типа (здесь и далее речь будет идти только о нем) диагностирован у более чем 450 миллионов человек в мире [39]. Это многофакторное заболевание, при котором чаще всего у больных развивается инсулинорезистентность — клетки их организма больше не реагируют на инсулин, что приводит к проблемам обмена углеводов, а затем и множеству других осложнений со стороны эндокринной и сердечно-сосудистой систем. При сахарном диабете генетика играет роль, но только отчасти. Известно более 700 генетических вариантов, ассоциированных с сахарным диабетом [40]. Также есть транскриптомные данные клеток поджелудочной железы образцов, не связанных с диабетом, но способных в совокупности с геномикой пролить свет на конкретные механизмы нарушения работы клеток [38].
Есть еще диабет I типа — аутоиммунный: при нем реакция на инсулин сохраняется, но прекращается выработка самого этого гормона из-за аутоиммунной атаки и разрушения вырабатывающих его клеток поджелудочной железы: «Сахарный диабет I типа, или Охота на поджелудочную железу» [41].
Исследовательская группа из Швейцарии провела анализ геномных и транскриптомных данных. Из нескольких сотен потенциально связанных с диабетом генов ученые выделили 47 наиболее перспективных для дальнейшего изучения — и среди них DGKB и TCF7L2 [42]. Первый кодирует фермент под названием диацилглицеролкиназа бета, который участвует в превращении диацилглицерола в фосфатидную кислоту. Последняя в клетках поджелудочной железы необходима для высвобождения инсулина и, следовательно, контроля над концентрацией сахара в крови. В свою очередь, ген TCF7L2 задействован в сигнальном пути Wnt [43], и мутации в нем связаны не только с диабетом, но и другими сложными патологиями — например, колоректальным раком. Есть сведения, что некоторые варианты этого гена связаны со снижением реакции на терапию диабета сульфонилмочевиной [44].
В этом и других подобных исследованиях ученые работали с данными посмертных образцов. Первое в своем роде исследование с живыми пациентами провела другая группа ученых [45]. Они сумели использовать большее количество омиксных данных: транскриптомных, протеомных и липидомных. Результат проиллюстрировал клеточную гетерогенность в случае диабета. Потенциальными биомаркерами определены гликолитический фермент ALDOB, транспортер глюкозы SLC2A2 и маркер гликемии HbA1c. Ген ALDOB кодирует фермент альдолазу B, участвующий в расщеплении фруктозы и глюкозы. В образцах пациентов с диабетом активность гена оказалась повышенной, что требует дальнейшего изучения. В свою очередь, ген SLC2A2 кодирует гликопротеин, наиболее активный в клетках печени и поджелудочной железы, где он принимает непосредственное участие в транспорте глюкозы. Мутации в гене могут приводить к его инактивации и последующему накоплению глюкозы в тканях, трудностям с ее выведением и развитию сопутствующих патологий, в том числе и диабета [46]. HbA1c отражает долю гликированного гемоглобина — необратимой химической модификации этого белка, произошедшей под влиянием глюкозы. Поскольку HbA1c демонстрирует средний уровень гликемии за предыдущие два-три месяца, то есть за время жизни эритроцита, он часто используется в качестве теста для диагностики диабета и гликемического контроля у людей с диабетом [47]. Также ученые предполагают, что сахарный диабет связан с нарушением процессов экспрессии в зрелых клетках железы, а не с проблемами развития или дифференцировки молодых клеток.
Болезнь Альцгеймера
Болезнь Альцгеймера (БА) — наиболее распространенная форма деменции [48], сопровождающаяся накоплением β-амилоидных бляшек и тау-содержащих нейрофибриллярных клубков в коре головного мозга. Подробнее о ней мы уже писали: «Болезнь Альцгеймера: ген, от которого я без ума» [49] и «На руинах памяти: настоящее и будущее болезни Альцгеймера» [50].
Снова омиксы направляют в бой с гетерогенным заболеванием, что уже не должно удивлять. О генетике этой патологии известно уже довольно много, однако сложности при получении тканей для анализа затрудняют работу. Созданы молекулярные профили с данными дифференциальной экспрессии для конкретных областей мозга. Анализ одиночных клеток добавил четкости картине, до этого словно бы нарисованной крупными мазками [51]. Также созданные карты головного мозга и транскриптом одиночных клеток, несомненно, помогут в борьбе и с другими типами деменции.
Исследователи проанализировали 191 890 ядер префронтальной коры с одноклеточным разрешением, обнаружив патогенные варианты генов APOE и CLU, вариант SREBF1 в глиальных клетках и олигодендроцитах [52]. Другие ученые подошли к вопросу нестандартно и объединили данные протеомики и фосфопротеомики, что позволило выявить 173 белка, связанных с развитием БА. Наиболее значимый вклад в развитие патологии внесли уже знакомые по многим другим исследованиям мутации в генах APOE и MAPT [53]. Ген APOE кодирует аполипопротеин Е и связан с более чем половиной всех случаев БА; в норме он участвует в транспорте липидов в головном мозге, но его патогенетическая форма приводит к формированию амилоидных бляшек [54]. Ген MAPT кодирует ассоциированный с микротрубочками Tay-белок, а его мутации приводят к нарушениям сборки микротрубочек и последующему сбою в транспорте белков и их накоплению в тканях.
Боковой амиотрофический склероз (БАС)
При этой болезни у пациента постепенно разрушаются корковые и спинальные мотонейроны; подобная нейродегенерация приводит к мышечной слабости и последующему параличу. БАС — тоже гетерогенное заболевание, потому многие особенности и механизмы его течения до сих пор неизвестны. Геномика помогла провести ревизию в сложной системе клинических случаев, и теперь выделяют несколько форм патологии.
Еще в 1993 году с БАС впервые связали изменения в последовательности гена SOD1, который кодирует антиоксидантный фермент, защищающий клетку от токсичности активных форм кислорода [55]. Мутантные варианты фермента (их известно около 200!) приводят к накоплению вредных супероксидных радикалов, увеличению производства других типов токсичных радикалов и гибели клеток [56], [57]. Это мутации в таких генах, как C9orf72, FUS и TARDBP: C9orf72 кодирует белок, принимающий участие в отправке и получении сигналов между нейронами; белки генов FUS и TARDBP участвуют в регуляции транскрипции и альтернативного сплайсинга. Мутации в генах могут мешать транспорту мРНК из ядра клетки. В результате белок FUS, TARDBP и мРНК задерживаются внутри клеток и, вероятно, образуют сгустки (агрегаты) в нервных клетках, контролирующих движение мышц (мотонейронах).
Помимо геномики свой вклад внесла и транскриптомика — открытие белковых биомаркеров способствовало ранней диагностике, разделению разных подтипов БАС, что может помочь в выборе терапии [58]. Также есть сведения о вкладе эпигенома в патогенез [59]. Среди нескольких типов эпигенетических модификаций сильнее всего с развитием БАС связаны модификации белков-гистонов — как локальные, так и охватывающие весь геном. Так как эпигенетические модификации обратимы, здесь можно подумать о разработке нового «эпигенетического» лечения.
Часть 5. Тренд для тренда: попытка использовать омиксные данные против старения
(Мульти)омиксные технологии не обошли стороной и другую область, популярную уже в течение тысячелетий, — геронтологию (рис. 8) [60]. Мечты о буквально вечной жизни вряд ли кто-то считает реальными, но вот оставаться здоровым на протяжении долгих лет кажется уже мечтой не столь недостижимой. На сегодня от исследователей ждут разработки подходов для улучшения работоспособности и качества жизни пожилых людей. Что же здесь могут предложить омиксные данные уже сегодня?
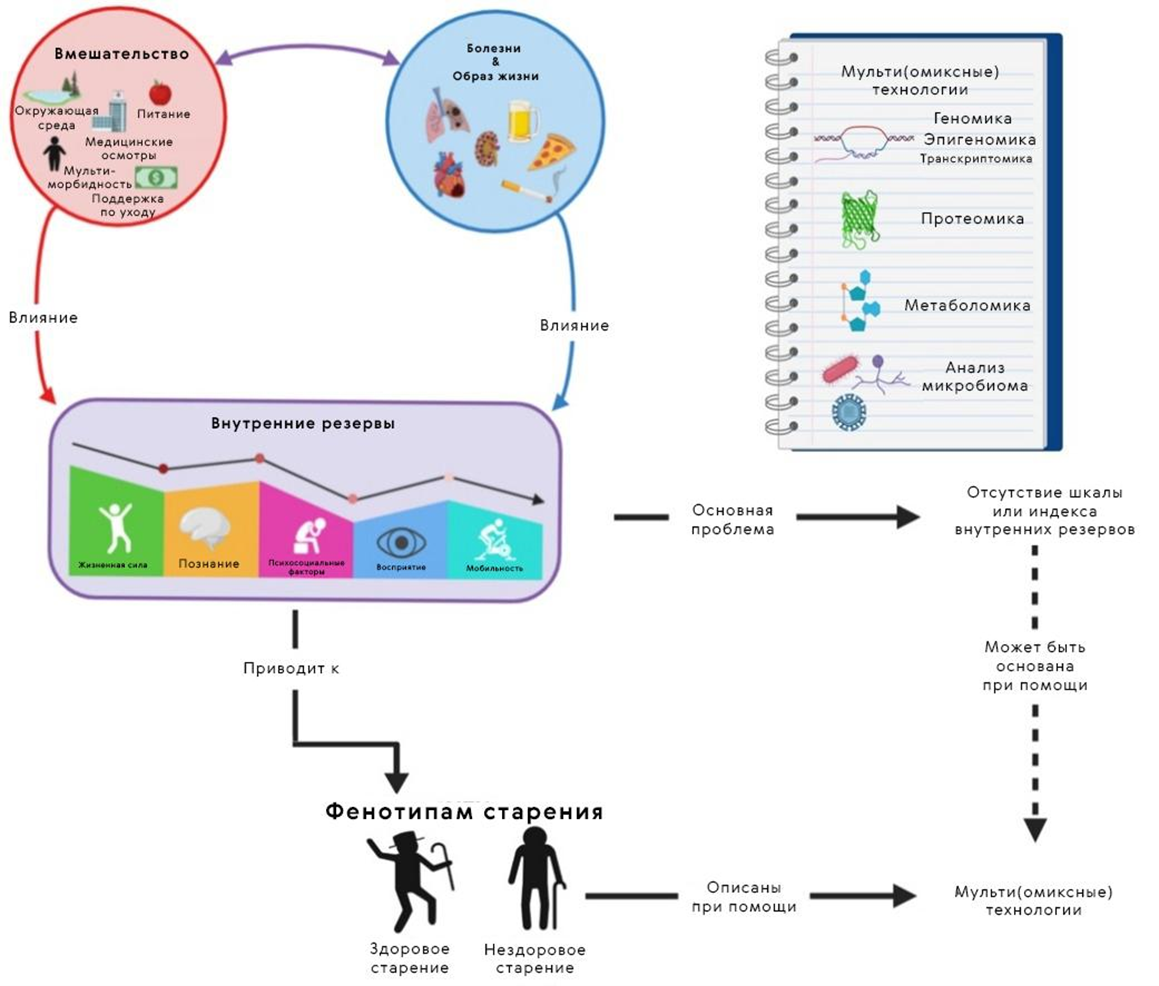
Рисунок 8. На протяжении жизни наш возрастной «потенциал» постоянно находится под давлением внешних сил: экологической обстановки, образа жизни, употребления лекарств и прочего. Так формируется характерный для конкретного человека фенотип старения. Он может быть описан и предсказан заранее при помощи (мульти)омиксных технологий — когда еще можно что-то изменить в выбранном пути.
Одна из основных трудностей, препятствующая пониманию природы старения — путаница между причинами и следствиями этого процесса. Возможно, омиксные данные помогут разобраться в этой интриге. Ведь применение всего набора данных метаболома, метилома, транскриптома, протеома и метагенома при разработке биомаркеров старения — единственный способ создать целостное представление о биологии старения. Тем не менее, весь существующий прогресс преимущественно касается разработки подходов к определению биологического возраста. Подробнее о том, как его определяют, рассказано в статье «Как и почему искусственный интеллект биологический возраст определял» [61] на «Биомолекуле».
На сегодняшний день не существует точно установленного независимого биомаркера старения, который мог бы наверняка отражать состояние или скорость старения людей. Тем более нет такого показателя, воздействие на который волшебным образом прекращало бы старение. Работа над тем, чтобы найти подобный биомаркер и использовать его, только-только началась. Так, созданы часы DNA GrimAge — инструмент, который может предсказать время до смерти и количество сопутствующих заболеваний, время до возникновения рака или ишемической болезни сердца [62]. Для оценки часы используют показатели семи белков плазмы крови, данные о хронологическом возрасте и образе жизни.
Часть 6. Перспективное приложение — омиксы в фарме
Создание профилей экспрессии генов на основе (мульти)омиксных технологий уже используют для перепрофилирования лекарств. Например, существует база данных CMap, позволяющая выявлять функциональные связи между низкомолекулярными соединениями, генами и заболеваниями.
Есть и другие примеры: так, группа ученых из Японии предложила вычислительный подход для перепрофилирования лекарств на основе анализа омиксных данных: транскриптома, протеома и интерактома [63]. Изначально алгоритм предложен для поиска лекарства от геморрагической лихорадки денге. Транскриптомные данные ученые извлекли из базы данных Gene Expression Omnibus (GEO), лекарства-кандидаты отобраны из CMap. Также исследователи построили сеть белок-белковых взаимодействий человека и вируса денге и использовали подход под названием GSEA — анализ обогащения набора генов (рис. 9) [64]. Входными данными при этом служат данные с ДНК-микрочипов. Алгоритм ранжирует гены на основе корреляции между их экспрессией и информацией о биохимических путях. Этот пример иллюстрирует отсутствие необходимости в лабораторных экспериментах на первых порах исследования: для того, чтобы отобрать наиболее перспективные кандидатные лекарства, достаточно баз данных и вычислительных мощностей компьютера.
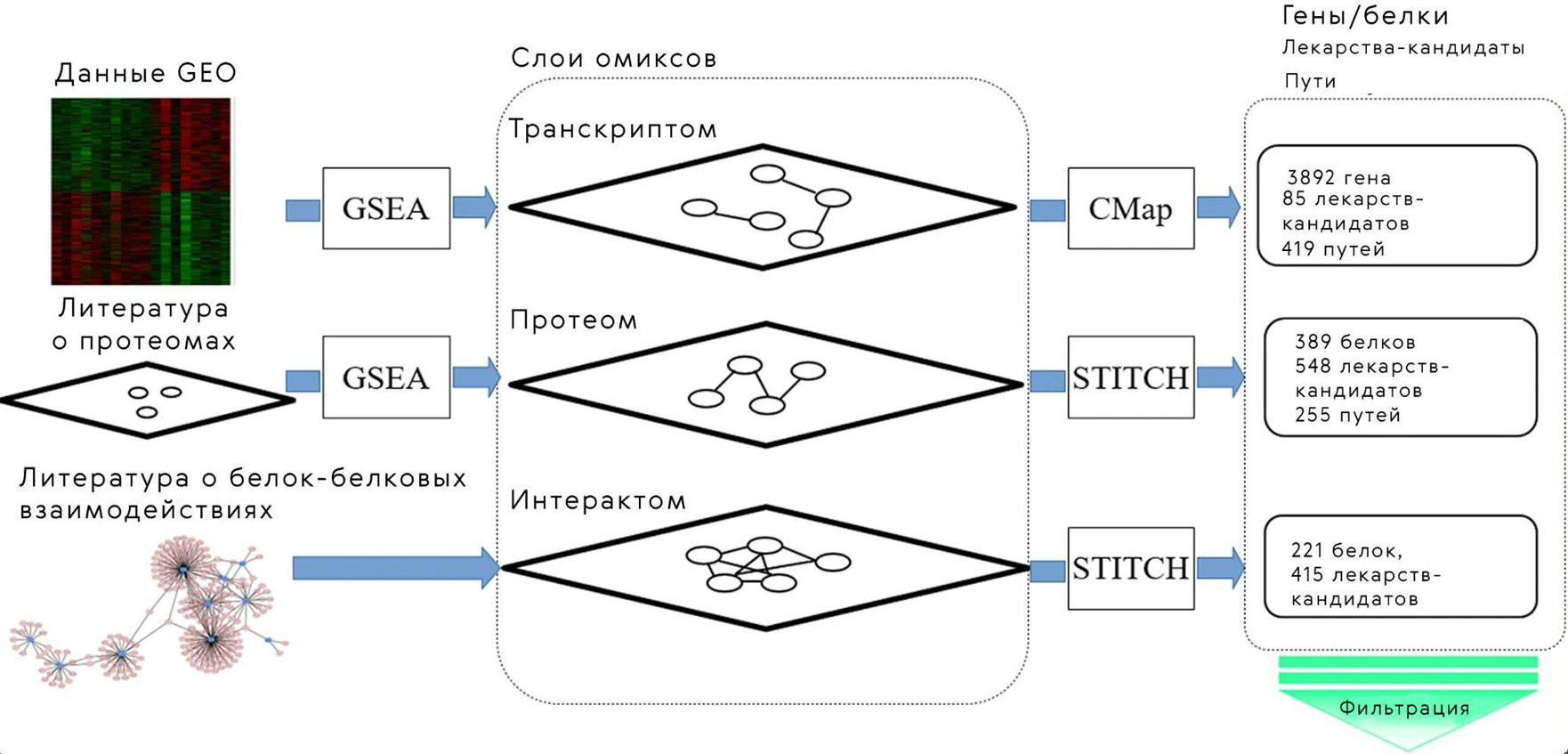
Рисунок 9. При помощи (мульти)омиксного подхода ученые определили наиболее перспективные лекарства-кандидаты против лихорадки Денге. Входными данными стали сведения о транскриптомах с микрочипов, хранящиеся в GEO, сведения о протеоме и белок-белковых взаимодействиях из литературы. Таким образом получилось три слоя данных: транскриптомные, протеомные и интерактомные. Алгоритм ИИ позволил выявить наиболее перспективные для дальнейших исследований гены, лекарства-кандидаты и метаболические пути. Таким образом, (мульти)омиксный подход позволил сократить количество итераций последующего анализа, сузив круг «подозреваемых».
Подобный подход для поиска эффективных лекарственных препаратов среди уже существующих особенно актуален для сложных заболеваний, о которых мы уже рассказывали ранее. Дело в том, что поиск и разработка лекарств в их случае занимает слишком много времени, которое столь ценно для пациента. Попытка найти спасение среди проверенных средств значительно ускоряет процесс. Как раз вторым важным фактором, ускоряющим процесс, и стали (мульти)омиксные технологии, которые сокращают время на экспериментальную проверку всех кандидатов-лекарств, изначально отбирая наиболее перспективные [65].
Первым лекарством, найденным при помощи искусственного интеллекта и омиксных данных, стал препарат DSP-1181 для терапии обсессивно-компульсивного расстройства (ОКР). От первоначального скрининга до выхода на клинические испытания прошло меньше года, в то время как обычно этот процесс занимает около четырех лет. Препарат схож с галоперидолом — мощным антипсихотическим средством, запатентованным еще в 1958 году. Галоперидол используется для терапии шизофрении, маниакальных расстройств и других психических заболеваний. Он угнетает дофаминовые рецепторы, нарушает процесс обратного захвата и депонирования норадреналина. Для лечения ОКР препарат официально не одобрен, тем не менее есть сведения, что он используется для усиления селективных ингибиторов обратного захвата серотонина у пациентов с ОКР. В свою очередь, DSP-1181 представляет собой полный агонист рецептора серотонина 5-HT1a [66].
Заключение
Как мы уже говорили, (мульти)омиксные технологии относительно недавно вступили в большую игру биомедицинских исследований. На уровне первоначальных исследовательских открытий стало известно многое. Где-то, как в области онкологии — больше; где-то (например, в геронтологии) — меньше. Станут ли мультиомики панацеей и внесут ли они весомый практический вклад в терапию, покажет время.
Литература
- Я б в биоинформатики пошёл, пусть меня научат!;
- Факты и легенды о мультиомиксных технологиях;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- 12 методов в картинках: структурная биология;
- 12 методов в картинках: микроскопия;
- 12 методов в картинках: протеомика;
- «Омики» — эпоха большой биологии;
- Zeeshan Ahmed. (2020). Practicing precision medicine with intelligently integrative clinical and multi-omics data analysis. Hum Genomics. 14;
- От медицины для всех — к медицине для каждого!;
- Michael Olivier, Reto Asmis, Gregory A. Hawkins, Timothy D. Howard, Laura A. Cox. (2019). The Need for Multi-Omics Biomarker Signatures in Precision Medicine. IJMS. 20, 4781;
- Омикс-биомаркеры и ранняя диагностика: когда счастье возможно;
- Efi Athieniti, George M. Spyrou. (2023). A guide to multi-omics data collection and integration for translational medicine. Computational and Structural Biotechnology Journal. 21, 134-149;
- Lance M. O'Connor, Blake A. O'Connor, Su Bin Lim, Jialiu Zeng, Chih Hung Lo. (2023). Integrative multi-omics and systems bioinformatics in translational neuroscience: A data mining perspective. Journal of Pharmaceutical Analysis. 13, 836-850;
- Gordon M. Shepherd, Jason S. Mirsky, Matthew D. Healy, Michael S. Singer, Emmanouil Skoufos, et. al.. (1998). The Human Brain Project: neuroinformatics tools for integrating, searching and modeling multidisciplinary neuroscience data. Trends in Neurosciences. 21, 460-468;
- Allen Brain Atlas: транскриптом мозга;
- Шизофрения и гены: поиск иголки в стоге сена;
- Sam Washer, Joe Burrage, Jonathan Mill, Aaron Jeffries, Emma Dempster. (2019). FUNCTIONAL ANALYSIS OF THE SCHIZOPHRENIA ASSOCIATED GENE AS3MT IN SH-SY5Y NEUROBLASTOMA CELLS. European Neuropsychopharmacology. 29, S986;
- Chu-Yi Zhang, Xiao Xiao, Zhuohua Zhang, Zhonghua Hu, Ming Li. (2022). An alternative splicing hypothesis for neuropathology of schizophrenia: evidence from studies on historical candidate genes and multi-omics data. Mol Psychiatry. 27, 95-112;
- Dongjian Zhu, Jingwen Yin, Chunmei Liang, Xudong Luo, Dong Lv, et. al.. (2019). CACNA1C (rs1006737) may be a susceptibility gene for schizophrenia: An updated meta‐analysis. Brain and Behavior. 9;
- Thomas C. Südhof. (2017). Synaptic Neurexin Complexes: A Molecular Code for the Logic of Neural Circuits. Cell. 171, 745-769;
- F. Kyle Satterstrom, Jack A. Kosmicki, Jiebiao Wang, Michael S. Breen, Silvia De Rubeis, et. al.. (2020). Large-Scale Exome Sequencing Study Implicates Both Developmental and Functional Changes in the Neurobiology of Autism. Cell. 180, 568-584.e23;
- Sacha B. Nelson, Vera Valakh. (2015). Excitatory/Inhibitory Balance and Circuit Homeostasis in Autism Spectrum Disorders. Neuron. 87, 684-698;
- Akira Gokool, Firoz Anwar, Irina Voineagu. (2020). The Landscape of Circular RNA Expression in the Human Brain. Biological Psychiatry. 87, 294-304;
- Mathieu Quesnel-Vallières, Robert J. Weatheritt, Sabine P. Cordes, Benjamin J. Blencowe. (2019). Autism spectrum disorder: insights into convergent mechanisms from transcriptomics. Nat Rev Genet. 20, 51-63;
- Giorgio Sirugo, Scott M. Williams, Sarah A. Tishkoff. (2019). The Missing Diversity in Human Genetic Studies. Cell. 177, 26-31;
- Фармакогеномика: изучение генов на службе персонализированной медицины;
- Органы-на-чипе: когда технология сплетается с жизнью;
- Синтетические эмбрионы человека: новое направление в исследованиях развития жизни;
- От медицинской онкологии к молекулярной биологии рака;
- Sarah S Knox. (2010). From 'omics' to complex disease: a systems biology approach to gene-environment interactions in cancer. Cancer Cell International. 10, 11;
- Guillermo de Anda-Jáuregui, Enrique Hernández-Lemus. (2020). Computational Oncology in the Multi-Omics Era: State of the Art. Front. Oncol.. 10;
- Wolfgang Huber, Vincent J Carey, Robert Gentleman, Simon Anders, Marc Carlson, et. al.. (2015). Orchestrating high-throughput genomic analysis with Bioconductor. Nat Methods. 12, 115-121;
- Euna Jeong, Sung Ung Moon, Mee Song, Sukjoon Yoon. (2017). Transcriptome modeling and phenotypic assays for cancer precision medicine. Arch. Pharm. Res.. 40, 906-914;
- Sijia Huang, Kumardeep Chaudhary, Lana X. Garmire. (2017). More Is Better: Recent Progress in Multi-Omics Data Integration Methods. Front. Genet.. 8;
- Ana Conesa, Stephan Beck. (2019). Making multi-omics data accessible to researchers. Sci Data. 6;
- Zhi Huang, Xiaohui Zhan, Shunian Xiang, Travis S. Johnson, Bryan Helm, et. al.. (2019). SALMON: Survival Analysis Learning With Multi-Omics Neural Networks on Breast Cancer. Front. Genet.. 10;
- Bjoern Titz, Justyna Szostak, Alain Sewer, Blaine Phillips, Catherine Nury, et. al.. (2020). Multi-omics systems toxicology study of mouse lung assessing the effects of aerosols from two heat-not-burn tobacco products and cigarette smoke. Computational and Structural Biotechnology Journal. 18, 1056-1073;
- Peter Kreitmaier, Georgia Katsoula, Eleftheria Zeggini. (2023). Insights from multi-omics integration in complex disease primary tissues. Trends in Genetics. 39, 46-58;
- Theo Vos, Stephen S Lim, Cristiana Abbafati, Kaja M Abbas, Mohammad Abbasi, et. al.. (2020). Global burden of 369 diseases and injuries in 204 countries and territories, 1990–2019: a systematic analysis for the Global Burden of Disease Study 2019. The Lancet. 396, 1204-1222;
- Anubha Mahajan, Daniel Taliun, Matthias Thurner, Neil R. Robertson, Jason M. Torres, et. al.. (2018). Fine-mapping type 2 diabetes loci to single-variant resolution using high-density imputation and islet-specific epigenome maps. Nat Genet. 50, 1505-1513;
- Сахарный диабет I типа, или Охота на поджелудочную железу;
- Ana Viñuela, Arushi Varshney, Martijn van de Bunt, Rashmi B. Prasad, Olof Asplund, et. al.. (2020). Genetic variant effects on gene expression in human pancreatic islets and their implications for T2D. Nat Commun. 11;
- Важнейшие стрелочники клеток организма: белки Wnt;
- Laura del Bosque-Plata, Eduardo Martínez-Martínez, Miguel Ángel Espinoza-Camacho, Claudia Gragnoli. (2021). The Role of TCF7L2 in Type 2 Diabetes. Diabetes. 70, 1220-1228;
- Leonore Wigger, Marko Barovic, Andreas-David Brunner, Flavia Marzetta, Eyke Schöniger, et. al.. (2021). Multi-omics profiling of living human pancreatic islet donors reveals heterogeneous beta cell trajectories towards type 2 diabetes. Nat Metab. 3, 1017-1031;
- F. H. Sansbury, S. E. Flanagan, J. A. L. Houghton, F. L. Shuixian Shen, A. M. S. Al-Senani, et. al.. (2012). SLC2A2 mutations can cause neonatal diabetes, suggesting GLUT2 may have a role in human insulin secretion. Diabetologia. 55, 2381-2385;
- Chloé Sarnowski, Aaron Leong, Laura M. Raffield, Peitao Wu, Paul S. de Vries, et. al.. (2019). Impact of Rare and Common Genetic Variants on Diabetes Diagnosis by Hemoglobin A1c in Multi-Ancestry Cohorts: The Trans-Omics for Precision Medicine Program. The American Journal of Human Genetics. 105, 706-718;
- Julie A. Schneider, Zoe Arvanitakis, Sue E. Leurgans, David A. Bennett. (2009). The neuropathology of probable Alzheimer disease and mild cognitive impairment. Annals of Neurology. 66, 200-208;
- Болезнь Альцгеймера: ген, от которого я без ума;
- На руинах памяти: настоящее и будущее болезни Альцгеймера;
- M. Ryan Corces, Anna Shcherbina, Soumya Kundu, Michael J. Gloudemans, Laure Frésard, et. al.. (2020). Single-cell epigenomic analyses implicate candidate causal variants at inherited risk loci for Alzheimer’s and Parkinson’s diseases. Nat Genet. 52, 1158-1168;
- Samuel Morabito, Emily Miyoshi, Neethu Michael, Saba Shahin, Alessandra Cadete Martini, et. al.. (2021). Single-nucleus chromatin accessibility and transcriptomic characterization of Alzheimer’s disease. Nat Genet. 53, 1143-1155;
- Bing Bai, Xusheng Wang, Yuxin Li, Ping-Chung Chen, Kaiwen Yu, et. al.. (2020). Deep Multilayer Brain Proteomics Identifies Molecular Networks in Alzheimer’s Disease Progression. Neuron. 105, 975-991.e7;
- Ana-Caroline Raulin, Sydney V. Doss, Zachary A. Trottier, Tadafumi C. Ikezu, Guojun Bu, Chia-Chen Liu. (2022). ApoE in Alzheimer’s disease: pathophysiology and therapeutic strategies. Mol Neurodegeneration. 17;
- Активный кислород: друг или враг, или О пользе и вреде антиоксидантов;
- Rosie K. A. Bunton-Stasyshyn, Rachele A. Saccon, Pietro Fratta, Elizabeth M. C. Fisher. (2015). SOD1 Function and Its Implications for Amyotrophic Lateral Sclerosis Pathology. Neuroscientist. 21, 519-529;
- Daniel R. Rosen, Teepu Siddique, David Patterson, Denise A. Figlewicz, Peter Sapp, et. al.. (1993). Mutations in Cu/Zn superoxide dismutase gene are associated with familial amyotrophic lateral sclerosis. Nature. 362, 59-62;
- Thomas J. Hedl, Rebecca San Gil, Flora Cheng, Stephanie L. Rayner, Jennilee M. Davidson, et. al.. (2019). Proteomics Approaches for Biomarker and Drug Target Discovery in ALS and FTD. Front. Neurosci.. 13;
- Seth A. Bennett, Royena Tanaz, Samantha N. Cobos, Mariana P. Torrente. (2019). Epigenetics in amyotrophic lateral sclerosis: a role for histone post-translational modifications in neurodegenerative disease. Translational Research. 204, 19-30;
- N.A. Rivero-Segura, O.Y. Bello-Chavolla, O.S. Barrera-Vázquez, L.M. Gutierrez-Robledo, J.C. Gomez-Verjan. (2020). Promising biomarkers of human aging: In search of a multi-omics panel to understand the aging process from a multidimensional perspective. Ageing Research Reviews. 64, 101164;
- Как и почему искусственный интеллект биологический возраст определял;
- Ake T. Lu, Austin Quach, James G. Wilson, Alex P. Reiner, Abraham Aviv, et. al.. (2019). DNA methylation GrimAge strongly predicts lifespan and healthspan. Aging. 11, 303-327;
- Takayuki Amemiya, M. Michael Gromiha, Katsuhisa Horimoto, Kazuhiko Fukui. (2019). Drug repositioning for dengue haemorrhagic fever by integrating multiple omics analyses. Sci Rep. 9;
- A. Subramanian, P. Tamayo, V. K. Mootha, S. Mukherjee, B. L. Ebert, et. al.. (2005). Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences. 102, 15545-15550;
- Xiangxiang Zeng, Siyi Zhu, Weiqiang Lu, Zehui Liu, Jin Huang, et. al.. (2020). Target identification among known drugs by deep learning from heterogeneous networks. Chem. Sci.. 11, 1775-1797;
- Talha Burki. (2020). A new paradigm for drug development. The Lancet Digital Health. 2, e226-e227.






