Наследование и эпигеном
15 октября 2021
Наследование и эпигеном
- 4747
- 0
- 16
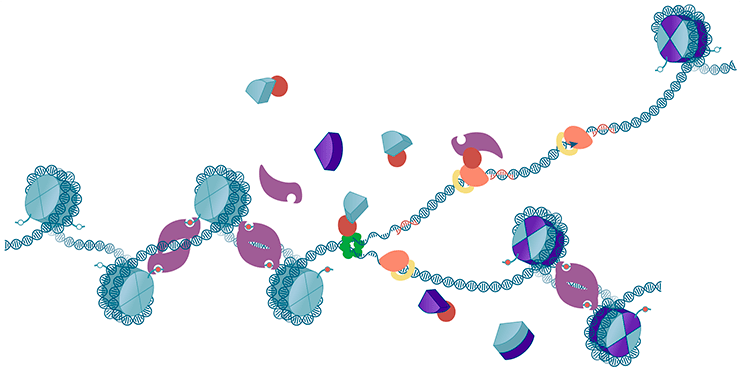
Эпигенетическое наследование гистоновых и негистоновых белковых меток во время репликации. Старые и новые гистоны, перемешиваясь, собираются в новые нуклеосомы; благодаря белкам репликации некоторые негистоновые белки перераспределяются с родительского дуплекса на дочерние цепи.
иллюстрация Михаила Гурьева
-
Автор
-
Редакторы
-
Иллюстратор
Темы
В продолжение спецпроекта по эпигенетике мы расскажем о наследовании эпигенетической информации — как она передается между клетками одного организма при делении и как ее получают наши потомки. (Спойлер: да, это может повлиять на то, какими они будут!) Речь пойдет о метилировании ДНК; гистоновых и негистоновых белках; активном и неактивном хроматине; роли во всем этом малых РНК; и даже о наследовании позиций нуклеосом на ДНК. Напоследок оставим важнейший вопрос — можно ли на самом деле клонировать мамонта?
Эпигенетика
Эпигенетика — с одной стороны, молодая и бурно развивающаяся наука. С другой стороны, в этой науке уже сложился фундамент основных понятий и концепций. Цель спецпроекта — рассказать об основных концепциях эпигенетики просто, но в то же время со всех сторон. Читатель узнает, какие молекулы и структуры стоят за передачей наследственной информацией и какими механизмами осуществляется эта передача. Часть контента позаимствована из блога автора Friends of Chromatin.

Генеральный партнер спецпроекта — компания «Диаэм».
Это крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Эпигенетика — это наука о наследовании не генов как таковых (это прерогатива «обычной» генетики), но паттернов активности разных участков генома, определяющих, какие гены в клетке будут активны (и, значит, повлияют на ее жизнь), а каким суждено молчать. И связано это не с последовательностью ДНК (с некоторыми оговорками [1], [2] ее можно считать неизменной), но с небольшими модификациями нуклеотидов; воздействием одних участков ДНК на другие; или «инкрустирующими» ДНК молекулами, меняющими активность генов.
В первой статье спецпроекта «Молекулы и эпигеном» [3] мы разобрались, что же в принципе входит в понятие «хроматин», помимо ДНК; потом (в публикации «Ядро и эпигеном» [4]) первый раз столкнулись с эпигенетическим наследованием; а теперь давайте попробуем обобщить, как такое наследование осуществляется. Условно можно выделить три основных «носителя» эпигенетики, каждый из которых наследуется отдельно (и главное — независимо от самой ДНК):
- Это может быть сама молекула ДНК, но только не составляющая ее последовательность нуклеотидов, а их химические модификации (главным образом, метилирование цитозина) или воздействия, которые оказывают некодирующие элементы (такие как промоторы, сайленсеры или энхансеры) на активность кодирующих белки и РНК участков — генов.
- Также на активность генов сильно влияют белки (например гистоны) и РНК хроматина. Среди последних можно назвать длинную некодирующую РНК Xist, ген которой находится в X-хромосоме, а сама она служит для дозовой компенсации, чтобы в женских клетках работала только одна такая хромосома, а не сразу обе. Ген Xist активен лишь на одной X-хромосоме из двух, и соответствующая РНК, экспрессируясь, облепляет хромосому и инактивирует ее, не давая работать остальным генам. Кстати, это пример, когда важен пространственный контекст, в котором находится ген: если Xist искусственно перенести в другую хромосому, он «заткнет» и ее [5], [6].
- Наконец, это могут быть внехроматиновые (и даже неядерные — цитоплазматические) молекулы — также белки и РНК. Они взаимодействуют с хроматином лишь время от времени, меняя его структуру и активность генов — так поступают некоторые малые РНК.
Как воспроизводятся и наследуются эпигенетические молекулы и метки?
Во-первых, во время репликации. ДНК удваивается, а значит, нужно удвоить и все эпигенетические модификации и молекулы и поместить их в правильные (обычно те же) места ДНК. Разумеется, всего этого сразу фермент для репликации — ДНК-полимераза — делать не умеет, так что тут в ход идут особые механизмы.
Во-вторых, во время деления клетки — митоза и мейоза. Структура хромосом (в том числе, эпигенетические метки) в это время меняется, и нужно «запомнить» организацию ядра в интерфазе и восстановить ее по окончании деления.
Отдельно следует рассмотреть наследование также играющих эпигенетическую роль малых РНК — просто потому, что они главным образом плавают в цитоплазме и наследуются по-своему, не заодно с ДНК.
Наконец, наследование может происходить уже в контексте целого организма — и тут передача признаков по наследству определяется не генетически, а эпигенетически.
Репликация: как скопировать не только ДНК, но и метилирование, гистоны с метками и прочие белки
Метилирование ДНК: безмолвные гены
Метилирование ДНК как генетический регулятор встречается в основном у эукариот. Фактически это просто навешивание метильных групп на азотистые основания нуклеотидов (в основном на цитозин, хотя бывает и аденин [7], [8]), не меняющее глобальной последовательности нуклеиновой кислоты. Это, пожалуй, самая изученная и важная модификация ДНК (хотя встречается еще гидроксиметилирование, формилирование и карбоксилирование [9]), ослабляющая или усиливающая распознавание генетического фрагмента белками, включающими чтение генов (транскрипцию), и таким образом управляющая активностью гена. Нужно понимать, что удвоение генетического материала (репликация) — копирование молекулы ДНК на базе принципа комплементарности — не воспроизводит таких модификаций, а значит, клетке требуется отдельная система наследования метильных меток.
Самая понятная функция метилирования — это выключение генов, если метилированы их промоторы [10]. У человека метилировано около 1% ДНК, но чаще всего (в 75% случаев) метилированы цитозины в последовательностях ЦГ (их даже называют CpG-динуклеотидами, а при затяжном повторении — ЦГ-островками ) и в перицентромерном хроматине . Паттерны метилирования меняются в ходе развития половых клеток, а также в эмбриогенезе — метилирование стирается, а потом восстанавливается заново при помощи de novo ДНК-метилтрансфераз.
Интересно, что такие ЦГ-островки часто встречаются в промоторах генов и внутри самих генов, так вот в первом случае они не метилированы (если это промотор активного гена — собственно, возможно, именно это и определяет активный промотор), а во втором — напротив, метилированы (а потому не работают как промоторы — и правда, какой же промотор посередине гена?) [11].
Насчет перицентромерного хроматина известно, что метилирование ДНК может вызывать характерную для него модификацию гистонов H3K9me2/3, и наоборот — так разные типы эпигенетических меток вызывают одна другую [12–14].
Есть два класса белков, метилирующих ДНК. Одни могут делать это заново (de novo метилтрансферазы), а другим (поддерживающие метилтрансферазы) узор метилирования нужно откуда-то «скопировать» — с другой, метилированной цепи ДНК. Это и становится ключом к наследованию паттерна метилирования.
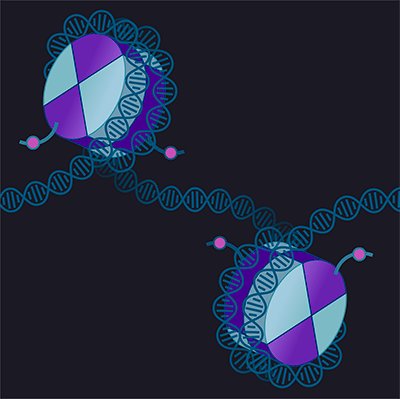
У млекопитающих поддерживающее метилирование проводит ДНК-метилтрансфераза 1 (Dnmt1). Во время удвоения ДНК она включается в работу репликативной вилки, где старая (материнская) цепь ДНК метилирована, а вновь синтезированная (дочерняя) — нет. Вот на вторую-то цепочку Dnmt1 и копирует узор метильных групп (рис. 1).
Любопытно, что так же поведет себя Dnmt1 и в пробирке: если ее добавить к двухцепочечной ДНК, где одна цепь метилирована, а вторая нет, этот фермент немедля займется восстановлением метилирования на второй цепи [14], [15].
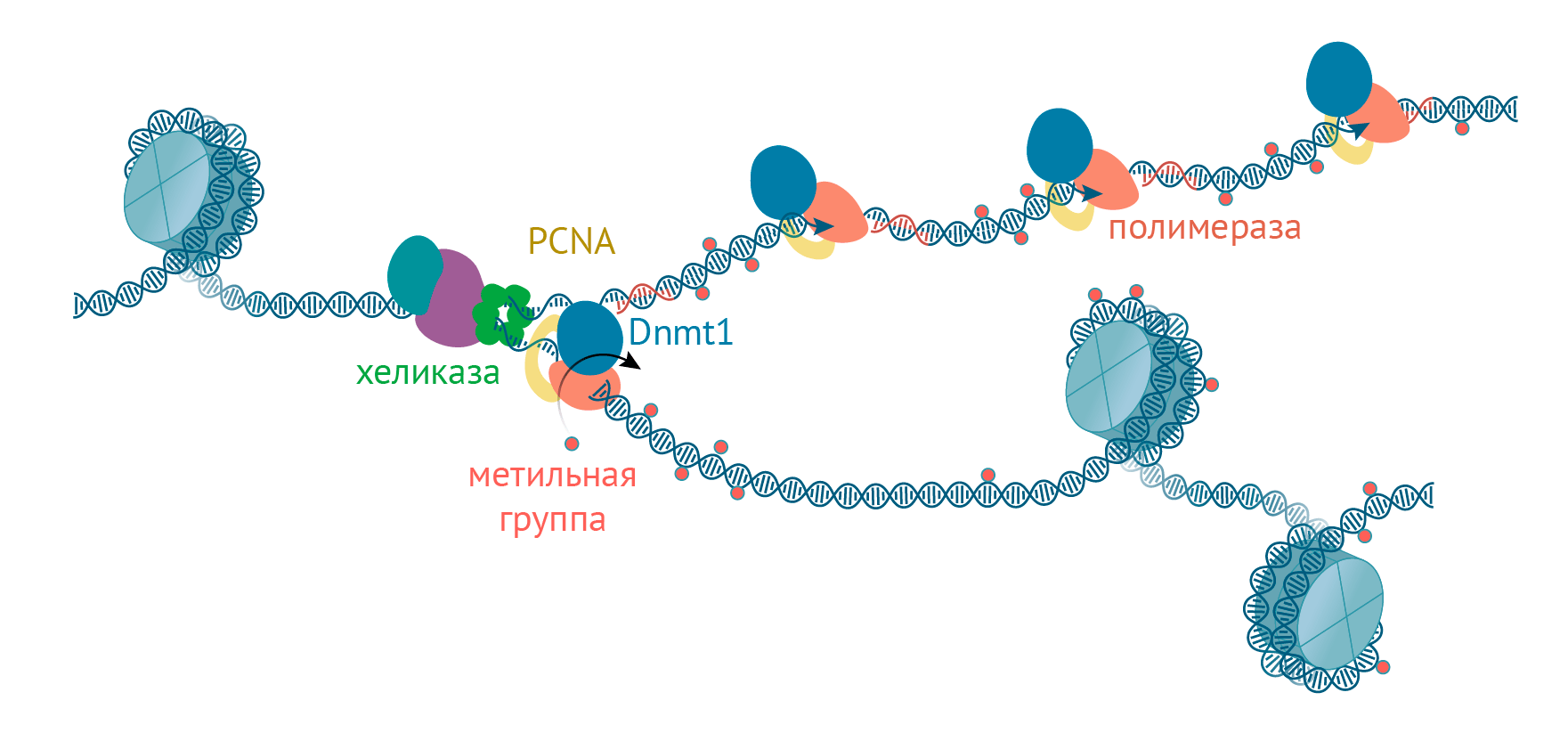
Рисунок 1. Поддержание метилирования ДНК во время репликации. Во время удвоения ДНК — репликации — к каждой из «родительских» цепей при помощи ДНК-полимеразы достраиваются «дочерние» цепи, образуя «репликативную вилку». Но метилирование ДНК — это эпигенетическая модификация, а значит, ДНК-полимераза просто так его не скопирует, и в составе вновь синтезированных двойных спиралей ДНК одна цепь будет метилирована, а вторая — нет. Чтобы скопировать метилирование и на дочерние цепи, в репликативный комплекс входит ДНК-метилтрансфераза Dnmt1, привлекаемая туда вспомогательным белком PCNA. Dnmt1 узнает метилированные места на родительских цепях ДНК и метилирует дочерние цепи примерно в тех же местах.
иллюстрация Михаила Гурьева
В отличие от поддерживающих, de novo ДНК-метилтрансферазы играют роль не при репликации, а во время развития половых клеток и эмбриона [16], когда паттерны метилирования стираются. Как именно эти ферменты определяют, что именно нужно метилировать, — толком пока что не понятно, по крайней мере у млекопитающих.
А вот у растений в этом процессе помогает РНК по механизму, напоминающему образование гетерохроматина у дрожжей (см. «Ядро и эпигеном» [4]). У растений есть ферменты РНК-полимеразы IV и V, которые могут читать некодирующие области ДНК, где уже есть репрессивные эпигенетические метки. Синтезированная цепочка РНК конвертируется в двуцепочечную РНК-полимеразой и нарезается белком Dicer на небольшие (около 20 остатков) двухцепочечные фрагменты. Такие фрагменты связываются с белком Argonaute, оставляющим для своих целей только одну цепочку из двух. А цель вот какая: Argonaute направляется к длинной РНК, синтезируемой РНК-полимеразой IV/V и все еще расположенной вблизи ДНК, и, используя короткий фрагмент РНК как «прицел», позиционируется возле комплементарного участка длинной РНК. Ну а дальше Argonaute привлекает de novo ДНК-метилтрансферазу на близлежащие места ДНК (помним, что «наводку» дал короткий фрагмент РНК), и фермент метилирует, например, промоторы генов, которые таким образом дополнительно выключаются [17]. То есть, хотя такие метилтрансферазы и формально относят к классу de novo, по сути они поддерживают репрессивный хроматин.
Интересно, что у гриба Cryptococcus neoformas есть только поддерживающий метилирование белок, а вот ген de novo метилтрансферазы, похоже, был утерян 50–150 миллионов лет назад. Соответственно, нового метилирования у этого гриба не возникает, и лишь поддерживается старое, передающееся по наследству.
Гистоны и их варианты: быстро пересобрать нуклеосомы, потом настроить
Нуклеосомы — октамеры из гистонов (по два каждого из типов H2А, H2B, H3 и H4), на которые, подобно нитке на катушку, наматывается ДНК (только она делает всего около полутора оборотов на одной нуклеосоме, а потому таких катушек очень-очень много [20]). N-концевые части гистонов торчат из нуклеосом и могут быть модифицированы — метилированы, ацетилированы, фосфорилированы и так далее. Такие эпигенетические метки (а это совершенно конкретные модификации на абсолютно конкретных аминокислотных остатках) узнаются специальными белками, которые влияют на хроматин и могут усиливать или ослаблять транскрипцию генов.
Нуклеосомы — серьезное препятствие для репликативной вилки, а потому для копирования ДНК их нужно временно разобрать, и потом собрать вновь. Сборкой и разборкой нуклеосом занимаются специальные белки, называемые шаперонами гистонов. Некоторые из них взаимодействуют с репликативной вилкой, а некоторые передают гистоны шаперонам первого типа по цепочке «из рук в руки». Сами гистоны во время репликации активно синтезируются (ведь удваивается ДНК, а значит, и остальной хроматин подлежит удвоению), а потому вновь собранные нуклеосомы состоят из смеси старых и новых гистонов (рис. 2) [21], [22].
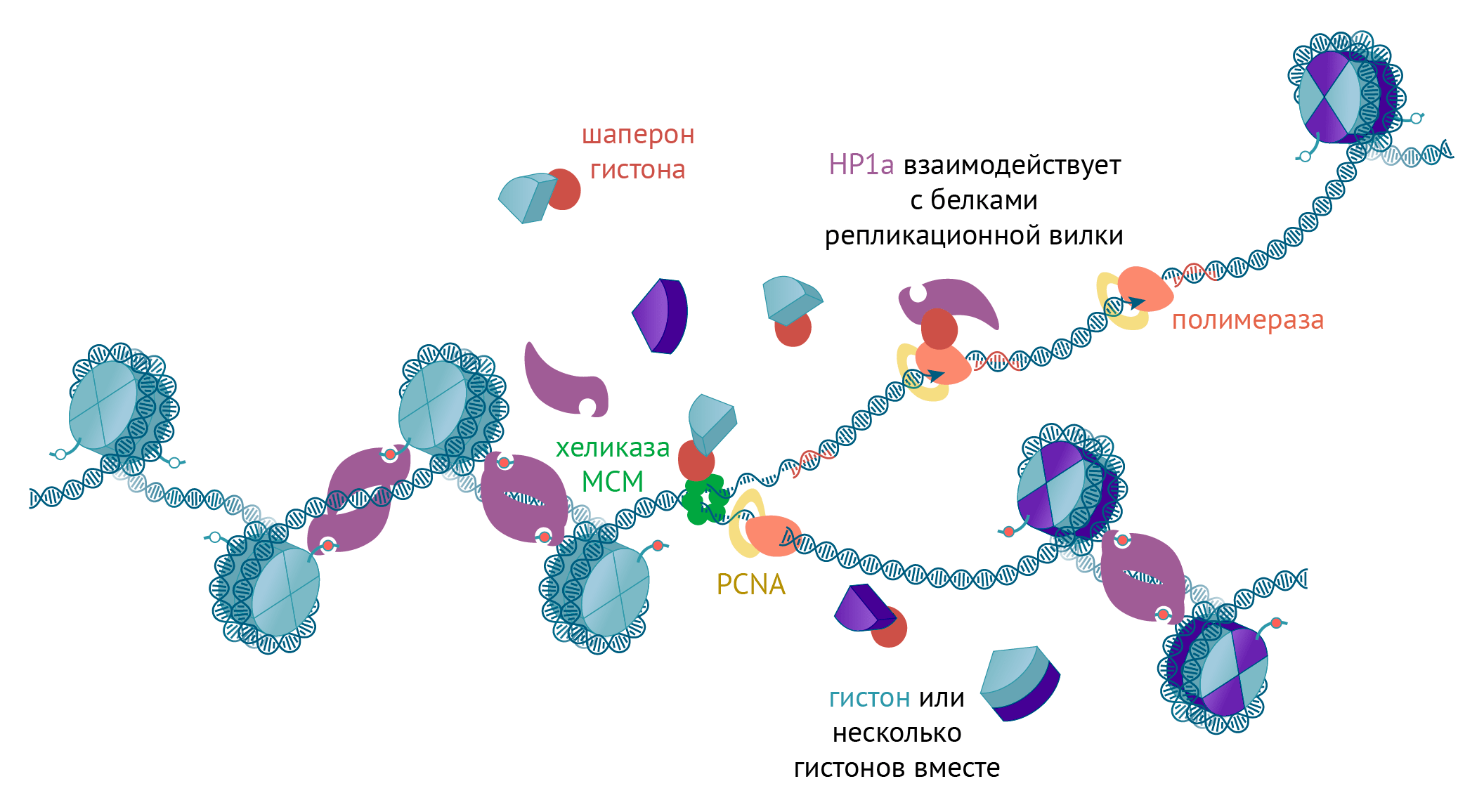
Рисунок 2. Наследование гистонов и негистоновых белков во время репликации. Новые нуклеосомы собираются из старых и новых гистонов, и по крайней мере некоторые негистоновые белки перераспределяются на дочерние цепи, взаимодействуя с белками репликативной вилки.
иллюстрация Михаила Гурьева
Интересно, что в сортировке гистонов задействованы не только классические шапероны, но и белки с совершенно другими на первый взгляд функциями. Например, хеликаза MCM2, расплетающая двойную спираль ДНК на отдельные цепи, чтобы их можно было скопировать, внезапно оказывается способна связывать гистоны (конкретнее, H3/H4-тетрамер). MCM2 может «шаперонить» как старые, так и новые гистоны, а еще центромерный вариант H3 — CenpA [23].
Занятно, что некоторые варианты гистонов вставляются в нуклеосомы во время репликации «насовсем», а некоторые лишь на время, и позже заменяются другими. Например, у гистона H3 есть три «классических» варианта — H3.1, H3.2 и H3.3. Однако при сборке новых нуклеосом используются только первые два из них, и H3.1 уже после репликации может заменяться на H3.3 и CenpA [24]. Аналогично, H2A может заменяться на H2A.X (участвует в репарации ДНК) и H2A.Z, задействованный в регуляции транскрипции: нуклеосомы с H2A.Z часто располагаются на промоторах и энхансерах . За первичную сборку нуклеосомы и за дальнейшую «замену» гистонов отвечают разные шапероны.
Разные варианты гистонов существуют не просто так: они располагаются в различных участках генома, и размещают их туда, как правило, шапероны. Например, шапероны H2A.Z распознают промоторы и энхансеры, куда и помещают свои гистоны. Распознавание это устроено по-разному: шапероны могут взаимодействовать с транскрипционными факторами, а могут узнавать гистоновые метки на промоторах и энхансерах [25].
Как наследуются модификации гистонов? Во-первых, модификации «старых» гистонов сохраняются, а сами они встраиваются в новые нуклеосомы, как правило, не далее 400 нуклеотидов от начального положения [26], [27]. Во-вторых, некоторые гистоновые модификации распространяются на соседние нуклеосомы — при участии тех самых белков, которые исходно навешивают эти модификации , [27], [28], — и именно так гистоновые метки восстанавливаются на вновь синтезированных и только что встроенных в нуклеосому гистонах после репликации ДНК.
Этот эффект наблюдали для метки самого компактного гетерохроматина H3K9me2/3 и метки polycomb-хроматина H3K27me3 (см. статью «Ядро и эпигеном» [4]).
Наследование гистонов — сохранение их в нуклеосомах с исходными модификациями. Ведь, поскольку гистоны в нуклеосомах заменяются, модификации постепенно пропадают. Наследование гистонов неоднородно по всему ядру и зависит от пространственного расположения ДНК, (вероятно) от компактности хроматина и даже от активности генов.
- У дрожжей S. pombe гистоны лучше наследуются на периферии ядра, чем в центре (то есть медленнее обмениваются в нуклеосомах).
- То же можно сказать и про компактный гетерохроматин по сравнению с более «рыхлым». Возможно, это связано с меньшей доступностью плотного гетерохроматина для шаперонов, которые не могут так же эффективно заменять гистоны в нуклеосомах [29].
- Гистоны в нуклеосомах заменяются быстрее в активных, чем в неактивных генах — причем, активация гена ускоряет и замену гистонов. Активные процессы в хроматине также ускоряют замену гистонов — например, при репликации нуклеосомы то и дело разбираются и собираются обратно [30], а это ускоряет обмен гистонов. В довершение картины, гистоны быстрее заменяются на промоторах и инсуляторах, чем на самих генах — возможно, различные белки здесь делают хроматин еще менее компактным.
Гистоны наследуются в контексте хроматиновых доменов
Итак, мы поняли, что гистоны в активном хроматине заменяются гораздо быстрее, чем в неактивном [30]. Наблюдения за только что удвоенным хроматином в сравнении со старым позволили предположить, что хроматиновые участки наследуются в виде доменов — разные гистоновые модификации располагаются в разных областях ДНК, перекрывающихся только частично.
В предыдущей статье цикла мы подробно останавливались на том, что такое хроматиновые домены, и почему одни фрагменты генетической информации вообще стоит отличать от других по свойствам: «Ядро и эпигеном» [4].
Измерения показали, что на новосинтезированных гистонах метки устанавливались поэтапно, причем метка активного хроматина — H3K36me — устанавливалась намного быстрее неактивной метки — H3K27me. Более того, эти две метки как бы противостоят друг другу. Если заблокировать белок, навешивающий метильную группу на 27-й лизин гистона H3, то не просто уменьшится доля метки H3K27me, но метка H3K36me автоматически возрастет [28], подчеркивая антагонистический характер распространения этих двух гистоновых модификаций по хроматину. Из этого и можно заключить, что гистоновые модификации распространяются по хроматину в виде доменов, пока не сталкиваются с другими, несовместимыми с ними метками.
Наследование других белков во время репликации
Интересно, что во время удвоения ДНК некоторые белки репликативной вилки могут принимать активное участие в перераспределении не только гистонов, но и негистоновых белков с родительского дуплекса ДНК на дочерние. Так, главный белок гетерохроматина HP1a взаимодействует с шапероном CAF-1 (фактором сборки хроматина 1), а тот в свою очередь с одним из главных белков репликативной вилки — PCNA (см. рис. 2). По уже частично подтвержденной гипотезе, CAF-1 сидит на репликативной вилке и перераспределяет молекулы HP1a c родительского дуплекса ДНК на дочерние цепи. Есть и другие негистоновые белки, взаимодействующие с CAF-1 [21], [31].
Какие белки важны для наследования хроматина?
По самой общей классификации есть два типа хроматина — рыхлый эухроматин , где активно читаются гены; и компактный гетерохроматин , где транскрипция по большей части выключена. При репликации ДНК и делении клетки нужно «запомнить», какие гены читаются, а какие нет, чтобы потом восстановить эту картину. А значит, надо запомнить, какие гистоновые модификации, РНК и белки присутствуют в определенных участках генома. Как правило, этот набор признаков сильно различается между разными фрагментами ядра и ДНК, которые получили название хроматиновых доменов — мы посвятили этому практически всю предыдущую статью цикла [4].
Как мы помним из предыдущей статьи «Ядро и эпигеном» [4], в эухроматине много активных генов, гистоны промоторов которых несут метку H3K4me3, а сами гены — H3K36me3. Также в активном хроматине гистоны ацетилированы.
Гетерохроматин бывает конститутивный и факультативный. Первый несет метку H3K9me2/3 и белки: HP1a, связывающий эту метку и увеличивающий компактность такого гетерохроматина, и Su(var)3-9, который эту метку навешивает. Он практически не содержит генов и образует такие хромосомные структуры как центромеры, теломеры или ядрышковые организаторы. Признаки факультативного гетерохроматина — H3K27me3 и белки группы polycomb. Этот хроматин определяет молчание тканеспецифичных генов, то есть в разных клетках его образуют разные участки ДНК.
Так вот клетке приходится копировать не просто ДНК, но как бы домены целиком, поддерживая сложившийся там набор модификаций и молекул. Выше мы разобрали общие механизмы наследования метилирования ДНК, гистонов и негистоновых белков, а теперь давайте обсудим формирование и воспроизведение доменов эу- и гетерохроматина у дрожжей — что поделать, иногда молекулярная биология изучена только на каком-нибудь модельном организме [32] …
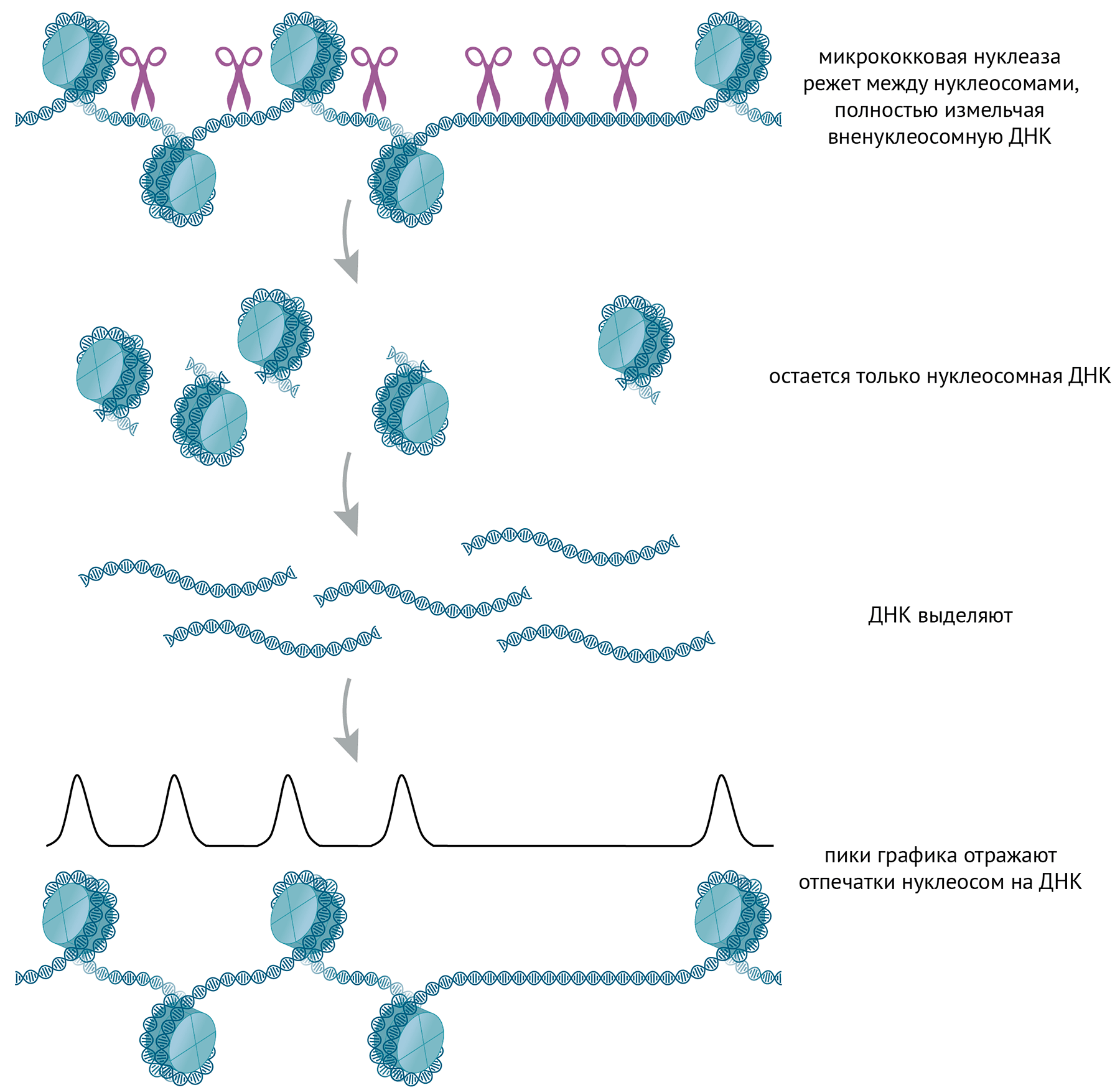
Геномом дрожжей легко манипулировать: создавать штаммы, мутантные по одному или нескольким генам, и определять, какие белки важны для правильной упаковки хроматина (рис. 3) [33]. Используя эти технологии, ученые выяснили, что в формировании (и поддержании) гетерохроматина обыкновенных делящихся дрожжей задействован каскад на основе малых РНК, похожий на путь метилирования ДНК в растениях. А именно, в деле оказался замешан белок Dicer — «мастер над киРНК». Малые РНК пускают в ход механизмы, формирующие и поддерживающие определенные гистоновые модификации, задающие свойства гетерохроматина.
Мы уже рассматривали этот механизм в предыдущей статье «Ядро и эпигеном» [4]. Напомним, о чем речь: Парадоксальным образом, в «неактивном» гетерохроматине (например, в перицентромерных участках) сохраняется базовая транскрипция, однако считывающиеся оттуда РНК не кодируют белков и не транслируются. Вместо этого, в силу того, что они читаются в обеих ориентациях, они принимают двухцепочечную форму, играющую в клетке особую роль. Такие двухцепочечные участки нарезаются на маленькие (около 20 звеньев) фрагменты, связываемые особым белком — аргонавтом (Argonaute). Оставив лишь одну цепь РНК, Argonaute использует ее как «прицел», комплементарно присоединяясь к считывающейся с центромер РНК. Укрепившись на ней, Argonaute привлекает метилтрансферазу, навешивающую на ДНК метку неактивного гетерохроматина — H3K9me3.
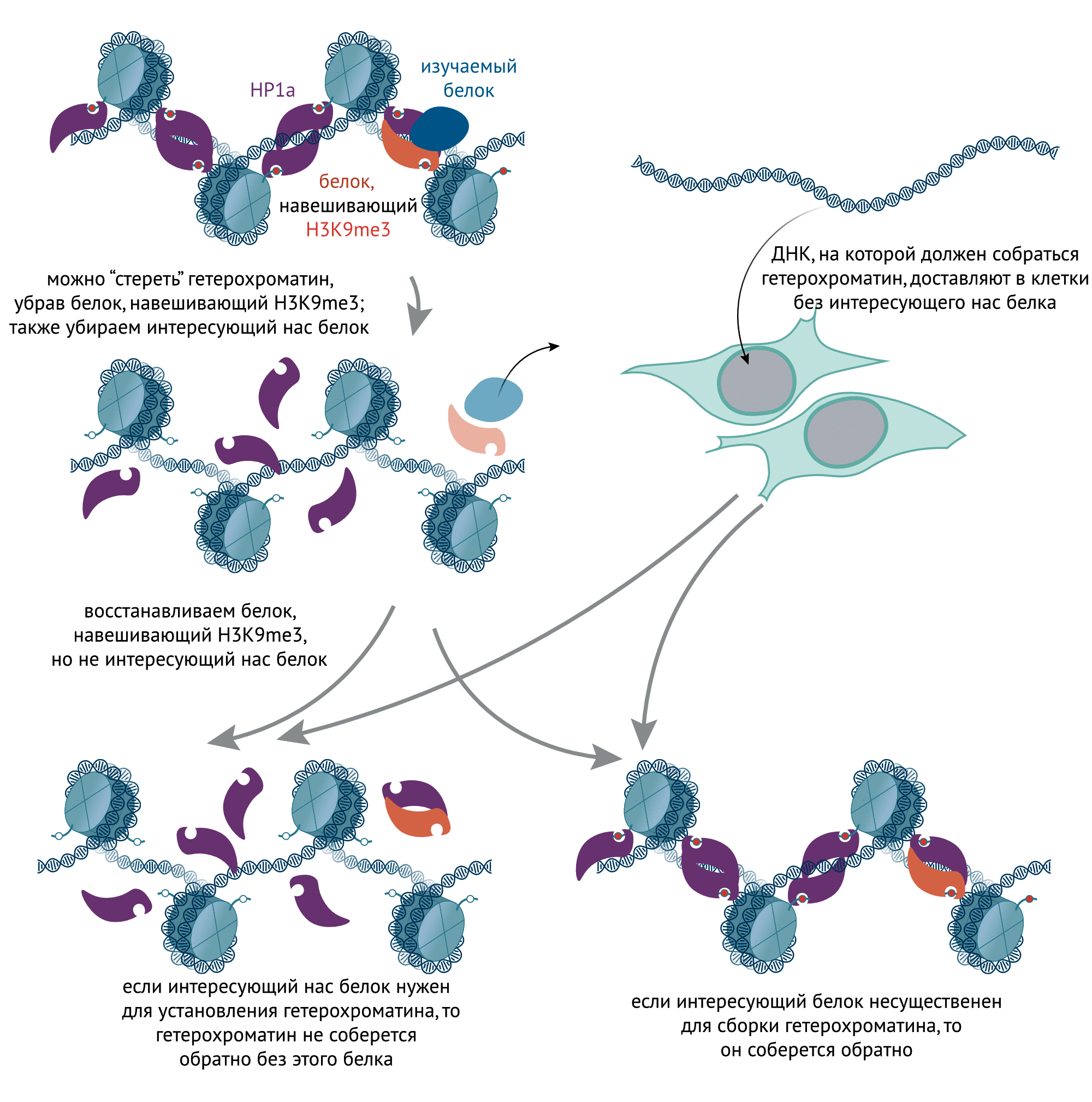
Рисунок 3. Какие белки нужны для формирования и поддержания гетерохроматина в дрожжах? Уже установлено, что формирование и поддержание гетерохроматина (определяемого во многом гистоновой меткой H3K9me3 и белками HP1a и Su(var)3-9) могут идти разными путями. Чтобы установить важность того или иного белка для поддержания гетерохроматина, можно провести нокаут этого белка и отследить (методом ChIP-секвенирования) количество метки H3K9me3: если оно начало снижаться — то белок важен. Чтобы ответить на вопрос, необходим ли ген для формирования гетерохроматина, нужны дополнительные эксперименты. Потребуется не только выключить интересующий ген, но и на время «стереть» весь гетерохроматин из клеток, дополнительно инактивировав ключевой для формирования гетерохроматина белок Su(var)3-9. Потом его включают обратно и смотрят: восстанавливается ли гетерохроматин? Если восстанавливается — значит, наш ген не важен для формирования гетерохроматина, ну а если нет — то важен. Можно устроить и другую проверку: поместить в клетки дикого типа и мутантные клетки ДНК, на которой может собираться гетерохроматин, и посмотреть, будет ли он там собираться или нет. С помощью подобных экспериментов стало известно, что для формирования (и иногда — поддержания) гетерохроматина на центромерах S. pombe важны белки, которые связываются с малыми РНК.
иллюстрация Михаила Гурьева по [33]
Оказалось, что механизмы наследования гетерохроматина немного различаются не только от домена к домену, но и даже в «естественном» и «искусственно созданном» гетерохроматине [34]. Роль малых РНК в процессе еще предстоит до конца осознать, но, видимо, они играют ее не только во время репликации, но и в течение всей интерфазы, когда работает транскрипция.
Наследование активного хроматина — это отдельная песня, слова которой известны гораздо хуже. Его можно инициировать, если искусственно снизить активность белка, снимающего ацетильные метки с гистонов [35] (признак активного хроматина) или удалить специальный регуляторный участок ДНК [36–38]. Однако естественные механизмы наследования активного хроматина стали исследовать только недавно.
Например, у пекарских дрожжей открыт совершенно неожиданный механизм, использующий свойства белков-прионов [38], приводящих у млекопитающих к весьма неприятным заболеваниям [39]. В этом случае активный хроматин формируется и поддерживается не модификацией нуклеосом, а наследственной передачей этих самых прионов, которые могут менять конформацию сами и «заставлять» это делать другие молекулы (по сути, вызывать фазовый переход: рис. 4).
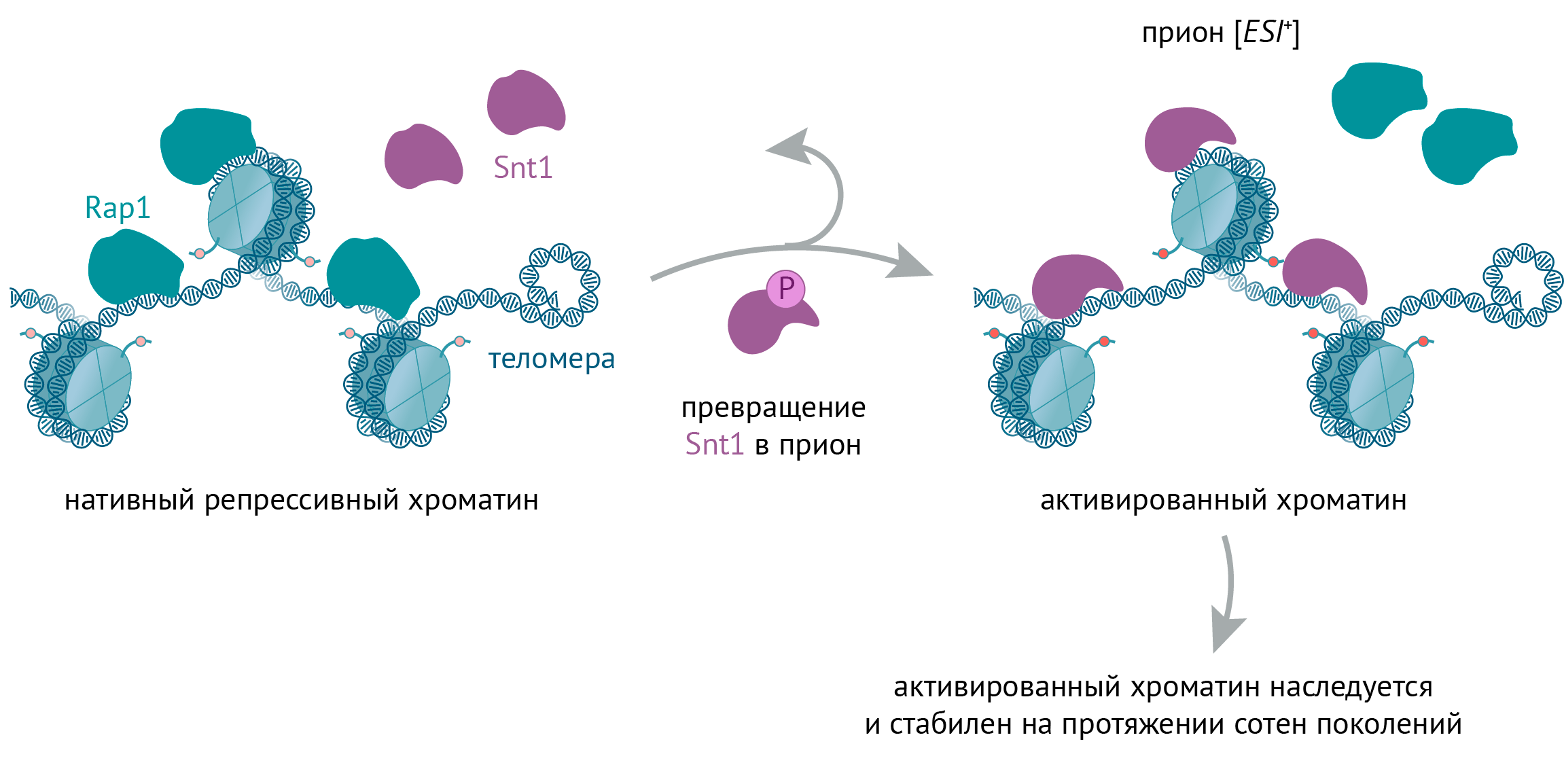
Рисунок 4. Активный хроматин дрожжей может наследоваться с помощью прионов. (Сразу напомним: прионы обладают минимум двумя конформациями, одна из которых «заразна»: конвертирует другие молекулы в «заразную» форму при встрече.) У дрожжей при неблагоприятных условиях клеточный цикл может остановиться, и тогда специальный белок-регулятор фосфорилирует хроматиновый белок Snt1, превращая его в прион. В норме этот белок сидит на хроматине в комплексе с гистонацетилтрансферазой, навешивающей ацетильные метки на гистоны и тем самым активирующей хроматин. А прионная форма Snt1 изменяет структуру всего комплекса: гистоны сильнее ацетилируются, в результате чего на хроматин садятся транскрипционные белки и гены читаются активнее. Интересно, что на концах хромосом — теломерах — этого комплекса в норме нет. Но Snt1-прион туда с удовольствием садится и генерирует активный хроматин и там. Важно, что Snt1-прион может передаваться по наследству через много-много поколений и продолжать вызывать эпигенетические изменения. Ну а дрожжи от этого адаптируются к неблагоприятным условиям и продолжают жить-поживать.
иллюстрация Михаила Гурьева по [38]
К сожалению, помимо этого захватывающего механизма у дрожжей, наследование активного хроматина почти не изучено. Непонятен механизм восстановления активных гистоновых модификаций после репликации. Известно, правда, что после репликации и митоза некоторые белки активного хроматина (транскрипционные факторы) распознают нужные последовательности ДНК, другие белки распознают гистоновые модификации, но более частные, конкретные механизмы еще только предстоит изучить.
Наследование позиций нуклеосом: вас тут не стояло!
На некоторых участках ДНК (например, на промоторах, энхансерах и инсуляторах) нуклеосомы расположены строго организованно — это важно для правильной транскрипции. К примеру, в точке начала транскрипции на промоторе обычно есть участок, свободный от нуклеосом, а уже от его краев нуклеосомы расположены «стройными рядами» (то есть с равномерными интервалами). Позиционируют нуклеосомы на ДНК белки, занимающиеся ремоделированием хроматина.
Интересно, что на активных промоторах и энхансерах свежереплицированного хроматина позиции нуклеосом сразу же после репликации довольно хаотичны, но восстанавливаются спустя минуты после прохода репликативной вилки [40]. Вероятно, восстановлением должного порядка нуклеосом занимаются специальные белки, в том числе транскрипционные факторы, отмечающие места, где нужно позиционировать нуклеосомы определенным образом (про один из таких белков — CTCF — рассказывается в следующей главе); и ремоделеры хроматина, непосредственно двигающие нуклеосомы. По-видимому, строгой «памяти» у нуклеосом нет; однако есть их склонность к AT-богатым участкам ДНК; взаимное их позиционирование по отношению к транскрипционным факторам, распознающим свои сайты на ДНК, и к сайту начала транскрипции; а также есть оптимальное расстояние между позиционированными нуклеосомами, которое и поддерживается ремоделерами хроматина, — и такой «порядок на местах» важен для правильного связывания с хроматином репликативных белков [41], [42].
Получается, что не только наследование определяет позиционирование нуклеосом на ДНК, но и упорядоченное положение нуклеосом важно для наследования.
Наследование во время митоза
А репликацией наследование не заканчивается. Вернее, ей оно только начинается — за стадией удвоения ДНК следует деление клетки — из курса школьной биологии известно, что это либо мейоз (в котором образуются только половые клетки), либо митоз, которым делятся все остальные (то есть соматические) клетки.
Итак, митоз следует за репликацией, и тут уже происходит масса разных процессов, меняющих облик хромосом — в частности, они «конденсируются» (приобретают оптически плотный вид и становятся видны в микроскоп), а в самой клетке появляется веретено деления. На молекулярном уровне мы увидим, что ТАДы (топологически ассоциированные домены хромосом) исчезают, конденсины упаковывают хромосомы в ряды петель, которые исходят из остова конденсина и топоизомеразы II, и покрыты оболочкой из белков и РНК [43]. Многие хроматиновые белки отсоединяются и уплывают в цитоплазму (или присоединяются к РНК-белковой оболочке хромосом). А потом, уже после митоза, садятся обратно на правильные места принявших свою исходную (интерфазную) форму хромосом.
Гистоны тоже меняют состояние — в частности, деацетилируются, что, видимо, служит для компактизации митотического хроматина. Детали этого процесса [44] пока неизвестны, но, возможно, им управляют киназы, фосфорилирующие белки (как правило, это меняет их активность, а в данном случае может придать специальные митотические свойства). Фосфорилированию могут подвергаться белки, модифицирующие гистоны, а это уже изменит «гистоновый код» в целом.
Метилирование ДНК во время митоза не изменяется (вся карта метилирования остается такой же, какая досталась с этапа репликации ДНК), но транскрипция полностью выключается, за исключением центромер — только там и продолжает работать РНК-полимераза [45]. И нужно запомнить, какие гены транскрибировались в интерфазе, чтобы включить их обратно, когда придет время превратить митотические хромосомы обратно в интерфазные.
Если обобщить, деление клетки сопровождается масштабной перестройкой хроматина, частично как бы «стирающей» почти весь эпигенетический статус. И крайне важно этот статус запомнить, а потом восстановить — это позволит дочерним клеткам походить на материнскую, как и положено потомкам.
Гистоновое наследство
Как уже было сказано, в митозе модификации гистонов изменяются [44]. Но не все, и те, что остаются, по-видимому, нужны для повторной активации генов. Дело в том, что во время митоза транскрипция большей части генов выключается, и ее надо снова включить после того, как клетка поделится. Однако механизмы «запоминания», какие гены надо включить после деления, изучены все еще довольно поверхностно. Чтобы изучить это, ученые провели эксперимент на плюрипотентных стволовых клетках мыши, у которых за митозом следует очень короткая фаза клеточного цикла G1, а значит, транскрипция обязана восстановиться крайней быстро. Оказалось, что после митоза быстрее всего включаются в работу гены, активные именно в плюрипотентных клетках, и для каждого из этих генов выполнялось как минимум одно условие из следующих:
- ацетилирование 27-го остатка лизина в гистоне H3 (H3K27ac);
- высокая доступность хроматина;
- наличие сайта связывания фактора транскрипции TPB (TATA-связывающего белка).
Роль гистоновых модификаций в этом процессе удалось подчеркнуть, ингибировав в митозе белок, ацетилирующий H3K27: в этом случае после деления клетки гены активировались куда менее охотно [47]. Получается, что определенные белки и гистоновые модификации могут оставаться на хромосомах в митозе и помогать заново включить гены в интерфазе.
Наследование позиций нуклеосом: волнующее путешествие на митотических хромосомах
В конце предыдущей главы мы уже обращали внимание, что во многих случаях важно, чтобы расположение нуклеосом было не хаотичным, а упорядоченным, иначе многие (эпи)генетические процессы могут идти неправильно [48], [49]. Однако там речь шла про восстановление позиций нуклеосом после репликации, а здесь мы расскажем об аналогичном процессе после деления всей клетки.
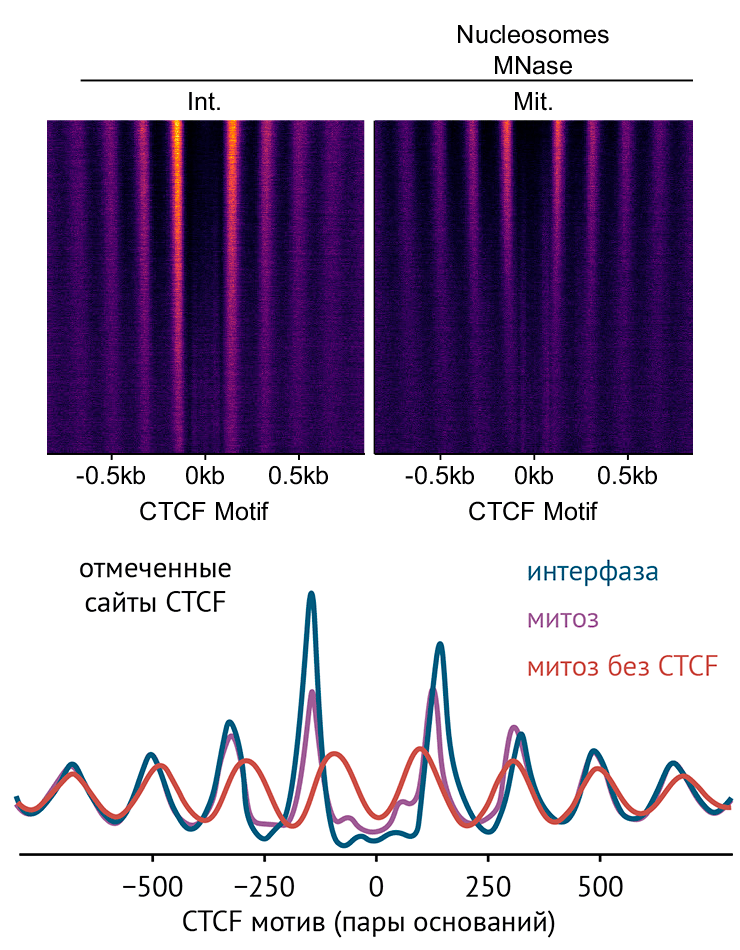
Один из белков, позиционирующих нуклеосомы в митозе, — CTCF, связывающийся с инсуляторами. Да-да, тот самый CTCF, на котором заканчиваются хроматиновые петли, когда об него «спотыкаются» когезины, эти петли вытягивающие. Под CTCF нуклеосом нет, а вот по обе стороны от него нуклеосомы не просто есть, но и строго упорядочены — расстояние между ними практически совпадает. Это свойство «строить» нуклеосомы, похоже, и используется в митозе: по мере деления клетки почти вся ДНК «молчит», и экспрессируются только небольшие фрагменты генома вроде центромер [45], [50]. А в прочих местах CTCF сохраняет позиции нуклеосом в порядке, чтобы по окончании деления гены могли быстро и правильно включиться (рис. 5, 6) [51].
Наследование малых РНК
Если судить исключительно по школьному учебнику, существует всего три разновидности молекул РНК: мРНК, перекидывающая информационный мостик между ДНК и белками; тРНК, реализующая генетический ход по мере трансляции; и рРНК, образующая функциональную основу рибосомы и вообще всего процесса синтеза белка. Однако в действительности разновидностей этой молекулы гораздо больше [52]. Особо важный класс составляют малые РНК длиной всего около 20 нуклеотидных остатков, на которых работают молекулярный механизм РНК-интерференции [52] и связанные пути регуляции активности генов. В частности, малые РНК могут присоединяться к хроматину и менять его структуру, «наводя» на него разные молекулярные комплексы, причем не только «в моменте», но еще и передаваясь по наследству!
киРНК — снайперская точность
Один из самых интересных подтипов малых РНК — короткие интерферирующие РНК (киРНК), знаменитые своим участием в РНК-интерференции. Они могут самостоятельно размножаться в цитоплазме с помощью ферментов и передаваться по наследству через много-много поколений, а еще вызывать эпигенетические изменения. Один из таких ферментов — РНК-зависимая РНК-полимераза — встречается только у некоторых изученных организмов: у дрожжей S. pombe [32], червячка C. elegans [53] (здесь РНК нужны для правильного развития [54] и передаются через много поколений) и растений (а вот их киРНК защищают от патогенов [55], [56]). Хотя такие РНК и имеют отношение к хроматину, наследуются они вне него: просто передаваясь через цитоплазму [57]. Давайте разберемся, каков же механизм действия таких РНК (рис. 7).
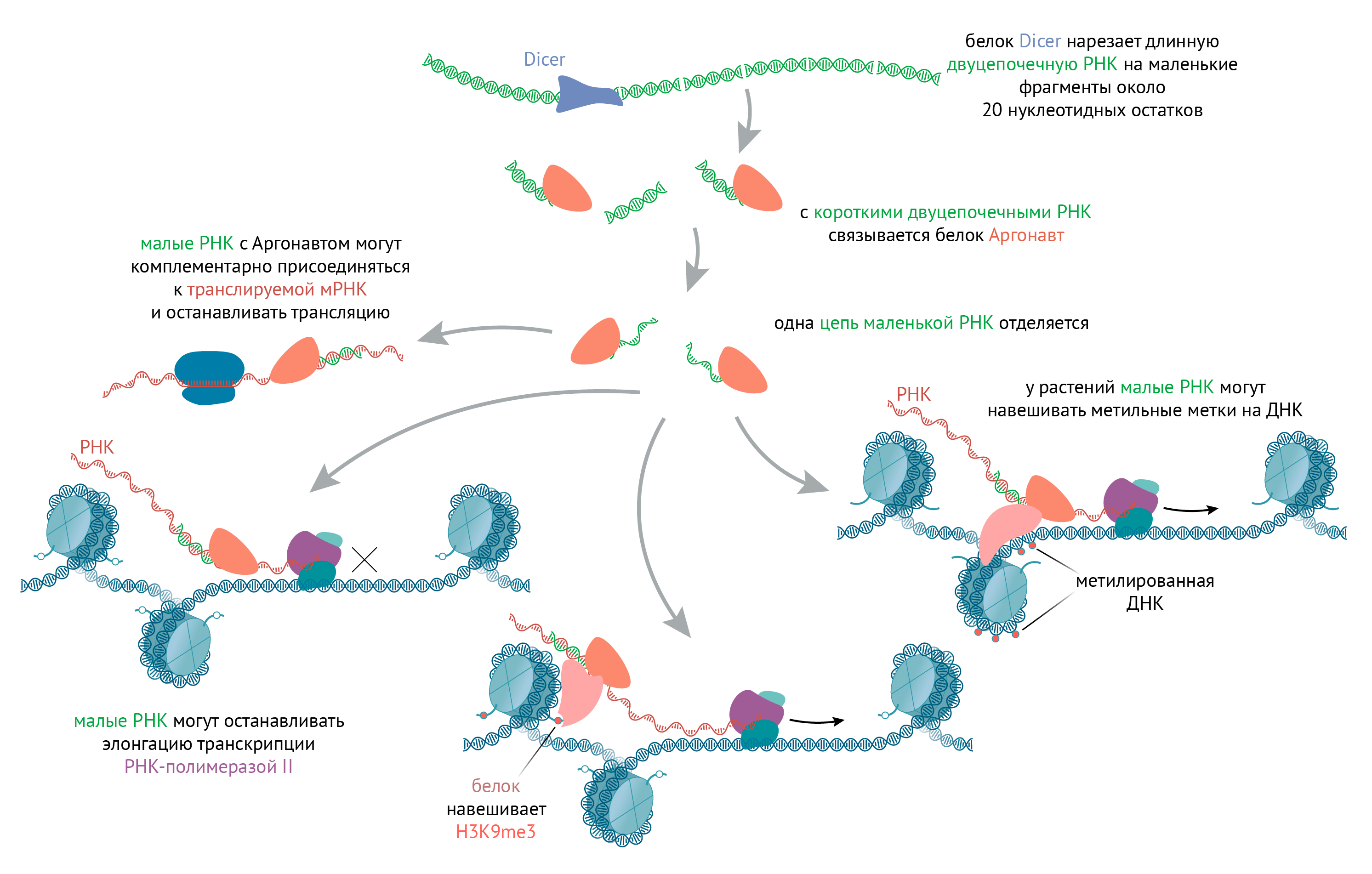
Рисунок 7. Как РНК-интерференция (и малые РНК) влияют на транскрипцию, трансляцию и хроматин. Иногда двухцепочечная РНК, которая появляется в клетке, может нарезаться на малые фрагменты. Это может быть либо когда ДНК читается сразу в обоих направлениях, и получаются комплементарные молекулы; либо когда внутри одной РНК есть палиндромы, и она формирует самокомплементарные «шпильки». дцРНК процессируются также, как и при наводке метилирования ДНК и гетерохроматина дрожжей: белками Dicer и Argonaute, причем последний оставляет для своих целей только одну цепь РНК, направляющую его подобно прицелу на рибосомы (в цитоплазме) или на хроматин (в ядре). Маленький кусочек РНК, связанный с Argonaute, комплементарно присоединяется к РНК гена. Если эта РНК транслируется на рибосомах, то комплекс останавливает ее трансляцию; если дело происходит в ядре, то тормозится фермент, осуществляющий транскрипцию, — РНК-полимераза II. А еще Argonaute может привлекать к месту действия белки, формирующие гетерохроматин или метилирующие ДНК (у растений).
иллюстрация Михаила Гурьева
В C. elegans и дрожжах киРНК могут не просто привести к модификации гистонов по типу H3K9me3 (напомним, это метка «молчащих» генов), но и самостоятельно удвоиться и передаться по наследству, чтобы делать гетерохроматин и останавливать трансляцию уже там. У растений же киРНК обеспечивают защиту от патогенов: геном внедрившегося РНК-вируса будет порезан на 20-членные фрагменты, которые скопируются и распространятся по всему растению, «выключив» вирусные гены [58].
Интересно, что у млекопитающих РНК-зависимой РНК-полимеразы нет, но киРНК все равно влияют на трансляцию. Правда, передаваться по наследству они уже не смогут.
пиРНК — каратели транспозонов
Есть похожие на киРНК молекулы, производящиеся только в гонадах и направленно защищающие геном от перемещающихся элементов ДНК — ретротранспозонов [18], а иногда и вирусов [59]. Имя им пиРНК (по названию белков PIWI, с которыми они взаимодействуют) [59]. Команда пиРНК/PIWI не дают ретротранспозонам захламлять наш геном, отлавливая их на стадии РНК, пока они не успели вновь встроиться в хромосомы. Интересно, что, как и киРНК у некоторых организмов, пиРНК могут самовоспроизводиться, вот только в этом механизме нет прямого синтеза РНК с РНК-матрицы (рис. 8). Недавно было показано, что пиРНК могут инициировать, но не поддерживать, наследуемое замалчивание генов [60]. То есть пиРНК ведут себя похоже на другие киРНК, но не совсем — последние важны и для поддержания хроматина, а не только инициации.
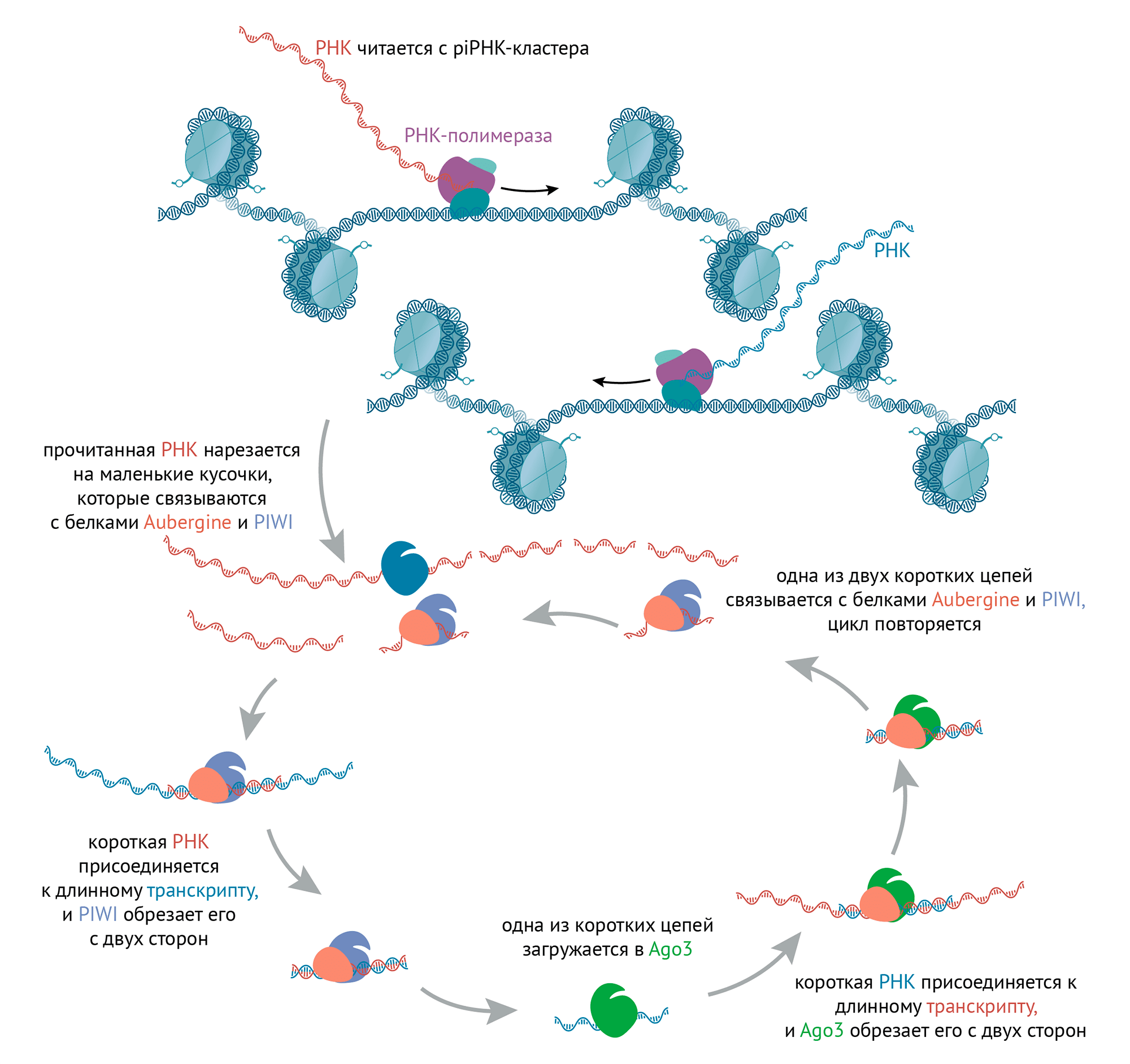
Рисунок 8. «Размножение» пиРНК по механизму пинг-понга. Сначала предшественники пиРНК считываются со специально предназначенных для этого мест в геноме — кластеров пиРНК. Потом они нарезаются на маленькие одноцепочечные пиРНК и связываются с белками Aubergine (с англ. — «баклажан») и PIWI (не спрашивайте, почему биологи так называют белки хроматина!). Комплекс из маленькой одноцепочечной РНК, Aubergine и PIWI связывается с комплементарной РНК транспозона и обрезает ее с двух сторон от двухцепочечного участка. Иначе говоря, комплекс вырезает из транспозона РНК, которая по комплементарности идеально подходит к пиРНК. Получается маленькая двухцепочечная РНК. Цепь, которая является вырезанным кусочком РНК транспозона, отделяется и загружается в белок Ago3 (Argonaute 3). Такой комплекс из куска транспозона и Ago3 комплементарно опять присоединяется к транспозону и вырезает из него комплементарный кусок. И получается больше пиРНК. Поскольку кусочки РНК, вырезаемые из транспозонов, передаются между комплексом из Aubergine и PIWI и белком Ago3 туда-обратно, механизм напомнил ученым игру в пинг-понг, и они его так и назвали: механизм пинг-понга.
иллюстрация Михаила Гурьева по [57]
От родителей к детям: мамин и папин метилом
Наследование в понимании обычного человека сильно отличается от картины, которую рисует себе молекулярный биолог — тут речь идет о родственниках и детях, а вовсе не о репликации, митозе или, прости господи, мейозе. Но дело здесь не только в барьере понимания разных людей — чтобы (эпи)генетические признаки передались по наследству, им придется преодолеть также барьеры, воздвигаемые половыми клетками (а потом и оплодотворением, то есть слиянием гамет). И вот уже у истоков новой жизни (то есть в зиготе) эпигенетические признаки получат возможность немного постоять у руля вновь запускающейся программы развития. Тут стоит вспомнить, как у мышей метилирование ДНК в гаметах влияет на репродуктивную функцию [61], а у пчел определенные гены оказываются во власти транскрипционных факторов, и их сети начинают неконтролируемо усложняться [62].
Давайте разберемся, какие эпигенетические метки и как именно передаются от родителей детям.
Геномный импринтинг
В самом общем случае этот термин обозначает ситуацию, когда гены в организме читаются неодинаково с материнских и отцовских хромосом. Еще в 1980-х годах было показано на млекопитающих, что если искусственно сделать зиготу путем слияния двух материнских или двух отцовских ядер, то итоговый организм, увы, будет нежизнеспособен [63]. Следовательно, есть что-то уникальное как на материнских, так и на отцовских хромосомах, что нужно для развития организма [64], и это не просто генетическая последовательность.
За некоторые случаи импринтинга отвечает метилирование ДНК, протекающее по специальным регуляторным участкам контроля импринтинга (ICR). Обычно они контролируют несколько генов и по крайней мере одну регуляторную длинную некодирующую РНК. ICR могут работать по-разному:
- Метилирование ICR может выключить чтение некодирующей РНК. Она, в свою очередь, «глушит» чтение других генов, а потому, если ее саму «выключить», эти гены читаются лучше. Вероятнее всего, это происходит так: ее последовательность в ДНК перекрывается с промотором подконтрольного гена и постоянно читается. И белки, которые ее читают, не дают связаться РНК-полимеразе с промотором замалчиваемого гена. Получается, что ее транскрипция мешает транскрипции этого гена [65].
- ICR может быть инсулятором, то есть влиять на сближение промотора и энхансера в пространстве (а это улучшает чтение гена). Если такой ICR метилирован, с ним не может связаться CTCF; не образуется характерная хроматиновая петля, «перехваченная» CTCF; и энхансер не может воздействовать на промотор. Разное метилирование участков контроля импринтинга на материнских и отцовских хромосомах может, в частности, обусловливать разный доступ участков к CTCF, что ведет к неодинаковому чтению генов [65].
Итак, определенные участки на отцовских и материнских хромосомах метилированы по-разному у мужчин и женщин, и от этого зависит регуляция чтения генов, контролируемых этими участками. Интересно, что в гаметах паттерн метилирования отличается и от материнского, и от отцовского. Как же так получается? Ведь по логике, поскольку у всех клеток организма двойной набор хромосом, а у гамет одинарный, гаметы при формировании (которое включает в себя деление клеток с двойным набором хромосом) должны унаследовать на части своих хромосом материнское метилирование, а на части — отцовское. Так вот, дело в том, что во время развития клеток, которые дают начало гаметам, вообще всё метилирование стирается, а потом устанавливается заново. Интересно, что и в зиготе после оплодотворения метилирование всей ДНК (кроме как раз участков контроля импринтинга), тоже стирается, а потом все метилируется заново [64], [66].
Как возникает импринтинг? Есть две гипотезы. Прежде чем их объяснить, стоит упомянуть, что импринтинг метилирования широко распространен только у млекопитающих, хотя и был открыт на насекомых. Первая гипотеза подразумевает, что отцовский импринтинг стимулирует, а материнский замалчивает чтение генов, важных для роста. Такие гены и правда есть: например, это ген инсулиноподобного фактора роста Igf2, экспрессия которого увеличивает размер плода [67]. По этой гипотезе, отцовский геном стремится как можно больше распространить свои гены, а материнский геном — распространить свои гены вместе с как можно большим числом партнеров и, соответственно, равномерно распределить свои ресурсы на все потомство [65], [68]. Согласно второй гипотезе, импринтинг — своеобразная защита от развития организма из неоплодотворенной яйцеклетки в материнском организме, который, вообще говоря, «оборудован» для этого всем необходимым [65], [69].
Импринтинг важен, поскольку контролирует гены развития организма. И как мы увидим в шестой статье «Медицина и эпигеном», делеции участков контроля импринтинга ведут к наследственным заболеваниям, например, синдрому Прадера—Вилли. Потеря импринтинга может вести и к заболеваниям во взрослом организме: например, она характерна для некоторых видов рака [70].
От эпигенетики к фенотипу: как передать его через гаметы?
Давайте рассмотрим несколько работ по наследованию эпигенетических регуляторов. Например, у рыбки Danio rerio есть длинная некодирующая РНК Cyrano, расположенная в половых клетках и напрямую передающаяся потомству. Если ее убрать, то у развивающегося организма будут проблемы с формированием мозга [71]. Интересно, что эта РНК может служить «губкой» для РНК-связывающих белков, оттягивая их на себя и не позволяя связываться с другими молекулами РНК и хроматином [72]. Возможно, именно в этом заключается ее роль в развивающемся мозге, хотя это еще только предстоит исследовать.
Отдельно стоит упомянуть про фрагменты транспортных РНК, уже не поставляющие аминокислоты к рибосомам во время трансляции, но вместо этого примеряющие на себя роль отдельного класса некодирующих РНК. Например, фрагмент глициновой тРНК подавляет транскрипцию генов, содержащих транспозоны! Причем действует он не напрямую, а через другие РНК, формирующие гетерохроматин в районе «зараженных» генов и «выключающие» их. Интересно, что фрагменты этой тРНК находятся в сперме мышей, причем их концентрация повышается, если посадить мышей на малобелковую диету. Причем тут диета — неизвестно, но вот сами фрагменты тРНК защищают эмбрион от транспозонов [73], [74].
Известен пример, когда фенотип передается через гистоновые модификации. У червячка C. elegans одна из главных гетерохроматиновых меток гистонов H3K9me2 связана с продолжительностью жизни. Есть комплекс COMPASS, который навешивает на промоторы метки H3K4me3, и если его отключить, червячок будет жить дольше. Но эффект можно обратить, если дополнительно выключить H3K9me2. Выходит, что метки противостоят друг другу, и увеличенная продолжительность жизни у C. elegans наследуется благодаря одной-единственной модификации гистона [75]!
Давно известно, что сайленсинг генов у червячка C. elegans при помощи дцРНК иногда передается по наследству: дело в том, что у этого организма (ну и у дрожжей с растениями) есть РНК-зависимая РНК-полимераза, «плодящая» малые РНК. Однако производство этих малых РНК возможно, например, и в ответ на заражение вирусом. Вирус может нарезаться на такие РНК, а потом они плодятся и выключают его РНК-интерференцией. Такие малые РНК, синтез которых запускается вирусами, тоже могут передаваться по наследству, вакцинируя последующие поколения [76].
Но передача по наследству «антивирусных» свойств — это еще цветочки. В нейронах того же C. elegans вырабатываются другие малые РНК, управляющие поиском еды при определенных температурах. Благодаря им червячки начинают искать еду или лучше, или хуже. И эта способность также передается по наследству, а если ее разрушить, то потомки уже не смогут искать пищу и будут голодать :-( [77].
Причем, как оказалось, число поколений, которым передаются малые РНК, можно регулировать, включая или выключая гены белков, манипулирующих этими РНК [78]. Наконец, в 2020 году вышла статья, описывающая основные правила наследования малых РНК. Они распределяются равномерно по потомству, а их «сила» устанавливается в каждом червячке-маме [79].
Изменения в хроматине половых клеток — все меняем, но помним!
Итак, мы разобрались, как наследуются малые и длинные некодирующие РНК (это происходит через гаметы); а также метилирование ДНК (оно попросту стирается, в зиготе за исключением участков контроля импринтинга). И то, и другое существенно отличается от репликации ДНК и деления одной клетки — особенно, если это клетка соматическая. Большинство подробностей эпигенетического наследования в контексте целого организма еще не изучено, но точно известно, что эпигеном половых клеток отличается от эпигенома соматических. Несмотря на такое отличие, предшествующая гаметам клеточная структура частично запоминается, чтобы воплотиться в новом организме.
Нужно отметить, что изменения в метилировании ДНК — это только начало. Структура хроматина в половых и соматических клетках местами заметно различается: например, в сперматозоидах вообще нет гистонов. Там ДНК намотана на белки-протамины, что приводит к гораздо более плотной упаковке. Однако даже у организмов, где в сперматозоидах гистоны таки есть, ДНК наматывается на очень специфические их варианты. Как и в сперме, в ооцитах млекопитающих есть свои особенности: специфические варианты гистонов и распределение гистоновой метки H3K4me3 по кодирующим генам, а не по промоторам, как в соматических клетках [80].
Трехмерная организация генома, и без того очень изменчивая от клетки к клетке, тоже перестраивается при оплодотворении. Вот пример: в женских пронуклеусах — гаплоидных женских ядрах в составе оплодотворенной яйцеклетки — нет компартментов A и B (характерных паттернов контактов между ДНК, которые есть во всех изученных клетках взрослого организма [81]).
Интересно, что даже в таком «перепрограммированном» состоянии гаметы способны получать стимулы и конвертировать их в эпигенетические изменения, которые влияют на будущий организм. Например, у крыс может наследоваться отвращение к запаху. Получается, нейроны взрослой крысы запоминают что-то и сигналят об этом половым клеткам, которые запоминают эпигенетический стимул и могут воспроизвести всю его сложность во взрослом организме [82].
Наследование трехмерной архитектуры хроматина
Подраздел написала Александра Галицына.
Наследование трехмерной архитектуры хроматина лучше всего изучено у мышей. После оплодотворения материнский и отцовский геномы некоторое время не смешиваются и существуют в разных областях ядра. Удивительно, что сразу после оплодотворения можно обнаружить домены, в которые сворачивается хроматин материнских и отцовских хромосом, причем эти домены разные в отцовском и материнском геномах (такое явление называется аллель-специфичностью). Такие «родительские» домены образуют взаимодействия по типу компартментов, и, возможно, являются наследуемыми метками Polycomb. Однако с последующими делениями эмбриона эта информация теряется, и на месте «родительских» доменов возникают новые, на этот раз одинаковые в материнских и отцовских хромосомах [83].
Интересно, что на первых этапах деления эмбриона сохраняется не только доменная структура, но и взаимное расположение хромосом. Так, когда эмбрион состоит всего из двух клеток, каждая из них «помнит» структуру хроматина одноклеточной зиготы. Возможно, это происходит благодаря тому, что хромосомы не успевают перемешиваться в ходе митоза, ведь деление ранних эмбрионов происходит очень быстро [84].
Как долго сохраняется память о трехмерной укладке хромосом после оплодотворения, точно не известно. Часть этой информации может передаваться посредством эпигенетических меток. В этом случае структура хроматина формируется по правилам, которые «записаны» в эпигенетических метках, переданных от родителей.
Можно ли воссоздать организм по коду ДНК?
В заключение давайте обсудим некоторые вопросы о том, как эпигенетические молекулы преобразуют генотип в фенотип. А также опровергнем популярное заблуждение о том, что можно клонировать мамонта.
Конечно, вы скажете, чтение генов, закодированных в ДНК, регулируется метилированием ДНК, а также белками и РНК хроматина. Но ведь все белки и РНК, регулирующие гены, закодированы в ДНК. Следовательно, по линейной ДНК можно воссоздать организм?
Увы, нет, все не так просто. Несмотря на то, что метилирование ДНК сохраняется на молекуле, во-первых, чтобы запустить транскрипцию этих генов точно как у мамонта, нужны точно такие же молекулы хроматина на тех же местах генома. Во-вторых, этот хроматин в ядре нужно уложить в пространстве в точности так, как это было у мамонта. Не знаю, удастся ли когда-нибудь этого добиться, но пока что это, кажется, за гранью фантастики.
Хорошо, спросите вы, а что будет, если взять две абсолютно одинаковые клетки и поместить их в абсолютно одинаковые условия, будет ли их эпигеном одинаков через какое-то время? Похоже, что нет, и если эти клетки дадут начало организму, даже геном будет разный, поскольку, по крайней мере, некоторые процессы на хроматине стохастичны, то есть осуществляются с разной интенсивностью от случая к случаю [85], [86]. Хорошим примером является генетическая мозаичность, появляющаяся в результате репликационных мутаций, характерная в том числе и для нашего с вами мозга [87].
После разбора эпигенетических механизмов наследования становится понятно, что проект клонирования мамонта, где ученые планируют взять ядро мамонта и ввести его в яйцеклетку слона с удаленным ядром, строго говоря, не приведет к клонированию мамонта. Потому что некоторые из белков и РНК слона, находящиеся в цитоплазме, будут влиять на хроматин в ядре. А еще есть маленькие молекулы, задействованные в метаболизме (о них мы поговорим в одной из следующих статей).
Тут, пожалуй, можно привести еще один пример: если удалить из зиготы ядро и перенести в нее ядро неполовой (соматической) клетки, хроматиновые домены в таком ядре изменят время репликации по сравнению с исходной клеткой. То есть что-то переместится из цитоплазмы зиготы в ядро соматической клетки и повлияет на эти хроматиновые домены [46].
Еще ученые планируют взять клетки слона и постепенно заменить там хотя бы часть генов генами мамонта, последовательность которых на сегодняшний день уже известна [89]. Представим себе, что мы взяли клетки слона и заменили ген, кодирующий белок, который модифицирует гистоны, на ген мамонта. Этот белок будет действовать на хроматине не так, как он действовал в мамонте, а в контексте хроматиновых белков и РНК слона. И так далее. Поэтому если даже заменить все гены слона генами мамонта, то, что получится в итоге, — все равно гибрид слона и мамонта.
Может быть, мамонта клонировать и не получится, но несомненно на нашем знании наследования в будущем будут основаны некоторые достижения эпигенетики. Например, очень вероятно, что мы научимся перепрограммировать половые клетки и вызывать эффекты во взрослом организме — что-то вроде отвращения к запаху у крыс, но намного более полезное. Однако это нужно будет делать с серьезной биоэтической оглядкой.
Заключение
Мы разобрали, какие молекулы обусловливают эпигенетическое наследование и как конкретно оно осуществляется. Мы познакомились с разными механизмами, от работы шаперонов во время репликации, которые перекидывают гистоны и другие молекулы с родительских на дочерние цепи ДНК, до гистоновых модификаций и специальных белков, которые «помнят», какие гены включить после митоза.
Мы узнали, что эпигеном половых клеток отличается от эпигенома соматических. Однако надо понимать, что в процессе развития организма часть клеток «перепрограммируется» обратно в соматические, и все клетки формируют фенотип, за определенные части которого нередко отвечают отдельные молекулы.
Знание эпигенетики помогает разбираться с проблемами наследования, а это, в свою очередь, много рассказывает об определенных заболеваниях (например, связанных с нарушением импринтинга). Но подробнее об этом — в шестой статье проекта «Медицина и эпигеном».
Литература
- Геномная головоломка: открой в себе мозаика;
- Путешествие во времени: как судьба первых клеток эмбриона влияет на его дальнейшее развитие и риск заболеваний;
- Молекулы и эпигеном ;
- Ядро и эпигеном;
- Загадочное путешествие некодирующей РНК Xist по X-хромосоме;
- Jun Jiang, Yuanchun Jing, Gregory J. Cost, Jen-Chieh Chiang, Heather J. Kolpa, et. al.. (2013). Translating dosage compensation to trisomy 21. Nature. 500, 296-300;
- D. B. Dunn, J. D. Smith. (1958). The occurrence of 6-methylaminopurine in deoxyribonucleic acids. Biochemical Journal. 68, 627-636;
- Шестое ДНК-основание: от открытия до признания;
- Qifan Zhu, Reinhard Stöger, Ramiro Alberio. (2018). A Lexicon of DNA Modifications: Their Roles in Embryo Development and the Germline. Front. Cell Dev. Biol.. 6;
- A. Zemach, I. E. McDaniel, P. Silva, D. Zilberman. (2010). Genome-Wide Evolutionary Analysis of Eukaryotic DNA Methylation. Science. 328, 916-919;
- Danuta M. Jeziorska, Robert J. S. Murray, Marco De Gobbi, Ricarda Gaentzsch, David Garrick, et. al.. (2017). DNA methylation of intragenic CpG islands depends on their transcriptional activity during differentiation and disease. Proc Natl Acad Sci USA. 114, E7526-E7535;
- François Fuks, Paul J. Hurd, Daniel Wolf, Xinsheng Nan, Adrian P. Bird, Tony Kouzarides. (2003). The Methyl-CpG-binding Protein MeCP2 Links DNA Methylation to Histone Methylation. Journal of Biological Chemistry. 278, 4035-4040;
- Bernhard Lehnertz, Yoshihide Ueda, Alwin A.H.A. Derijck, Ulrich Braunschweig, Laura Perez-Burgos, et. al.. (2003). Suv39h-Mediated Histone H3 Lysine 9 Methylation Directs DNA Methylation to Major Satellite Repeats at Pericentric Heterochromatin. Current Biology. 13, 1192-1200;
- Nathan R. Rose, Robert J. Klose. (2014). Understanding the relationship between DNA methylation and histone lysine methylation. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1839, 1362-1372;
- Albert Jeltsch. (2006). On the Enzymatic Properties of Dnmt1: Specificity, Processivity, Mechanism of Linear Diffusion and Allosteric Regulation of the Enzyme. Epigenetics. 1, 63-66;
- Eric Hervouet, Paul Peixoto, Régis Delage-Mourroux, Michaël Boyer-Guittaut, Pierre-François Cartron. (2018). Specific or not specific recruitment of DNMTs for DNA methylation, an epigenetic dilemma. Clin Epigenet. 10;
- Huiming Zhang, Zhaobo Lang, Jian-Kang Zhu. (2018). Dynamics and function of DNA methylation in plants. Nat Rev Mol Cell Biol. 19, 489-506;
- Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала;
- Sandra Catania, Phillip A. Dumesic, Harold Pimentel, Ammar Nasif, Caitlin I. Stoddard, et. al.. (2020). Evolutionary Persistence of DNA Methylation for Millions of Years after Ancient Loss of a De Novo Methyltransferase. Cell. 180, 263-277.e20;
- Karolin Luger, Armin W. Mäder, Robin K. Richmond, David F. Sargent, Timothy J. Richmond. (1997). Crystal structure of the nucleosome core particle at 2.8 Å resolution. Nature. 389, 251-260;
- Geneviève Almouzni, Howard Cedar. (2016). Maintenance of Epigenetic Information. Cold Spring Harb Perspect Biol. 8, a019372;
- Ilnaz M. Klimovskaia, Clifford Young, Caroline B. Strømme, Patrice Menard, Zuzana Jasencakova, et. al.. (2014). Tousled-like kinases phosphorylate Asf1 to promote histone supply during DNA replication. Nat Commun. 5;
- Hongda Huang, Caroline B Strømme, Giulia Saredi, Martina Hödl, Anne Strandsby, et. al.. (2015). A unique binding mode enables MCM2 to chaperone histones H3–H4 at replication forks. Nat Struct Mol Biol. 22, 618-626;
- Peter J. Skene, Steven Henikoff. (2013). Histone variants in pluripotency and disease. Development. 140, 2513-2524;
- Benedetto Daniele Giaimo, Francesca Ferrante, Andreas Herchenröther, Sandra B. Hake, Tilman Borggrefe. (2019). The histone variant H2A.Z in gene regulation. Epigenetics & Chromatin. 12;
- Marta Radman-Livaja, Kitty F. Verzijlbergen, Assaf Weiner, Tibor van Welsem, Nir Friedman, et. al.. (2011). Patterns and Mechanisms of Ancestral Histone Protein Inheritance in Budding Yeast. PLoS Biol. 9, e1001075;
- Nazaret Reverón-Gómez, Cristina González-Aguilera, Kathleen R. Stewart-Morgan, Nataliya Petryk, Valentin Flury, et. al.. (2018). Accurate Recycling of Parental Histones Reproduces the Histone Modification Landscape during DNA Replication. Molecular Cell. 72, 239-249.e5;
- Constance Alabert, Carolin Loos, Moritz Voelker-Albert, Simona Graziano, Ignasi Forné, et. al.. (2020). Domain Model Explains Propagation Dynamics and Stability of Histone H3K27 and H3K36 Methylation Landscapes. Cell Reports. 30, 1223-1234.e8;
- Sahana Holla, Jothy Dhakshnamoorthy, H. Diego Folco, Vanivilasini Balachandran, Hua Xiao, et. al.. (2020). Positioning Heterochromatin at the Nuclear Periphery Suppresses Histone Turnover to Promote Epigenetic Inheritance. Cell. 180, 150-164.e15;
- Thelma M. Escobar, Ozgur Oksuz, Ricardo Saldaña-Meyer, Nicolas Descostes, Roberto Bonasio, Danny Reinberg. (2019). Active and Repressed Chromatin Domains Exhibit Distinct Nucleosome Segregation during DNA Replication. Cell. 179, 953-963.e11;
- Jean-Pierre Quivy, Danièle Roche, Doris Kirschner, Hideaki Tagami, Yoshihiro Nakatani, Geneviève Almouzni. (2004). A CAF-1 dependent pool of HP1 during heterochromatin duplication. EMBO J. 23, 3516-3526;
- Модельные организмы: грибы;
- Robin C. Allshire, Hiten D. Madhani. (2018). Ten principles of heterochromatin formation and function. Nat Rev Mol Cell Biol. 19, 229-244;
- Alessia Buscaino, Erwan Lejeune, Pauline Audergon, Georgina Hamilton, Alison Pidoux, Robin C Allshire. (2013). Distinct roles for Sir2 and RNAi in centromeric heterochromatin nucleation, spreading and maintenance. EMBO J. 32, 1250-1264;
- Karl Ekwall, Tim Olsson, Bryan M. Turner, Gwen Cranston, Robin C. Allshire. (1997). Transient Inhibition of Histone Deacetylation Alters the Structural and Functional Imprint at Fission Yeast Centromeres. Cell. 91, 1021-1032;
- Giacomo Cavalli, Renato Paro. (1998). The Drosophila Fab-7 Chromosomal Element Conveys Epigenetic Inheritance during Mitosis and Meiosis. Cell. 93, 505-518;
- Giacomo Cavalli, Renato Paro. (1999). Epigenetic Inheritance of Active Chromatin After Removal of the Main Transactivator. Science. 286, 955-958;
- Zachary H. Harvey, Anupam K. Chakravarty, Raymond A. Futia, Daniel F. Jarosz. (2020). A Prion Epigenetic Switch Establishes an Active Chromatin State. Cell. 180, 928-940.e14;
- Прионные и неприонные амилоиды: определяет ли конформация разницу в инфекционности?;
- Srinivas Ramachandran, Steven Henikoff. (2016). Transcriptional Regulators Compete with Nucleosomes Post-replication. Cell. 165, 580-592;
- Ishara F Azmi, Shinya Watanabe, Michael F Maloney, Sukhyun Kang, Jason A Belsky, et. al.. (2017). Nucleosomes influence multiple steps during replication initiation. eLife. 6;
- M. L. Eaton, K. Galani, S. Kang, S. P. Bell, D. M. MacAlpine. (2010). Conserved nucleosome positioning defines replication origins. Genes & Development. 24, 748-753;
- Paul Batty, Daniel W. Gerlich. (2019). Mitotic Chromosome Mechanics: How Cells Segregate Their Genome. Trends in Cell Biology. 29, 717-726;
- Alisa Zhiteneva, Juan Jose Bonfiglio, Alexandr Makarov, Thomas Colby, Paola Vagnarelli, et. al.. (2017). Mitotic post-translational modifications of histones promote chromatin compaction in vitro. Open Biol.. 7, 170076;
- Georg O.M. Bobkov, Nick Gilbert, Patrick Heun. (2018). Centromere transcription allows CENP-A to transit from chromatin association to stable incorporation. Journal of Cell Biology. 217, 1957-1972;
- Yoel Shufaro, Orly Lacham-Kaplan, Ben-Zion Tzuberi, John McLaughlin, Alan Trounson, et. al.. (2010). Reprogramming of DNA Replication Timing. Stem Cells. N/A-N/A;
- Bobbie Pelham-Webb, Alexander Polyzos, Luke Wojenski, Andreas Kloetgen, Jiexi Li, et. al.. (2021). H3K27ac bookmarking promotes rapid post-mitotic activation of the pluripotent stem cell program without impacting 3D chromatin reorganization. Molecular Cell. 81, 1732-1748.e8;
- Vishwanath R. Iyer. (2012). Nucleosome positioning: bringing order to the eukaryotic genome. Trends in Cell Biology. 22, 250-256;
- Nicolas Nocetti, Iestyn Whitehouse. (2016). Nucleosome repositioning underlies dynamic gene expression. Genes Dev.. 30, 660-672;
- Carlos Perea-Resa, Leah Bury, Iain M. Cheeseman, Michael D. Blower. (2020). Cohesin Removal Reprograms Gene Expression upon Mitotic Entry. Molecular Cell. 78, 127-140.e7;
- Nick Owens, Thaleia Papadopoulou, Nicola Festuccia, Alexandra Tachtsidi, Inma Gonzalez, et. al.. (2019). CTCF confers local nucleosome resiliency after DNA replication and during mitosis. eLife. 8;
- Обо всех РНК на свете, больших и малых;
- Модельные организмы: нематода;
- Jonathan I Gent, Mara Schvarzstein, Anne M Villeneuve, Sam Guoping Gu, Verena Jantsch, et. al.. (2009). A Caenorhabditis elegans RNA-Directed RNA Polymerase in Sperm Development and Endogenous RNA Interference. Genetics. 183, 1297-1314;
- Savio T. de Farias, Ariosvaldo P. dos Santos Junior, Thais G. Rêgo, Marco V. José. (2017). Origin and Evolution of RNA-Dependent RNA Polymerase. Front. Genet.. 8;
- Natasha E. Weiser, John K. Kim. (2019). Multigenerational Regulation of the Caenorhabditis elegans Chromatin Landscape by Germline Small RNAs. Annu. Rev. Genet.. 53, 289-311;
- Stephane E. Castel, Robert A. Martienssen. (2013). RNA interference in the nucleus: roles for small RNAs in transcription, epigenetics and beyond. Nat Rev Genet. 14, 100-112;
- Waqar Islam, Ali Noman, Muhammad Qasim, Liande Wang. (2018). Plant Responses to Pathogen Attack: Small RNAs in Focus. IJMS. 19, 515;
- piРНК — аналог CRISPR у эукариот;
- Emily Niemitz. (2012). Transgenerational epigenetic inheritance and piRNAs. Nat Genet. 44, 840-840;
- Эпигенетика: невидимый командир генома;
- На пути к развитому социуму пчелы преодолевали разные генетические тернии;
- James McGrath, Davor Solter. (1984). Completion of mouse embryogenesis requires both the maternal and paternal genomes. Cell. 37, 179-183;
- Valter Tucci, Anthony R. Isles, Gavin Kelsey, Anne C. Ferguson-Smith, Valter Tucci, et. al.. (2019). Genomic Imprinting and Physiological Processes in Mammals. Cell. 176, 952-965;
- D. P. Barlow, M. S. Bartolomei. (2014). Genomic Imprinting in Mammals. Cold Spring Harbor Perspectives in Biology. 6, a018382-a018382;
- Yang Zeng, Taiping Chen. (2019). DNA Methylation Reprogramming during Mammalian Development. Genes. 10, 257;
- Veronica White, Alicia Jawerbaum, Maria Belen Mazzucco, Martin Gauster, Gernot Desoye, Ursula Hiden. (2018). IGF2 stimulates fetal growth in a sex- and organ-dependent manner. Pediatr Res. 83, 183-189;
- T Moore. (1991). Genomic imprinting in mammalian development: a parental tug-of-war. Trends in Genetics. 7, 45-49;
- Sue Varmuza, Mellissa Mann. (1994). Genomic imprinting — defusing the ovarian time bomb. Trends in Genetics. 10, 118-123;
- Teresa M. Holm, Laurie Jackson-Grusby, Tobias Brambrink, Yasuhiro Yamada, William M. Rideout, Rudolf Jaenisch. (2005). Global loss of imprinting leads to widespread tumorigenesis in adult mice. Cancer Cell. 8, 275-285;
- Mayuresh Anant Sarangdhar, Divya Chaubey, Nagesh Srikakulam, Beena Pillai. (2018). Parentally inherited long non-coding RNA Cyrano is involved in zebrafish neurodevelopment. Nucleic Acids Research. 46, 9726-9735;
- Jiyoung Kim, Kotb Abdelmohsen, Xiaoling Yang, Supriyo De, Ioannis Grammatikakis, et. al.. (2016). LncRNAOIP5-AS1/cyranosponges RNA-binding protein HuR. Nucleic Acids Res. 44, 2378-2392;
- Ana Boskovic, Xin Yang Bing, Ebru Kaymak, Oliver J. Rando. (2020). Control of noncoding RNA production and histone levels by a 5′ tRNA fragment. Genes Dev.. 34, 118-131;
- Upasna Sharma, Colin C. Conine, Jeremy M. Shea, Ana Boskovic, Alan G. Derr, et. al.. (2016). Biogenesis and function of tRNA fragments during sperm maturation and fertilization in mammals. Science. 351, 391-396;
- Teresa Wei-sy Lee, Heidi Shira David, Amanda Kathryn Engstrom, Brandon Scott Carpenter, David John Katz. (2019). Repressive H3K9me2 protects lifespan against the transgenerational burden of COMPASS activity in C. elegans. eLife. 8;
- Oded Rechavi, Gregory Minevich, Oliver Hobert. (2011). Transgenerational Inheritance of an Acquired Small RNA-Based Antiviral Response in C. elegans. Cell. 147, 1248-1256;
- Rachel Posner, Itai Antoine Toker, Olga Antonova, Ekaterina Star, Sarit Anava, et. al.. (2019). Neuronal Small RNAs Control Behavior Transgenerationally. Cell. 177, 1814-1826.e15;
- Leah Houri-Ze’evi, Yael Korem, Hila Sheftel, Lior Faigenbloom, Itai Antoine Toker, et. al.. (2016). A Tunable Mechanism Determines the Duration of the Transgenerational Small RNA Inheritance in C. elegans. Cell. 165, 88-99;
- Leah Houri-Zeevi, Yael Korem Kohanim, Olga Antonova, Oded Rechavi. (2020). Three Rules Explain Transgenerational Small RNA Inheritance in C. elegans. Cell. 182, 1186-1197.e12;
- Ana Bošković, Oliver J. Rando. (2018). Transgenerational Epigenetic Inheritance. Annu. Rev. Genet.. 52, 21-41;
- Ilya M. Flyamer, Johanna Gassler, Maxim Imakaev, Hugo B. Brandão, Sergey V. Ulianov, et. al.. (2017). Single-nucleus Hi-C reveals unique chromatin reorganization at oocyte-to-zygote transition. Nature. 544, 110-114;
- Brian G Dias, Kerry J Ressler. (2014). Parental olfactory experience influences behavior and neural structure in subsequent generations. Nat Neurosci. 17, 89-96;
- Samuel Collombet, Noémie Ranisavljevic, Takashi Nagano, Csilla Varnai, Tarak Shisode, et. al.. (2020). Parental-to-embryo switch of chromosome organization in early embryogenesis. Nature. 580, 142-146;
- Andrew C. Payne, Zachary D. Chiang, Paul L. Reginato, Sarah M. Mangiameli, Evan M. Murray, et. al.. (2021). In situ genome sequencing resolves DNA sequence and structure in intact biological samples. Science. 371;
- Christopher R. Brown, Changhui Mao, Elena Falkovskaia, Melissa S. Jurica, Hinrich Boeger. (2013). Linking Stochastic Fluctuations in Chromatin Structure and Gene Expression. PLoS Biol. 11, e1001621;
- Chrysa Nikopoulou, Giorgos Panagopoulos, Georgios Sianidis, Eleni Psarra, Ethan Ford, Dimitris Thanos. (2018). The Transcription Factor ThPOK Orchestrates Stochastic Interchromosomal Interactions Required for IFNB1 Virus-Inducible Gene Expression. Molecular Cell. 71, 352-361.e5;
- Геномная головоломка: открой в себе мозаика;
- Eleftheria Palkopoulou, Swapan Mallick, Pontus Skoglund, Jacob Enk, Nadin Rohland, et. al.. (2015). Complete Genomes Reveal Signatures of Demographic and Genetic Declines in the Woolly Mammoth. Current Biology. 25, 1395-1400;
- Alessia Buscaino, Erwan Lejeune, Pauline Audergon, Georgina Hamilton, Alison Pidoux, Robin C Allshire. (2013). Distinct roles for Sir2 and RNAi in centromeric heterochromatin nucleation, spreading and maintenance. EMBO J. 32, 1250-1264;
- M. F. Dion, T. Kaplan, M. Kim, S. Buratowski, N. Friedman, O. J. Rando. (2007). Dynamics of Replication-Independent Histone Turnover in Budding Yeast. Science. 315, 1405-1408;
- J. M. Engreitz, A. Pandya-Jones, P. McDonel, A. Shishkin, K. Sirokman, et. al.. (2013). The Xist lncRNA Exploits Three-Dimensional Genome Architecture to Spread Across the X Chromosome. Science. 341, 1237973-1237973;
- Nahid Iglesias, Joao A. Paulo, Antonis Tatarakis, Xiaoyi Wang, Amanda L. Edwards, et. al.. (2020). Native Chromatin Proteomics Reveals a Role for Specific Nucleoporins in Heterochromatin Organization and Maintenance. Molecular Cell. 77, 51-66.e8;
- Jun Jiang, Yuanchun Jing, Gregory J. Cost, Jen-Chieh Chiang, Heather J. Kolpa, et. al.. (2013). Translating dosage compensation to trisomy 21. Nature. 500, 296-300;
- Daniel H Kim, Louisa M Villeneuve, Kevin V Morris, John J Rossi. (2006). Argonaute-1 directs siRNA-mediated transcriptional gene silencing in human cells. Nat Struct Mol Biol. 13, 793-797;
- D. B. Marina, S. Shankar, P. Natarajan, K. J. Finn, H. D. Madhani. (2013). A conserved ncRNA-binding protein recruits silencing factors to heterochromatin through an RNAi-independent mechanism. Genes & Development. 27, 1851-1856;
- Atsuya Nishiyama, Luna Yamaguchi, Jafar Sharif, Yoshikazu Johmura, Takeshi Kawamura, et. al.. (2013). Uhrf1-dependent H3K23 ubiquitylation couples maintenance DNA methylation and replication. Nature. 502, 249-253;
- Paul Victor Sauer, Jennifer Timm, Danni Liu, David Sitbon, Elisabetta Boeri-Erba, et. al.. (2017). Insights into the molecular architecture and histone H3-H4 deposition mechanism of yeast Chromatin assembly factor 1. eLife. 6;
- Kaushikaram Subramanian, Martin Weigert, Oliver Borsch, Heike Petzold, Alfonso Garcia-Ulloa, et. al.. (2019). Rod nuclear architecture determines contrast transmission of the retina and behavioral sensitivity in mice. eLife. 8;
- Hideaki Tagami, Dominique Ray-Gallet, Geneviève Almouzni, Yoshihiro Nakatani. (2004). Histone H3.1 and H3.3 Complexes Mediate Nucleosome Assembly Pathways Dependent or Independent of DNA Synthesis. Cell. 116, 51-61;
- Michael A. Wheeler, Iain C. Clark, Emily C. Tjon, Zhaorong Li, Stephanie E. J. Zandee, et. al.. (2020). MAFG-driven astrocytes promote CNS inflammation. Nature. 578, 593-599;
- Ruby Yu, Xiaoyi Wang, Danesh Moazed. (2018). Epigenetic inheritance mediated by coupling of RNAi and histone H3K9 methylation. Nature. 558, 615-619;
- Iva B. Zovkic, Brynna S. Paulukaitis, Jeremy J. Day, Deepa M. Etikala, J. David Sweatt. (2014). Histone H2A.Z subunit exchange controls consolidation of recent and remote memory. Nature. 515, 582-586.